转录因子研究方法!
近年来,随着各种分子生物学技术的迅猛发展,越来越多的老师开始着眼于转录因子的研究,各类基金中涉及转录因子研究的比例也是逐年增加。因此,我们决定分享一些转录因子的研究方法供各位老师参考,有不到之处或不同观点可以随时沟通讨论。
1.什么是转录因子
转录因子(Transcriptionfactor,TF)的调控决定着基因的调控网络以及表达水平。基因的表达驱动着一系列的细胞活动,这些表达的调控需要通过转录因子(蛋白)和转录因子结合位点(DNA元件)的相互作用实现。转录水平的调控不是一个简单的独立过程,它是由上百个转录因子,目标序列以及共调控因子所组成的高度互作的基因调控网络。
2.为什么研究转录因子
一个课题最开始往往是研究某个基因的序列信息以及基因所行驶的功能作用,这些是属于基因的下游研究。但在下游研究累积到一定经验之后,老师往往开始着手于研究基因的上游调控机理,即不单关注基因A如何影响细胞活动,更关注哪些因素影响基因A的功能发挥。调控机理研究理论上会涉及多个层面的因素,例如:转录因子、蛋白激酶、miRNA、DNA甲基化、组蛋白修饰、lncRNA……
转录因子成为调控机理的研究大户是最正常不过的事情。因为一个转录因子能同时控制多个基因的表达,同时转录因子的功能作用也可能受多个共作用因子的影响。如果能阐明如此复杂的调控网络,这可是很有科研成就感的。
3.研究方法
现有主流的转录因子鉴别方法可分为两种:传统实验鉴别(experimental methods)以及通过计算机进行的生物信息学鉴别(computational methods)。简单来说,实验方法主要用于寻找不同类型目标,即通过已知TF寻找未知TFBS,或通过已知TFBS找出其对应TF。而生物信息学方法更多是用于同类型的预测,即通过序列保守性分析,通过已知TF预测未知TF,或通过已知TFBS预测未知TFBS。这些研究思路的关系如图1所示。
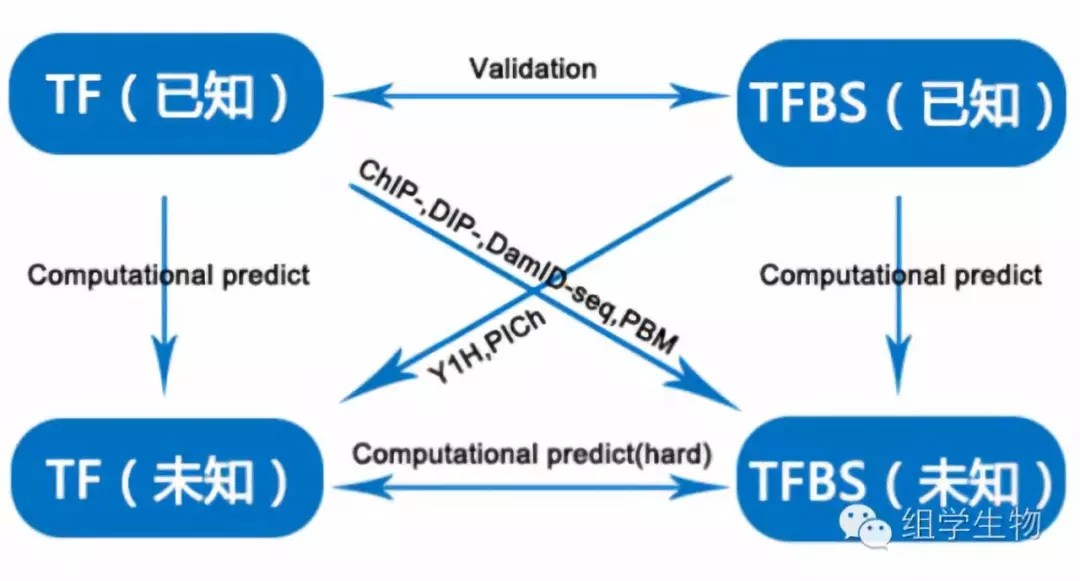
3.1生物信息学预测
无论是TF还是TFBS的同类预测,生物信息的研究手段主要依赖于蛋白质或者DNA的序列保守性进行。具体算法和软件有隐马尔可夫模型,Gibbs抽样,贪婪算法等,但这次重点在于介绍研究转录因子的实验方法,因此不过于叙述这部分信息。
3.2实验方法筛选
实验方法主要有两种思路:1.通过已知TF寻找未知TFBS;2.通过已知TFBS寻找未知TF。无论哪种手段,前提都是基于蛋白与DNA之间的结合关系寻找,即交叉寻找目标,这有别于计算机技术的同类型寻找。
3.2.1通过已知TF寻找未知TFBS
如果已经锁定了一个感兴趣的TF,那常用思路是确定其TFBS,然后得知道它控制的下游基因。从已知TF定位到未知TFBS的过程,主要的实验方法包括ChIP-seq、DNaseI-seq、DamID-seq等体内实验以及DIP-seq、SELEX(-seq)、PBM等体外实验。
这些研究的通量与应用范围如图2所示,其中,纵坐标表示可以从多大范围检测TFBS(检测通量),横坐标表示TF-TFBS的结合细节(检测精细程度)。以ChIP-seq为例,它可以在全基因组水平一次发现超过20万个结合位点信息,但只能确定到TFBS的序列信息(是ATTCG还是ACTCG),却难以了解到目标TF与TFBS之间的结合程度(TFBS能与TF结合多久,结合多牢固等信息)。
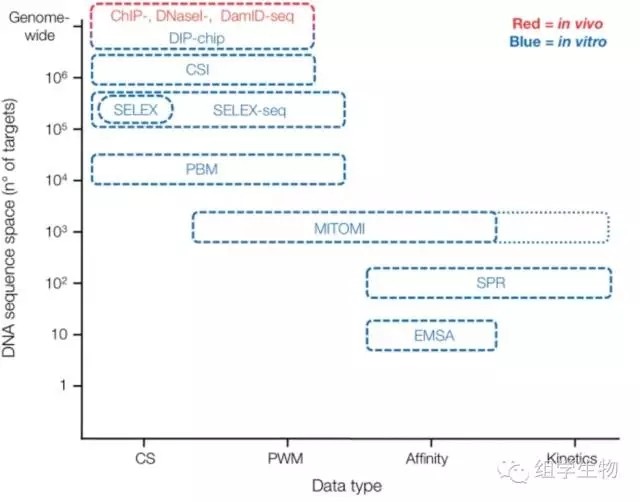
图2.多种研究方法的比较 (CS:Consensus Site,PWM: Position Weight Matrices)
3.2.1.1 SELEX技术
Systematic evolution of ligands by exponentialenrichment (SELEX) 是其中最早的一种体外检测转录因子结合位点技术,这项技术在20多年前已被科学家们利用,而且它不需要已知序列信息就可以检测到新的结合位点。它的原理是(图3a):1. 提取并纯化转录因子;2. 随机打断的DNA文库与目标转录因子孵育;3. 提取与TF结合的DNA,进一步PCR扩增片段;4. 把PCR扩增的片段重复与TF孵育,最终筛选出高结合率的DNA。SELEX技术不需要已知的DNA片段信息就可以检测新TFBS,但不足之处在于它只能找出结合率高的TFBS,因此鉴定效率较低。
为了解决鉴定效率较低的问题,高通量测序技术可以联合SELEX筛选TFBS。关联高通量技术后,SELEX-seq只需要一轮筛选而不像之前需要多轮筛选,但它的问题在需要大量的高质量纯化蛋白以及前期实验操作复杂。
3.2.1.2 Protein binding Microarrays (PBM) 技术
PBM技术(图3b)首先固定双链dna列阵在芯片当中,然后加入感兴趣的目标蛋白,孵育洗脱非结合蛋白后,加入带有荧光标记的抗体靶向目标蛋白,最后通过荧光信号鉴别蛋白-DNA结合关系。荧光强度会最终换算为Position Weight Matrices(PWM),从而计算DNA的结合序列。
3.2.1.3 DIP-seq
DIP-seq(图3c)是另外一种体外检测转录因子与DNA关系的技术。与PBM不同,这种技术不需要预先固定DNA,它是通过检测染色质DNA来实现目标鉴定。大体的实验流程可分为:1. 利用甲醛交联转录因子与DNA片段;2. 切断DNA为100bp到500bp的片段;3. 通过转录因子特异性抗体,富集与TF结合的DNA;4. 去交联后,富集DNA进行芯片或高通量测序。
3.2.1.4 ChIP-seq
ChIP-seq的实验过程与之间介绍的DIP-seq非常类似,唯一不同是,ChIP-seq可以通过甲醛原位交联TF与DNA,而DIP-seq只能在体外交联。ChIP-seq的优点是更高的分辨率,更低的噪音等。但它的弊端在于:1. 过于依赖蛋白数量;2. 依赖于交联效率;3. 抗体特异性及获取可能性;4. 难以分辨直接结合还是间接结合的TF。
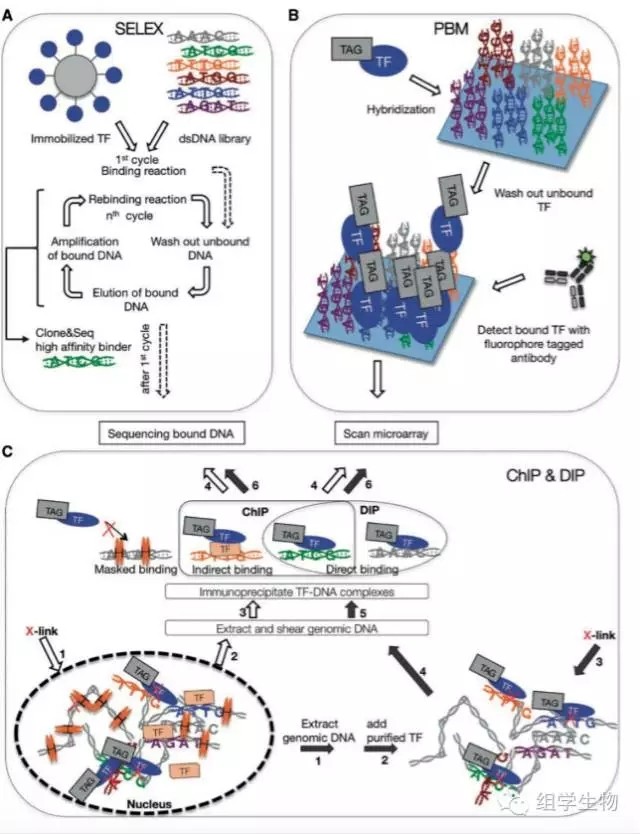
图3. 多种主流TF鉴定技术的操作流程
3.2.1.5 mechanically induced trapping of molecular interactions (MITOMI)
MITOMI是少数可以测量TF与DNA解离系数Kd的实验技术,它具有中等通量,但却不适合用于denovo的TFBS鉴定。MITOMI具体流程图4:
1. 将感兴趣的TF固定在玻璃上,接通微流管,倒入DNA;
2. DNA结合到固定的TF上;
3. 富集结合的DNA,并洗脱未结合的片段;
4. 通过富集的DNA数量计算蛋白-DNA解离系数Kd。
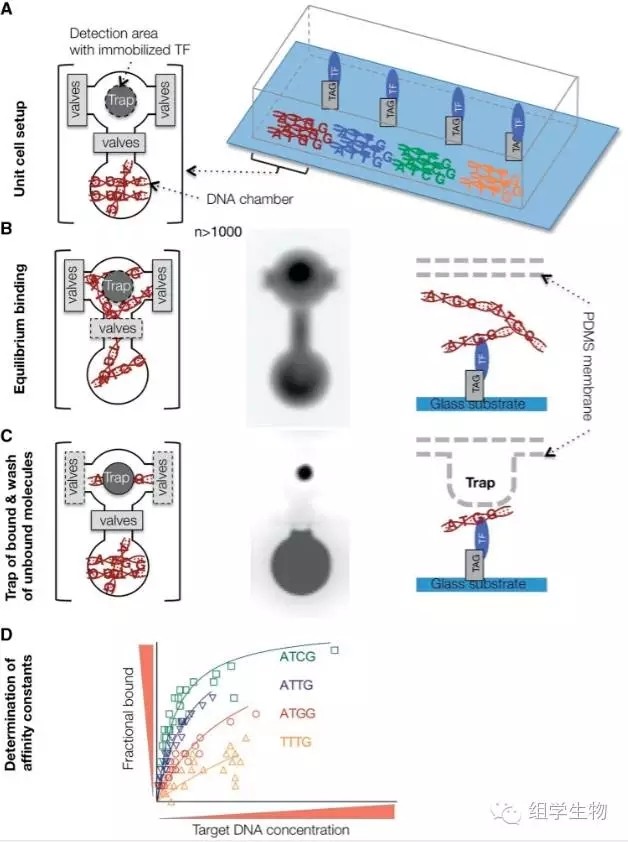
图4. MITOMI原理示意图
3.2.2通过已知TFBS寻找未知TF
比较常见的实验研究方法是酵母单杂交(Yeast one Hybrid,Y1H)及被称为反向ChIP实验的PICh(Proteomicsof IsolatedChromatin segments)。
3.2.2.1酵母单杂交(Y1H)
酵母单杂交能筛选到与DNA结合的蛋白质,并可直接从基因文库中得到编码该蛋白质的核苷酸序列,而无需复杂的蛋白质分离纯化操作,故在蛋白研究中,具有一定的优势;而且,酵母属真核细胞,通过酵母系统得到的结果,比其他体外技术获得的结果,更能体现真核细胞内基因表达调控的真实情况。主要实验过程为:
A.把感兴趣的TFBS序列(Bait)插入报告基因上游,并将带有TFBS与报告基因的质粒整合到酵母基因组中,构建含有报告基因的酵母;
B.提取mRNA构建转录因子cDNA文库,并将文库与酵母激活因子融合相连,构建“Prey”文库。把带有文库的质粒转入到步骤A的酵母中。
C.条件筛选生长酵母,如果TF与TFBS有结合,即报告基因可以顺利表达。
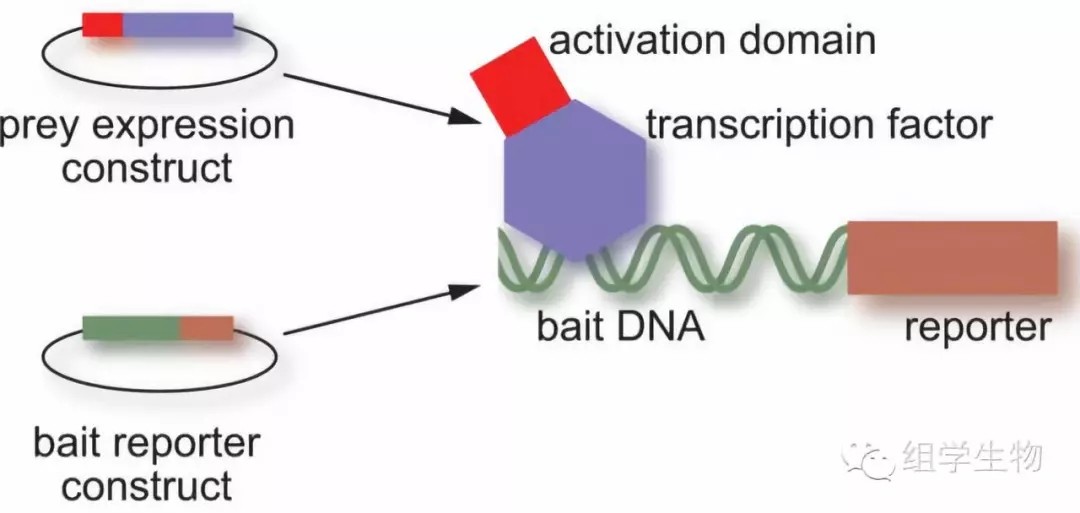
图5.酵母单杂交技术原理
3.2.2.2PICh实验
PICh,又被称为反向ChIP实验,实际是通过分离的感兴趣DNA片段富集转录因子,最终利用质谱技术鉴定所富集的蛋白质。PICh所分离的TF难以被识别为直接还是间接结合因子,同时,现阶段来说,这项技术的通量相对较低,不适合大批量筛选TF。
注意事项:
A. 体外实验虽然可以鉴别很多蛋白-DNA相互作用,但由于生物体复杂性(共调控因子,竞争性转录因子等),这些体外识别的相互作用难以在体内重复或发现。
B. 体内实验比较真实地还原转录因子-DNA之间的作用,却难以鉴别出直接还是间接作用关系。
C. 利用PBM方法可以完成denovo TFBS鉴别。
D. 检测通量一般受限于蛋白质的纯化质量及数量。
E. 大多数方法难以分析解离系数,因此对DNA或蛋白质的洗脱难以把握。
F. 除了实验方法,通过计算机技术鉴别结合位点的保守性从而预测新的转录因子目标区域已经被大量使用。但无论何种算法,这些预测都过于简单化TF-DNA之间的作用关系,从而造成极高的预测假阳性率。
G. (1) ChIP,HT-SELEX或PBM方法的目的在于发现结合序列或PWM;(2) MITOMI分析可用于进一步分析定量信息;(3) ChIP实验可用于分析体内结合信息。
写到这里,小编能想到的方法也介绍完了,如果哪位读者有更好的方法不妨分享一下,大家共同学习进步,如有不到之处敬请各位见谅。
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
6. NCBI数据上传
- 发表于 2018-06-20 10:13
- 阅读 ( 16562 )
- 分类:转录组
你可能感兴趣的文章
- 有参转录组数据分析镜像升级v2.0 2450 浏览
- 2区纯转录组文章如何写作? 5024 浏览
- 转录组扫盲系列--转录组如何建库? 15280 浏览
- 6个转录组发了BMC genomics,人家是怎么写的? 6354 浏览
- 转录组必备说明书! 12233 浏览
- 转录组结果解读及分析技巧 17281 浏览
