转录组测序分析数据库介绍
目前在对转录组数据进行分析的时候,通常使用的数据库有:
|
GO |
http://www.geneontology.org |
|
KEGG |
http://www.genome.jp/kegg/ |
|
NR |
ftp://ftp.ncbi.nih.gov/blast/db |
|
SwissProt |
http://web.expasy.org/docs/swiss-prot_guideline.html |
|
COG |
http://www.ncbi.nlm.nih.gov/COG |
|
Pfam |
http://pfam.xfam.org/ |
其中我们最常用来进行转录组分析的是GO和KEGG两个数据库。
GO数据库
什么是GO数据库?
GO(Gene Ontology, http://www.geneontology.org)数据库由基因本体论联合会建立,该数据库将全世界所有与基因有关的研究结果进行分类汇总。对不同数据库中关于基因和基因产物的生物学术语进行标准化,对基因和蛋白功能进行统一的限定和描述。
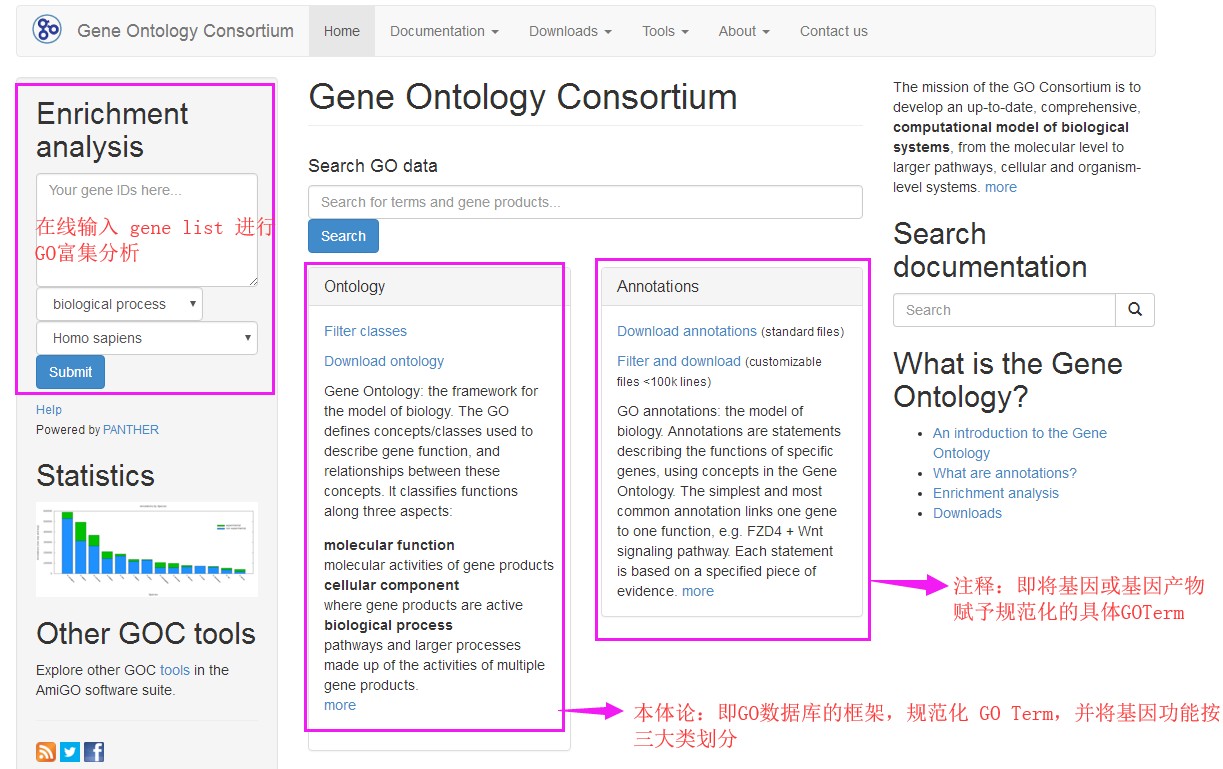 利用GO数据库,可以在以下三个方面对基因和基因产物进行分类注释。
利用GO数据库,可以在以下三个方面对基因和基因产物进行分类注释。
BP:Biological Process,生物过程
MF:Molecular Function,分子功能
CC:Cellular Component,细胞组分
在这三个大分支下面又分很多小层级(level),level级别数字越大,功能描述越细致。最顶层的三大分支视为level 1,之后的分级依次为level 2,level 3,level 4依次类推。通过GO注释,可以大致了解某个物种的全部基因产物的功能分类情况。
GO数据库的术语形式是什么?
GO定义的术语具有有向无环性(directed acyclic graphs,DAGs)的特点,而并非是传统的等级制定义方式。
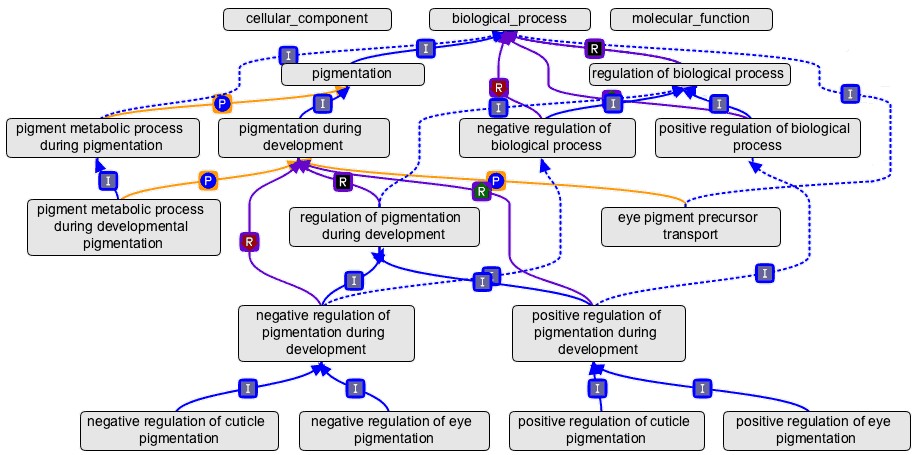
不理解有向无环没关系,这里给大家举个例子:

植物中有一个生物学途径叫做己糖合成,它的上一级为己糖代谢和单糖合成。当转录组数据中某个基因被注释为“己糖合成活性”后,它自动地获得了己糖代谢和单糖合成地注解。因为在GO中,每个术语必须遵循“真途径”法则,即如果下一代的术语可以用于描述此基因产物,其上一代术语也可以适用。
GO有向无环图里有很多箭头,每种箭头都具有不同的含义。
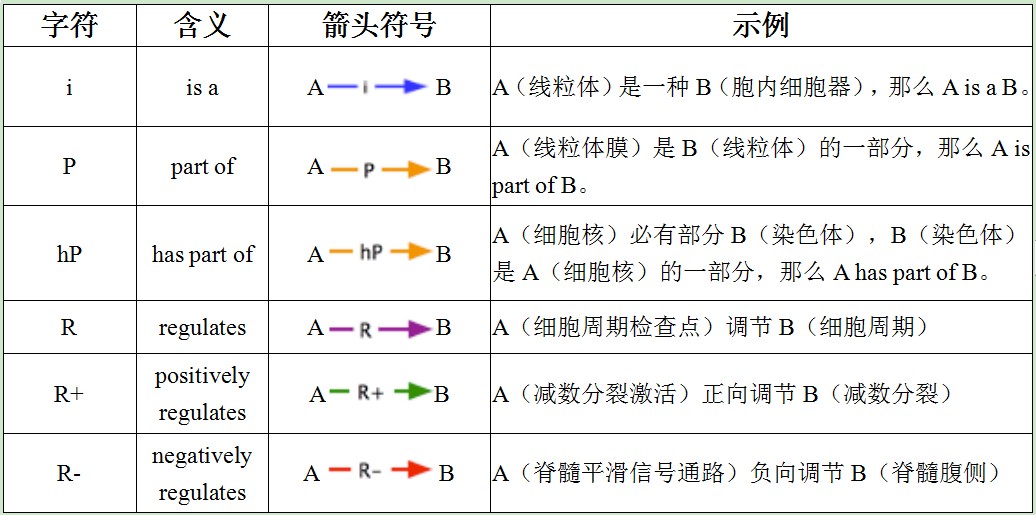
此外,箭头也具有导向性。例如线粒体(A)是细胞质(B)的一部分,细胞质又是细胞(C)的一部分,从而推导出:线粒体(A)是细胞(C)的一部分。
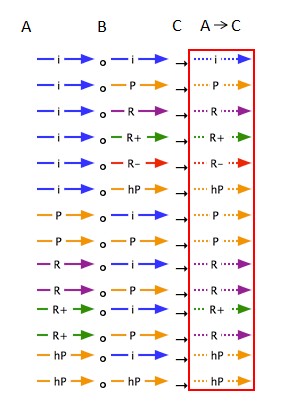 所有推导的结果箭头都以虚线表示,其他箭头导向性关系可以参考下面这张图。
所有推导的结果箭头都以虚线表示,其他箭头导向性关系可以参考下面这张图。
KEGG数据库
什么是KEGG数据库?
KEGG(Kyoto Encyclopedia of Genes and Genomes,http://www.genome.jp/kegg/)数据库是一个系统分析基因功能、联系基因组信息和功能信息的大型知识库。在生物体内,基因产物并不是孤立存在起作用的,不同基因产物之间通过有序的相互协调来行使其具体的生物学功能。因此,KEGG数据库中丰富的通路信息将有助于我们从系统水平去了解基因的生物学功能。通过与KEGG数据库比对,获得基因或转录本对应的KO编号,根据KO编号可以获得某基因或转录本可能参与的具体生物学通路情况。
KEGG数据库分为系统、基因组、化学和健康信息四大类,细分为以下16个主要的数据库,并用不同的颜色进行区分。
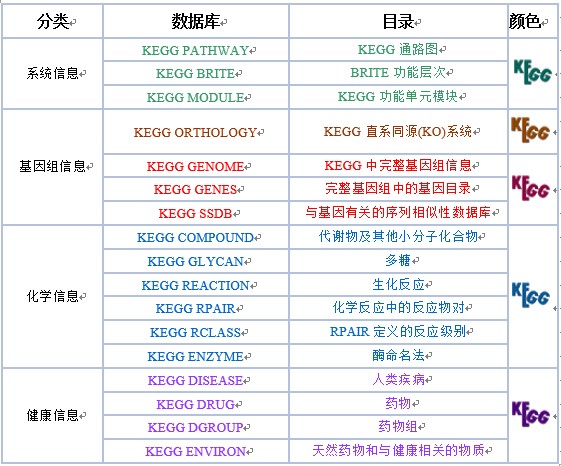
Genetic Information
Processing (遗传信息处理)
Metabolism (新陈代谢)
Environmental Information Processing (环境信息处理)
Cellular Processes (细胞过程)
Organismal Systems (生物系统)
Human Diseases(人类疾病)
Drug Development(药物开发)
其中新陈代谢通路是手工画出来的,其余几大分类都是通过计算机进行绘制的。
KEGG Pathway怎么看?
一般,KEGG中存在两种代谢图:Reference pathway,根据已有的知识绘制的、概括的、详尽的具有一般参考意义的代谢图,为白色小框,在KEGG中名字以map开头。另一种为Species-specific pathway,绿色小框为该物种特有的基因或酶,只有这些绿色的框有更详细的信息,KEGG中名字为物种种属英文缩写开头。
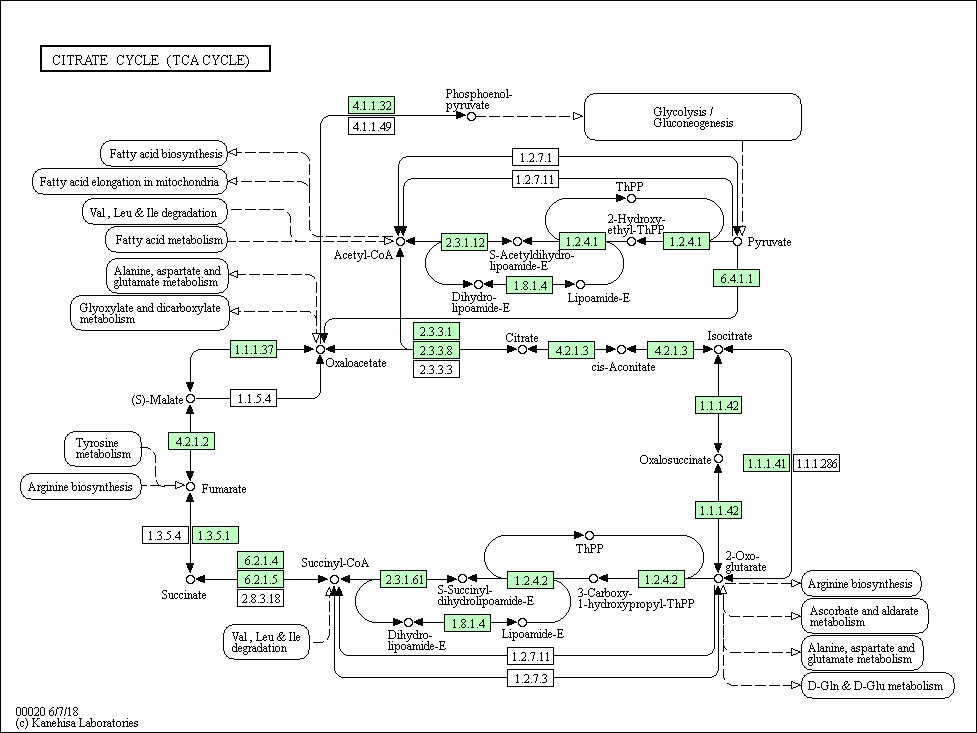
这是一张人类的KEGG Pathway中TCA循环的通路图。里面包含大量蛋白、代谢物的信息,以及展示它们之间的相互作用。在这张代谢通路上的基因或蛋白框、代谢产物可以点击,点击之后会给出对应详细的信息。
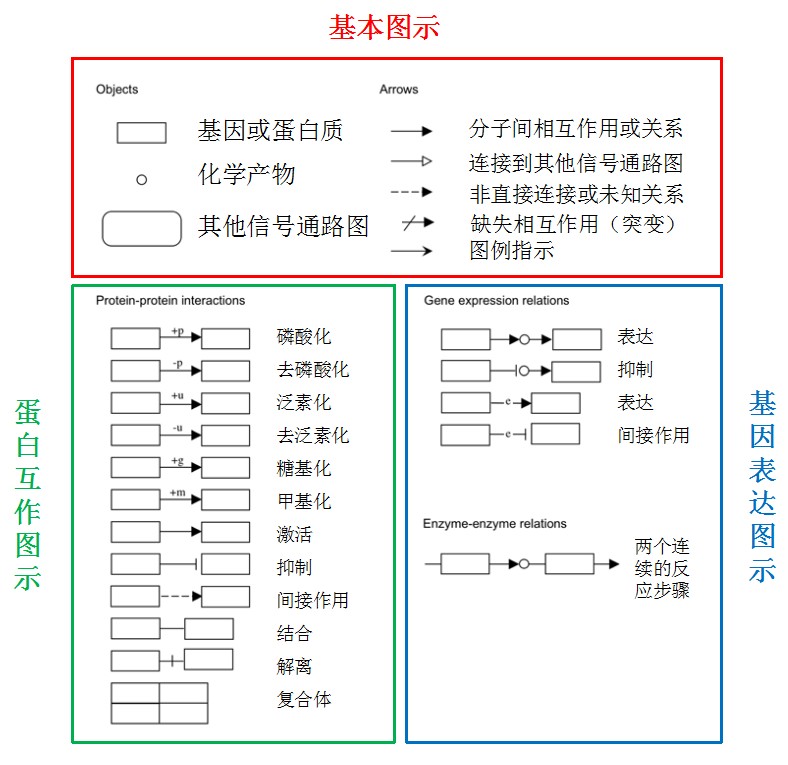
到这里有些小伙伴可能看不明白了。这些里的条条框框代表什么东西,感觉很复杂的样子。

这里我们就来看下图里这些东西代表什么。图示可以分为三大类,基本图示、蛋白互作图示、基因表达图示,他们的意义如下所示:
当我们通过KEGG官网搜索某一Pathway通路时,将鼠标静置在某一图例上,会加载出详细信息
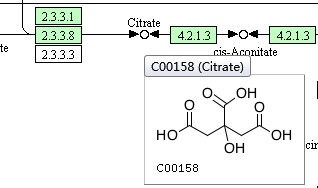
点击还可以进入详细界面,可以看到某一基因、蛋白或代谢产物的详细信息。
NR数据库
NR(Non-Redundant Protein Sequence Database)数据库是非冗余蛋白库,序列来源于GenPept、Swissprot、PIR、PDF、PDB和NCBI RefSeq数据库。对于所有已知的或可能的编码序列,NR都给出了相应的氨基酸序列(WP编号为已确定的蛋白序列;XP编号为计算机软件预测的蛋白序列)和对应的蛋白数据库序列号。简单来讲,NR相当于一个将核酸数据和蛋白数据联系起来的目录。
完整的NR数据库的蛋白序列和预先构建好的blast索引可以从NCBI的ftp (https://ftp.ncbi.nlm.nih.gov/blast/db/FASTA/)服务器上下载得到,但通常情况我们不选择直接下载数据库,而是选择下载其索引(ftp://ftp.ncbi.nih.gov/blast/db),这是因为NR数据库十分庞大,自己构建索引费时费力。而NCBI提供的blast索引在构建时已经加入序列的tax id,由此可以非常方便找到对应物种的注释信息。
Swiss-Prot数据库
Swiss-Prot(https://web.expasy.org/docs/swiss-prot_guideline.html)于1986年由瑞士生物信息学研究所(SIB)和欧洲生物信息学研究所(EBI)共同协作维护,目前以合并到UniProt数据库中 (The Universal Protein Resource,http://www.uniprot.org/),最新更新的氨基酸序列有558,898条,Swiss-Prot能提供详细的蛋白质序列、功能信息,如蛋白质功能描述、结构域结构、转录后修饰、修饰位点、变异度、二级结构、三级结构等,同时提供其他数据库,包括序列数据库、三维结构数据库、2-D凝聚电泳数据库、蛋白质家族数据库的相应链接。

COG数据库
COG(Cluster of Orthologous Groups of proteins),直系同源蛋白簇的缩写,该数据库认为构成每个COG的蛋白都是被假定为来自于一个祖先蛋白。根据系统进化关系,对细菌、藻类和真核生物,几十个完整基因组的编码蛋白,进行分类构建而成,将基因分成了25类。当前常用的COG包含COG和KOG两个子数据库:
COG对原核生物的同源蛋白进行聚类,适合原核生物的COG注释;KOG对真核生物的同源蛋白进行聚类,适合真核生物的COG注释。
但从2003年至2014年NCBI COG就一直未更新,KOG更是在2003年就停止更新了,所以目前我们更多使用的是EggNOG数据库,这是由欧洲分子生物学实验室(EMBL,European Molecular Biology Laboratory)建立的。继承了NCBI COG的衣钵,极大的扩展了基因组信息。升级后的eggNOG 提供了更细致的 OG 分析,可根据物种所属的clade选择参考数据集,可以有效的降低计算量,另一个特色还提供了与其它注释信息(KEGG/GO/SMART/PFAM) 的关联。
Pfam数据库
Pfam(http://pfam.xfam.org/)是一个大型的蛋白家族数据库,最新版本的Pfam数据库(2018年9月更新32.0版本)包含了17929个蛋白质家族注释及基于隐马尔科夫模型(Hidden Markov models,HMMs)的多序列比对信息。
Pfam可以为我们提供精确的蛋白家族及结构域分类,因此常被用于查询蛋白家族及蛋白结构域功能注释。Pfam包含两Pfam-A和Pfam-B两个家族数据库。Pfam-A来自基础序列数据库Pfamseq,是根据最新的UniProtKB(SwissProt)数据库建立,其质量及可信度相对较高。Pfam-B是一个未获得注释的低质量数据库,虽然其质量较低,但对于鉴定Pfam-A无法覆盖到的部分也可以起到补充作用。
以上数据库就简单介绍到这里啦~
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课,学习链接:linux系统使用、perl入门到精通、perl语言高级、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章,学习链接:TCGA-差异基因分析、GEO芯片数据挖掘、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他课程链接:二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读。
- 发表于 2019-03-25 11:08
- 阅读 ( 13738 )
- 分类:转录组
你可能感兴趣的文章
- Broad GDAC Firehose—TCGA数据分析中心 4303 浏览
- cBioportal使用教程 4620 浏览
- HPA:人类蛋白图谱数据库 4364 浏览
- PlantPAN数据库 4268 浏览
- TCGA数据库挖掘-肾细胞癌相关biomiarker筛选案例解析 3584 浏览
- GEO、TCGA多数据库联合挖掘胰腺导管腺癌预后关键基因 4836 浏览
