R语言统计数据范围并用ggplot2绘制柱状图
R语言,按数值范围统计频次,绘制柱状图
输入数据为一个向量,数值范围为0-100+:
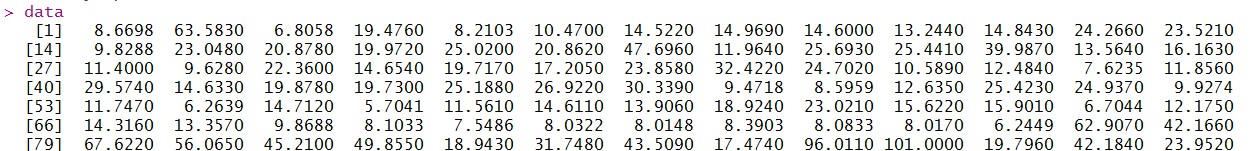
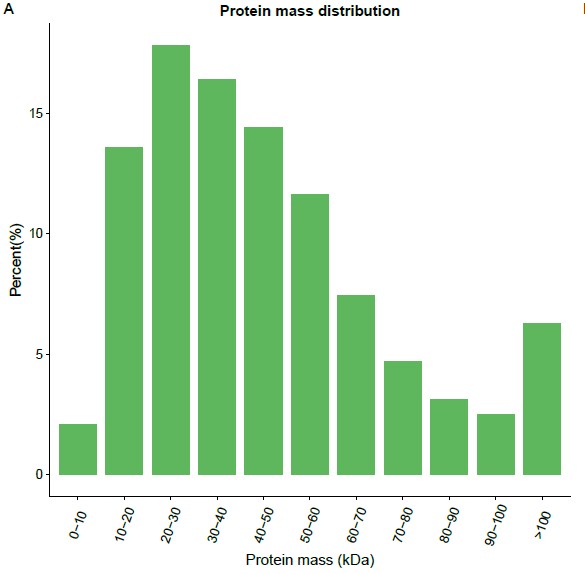
统计不同数值范围的数值分布频率,并绘制柱状图,如下所示,并把大于100的数值统计到一个柱子当中:
具体R语言代码如下:
注意本代码的主题为cowplot主题适合SCI文章发表,并设置了x轴刻度标签倾斜;
library(reshape2)
local({r <- getOption("repos") ;r["CRAN"] <- "http://mirrors.tuna.tsinghua.edu.cn/CRAN/" ;options(repos=r)})
library(ggplot2)
library(cowplot)
library(RColorBrewer)
pairjoin <- function(x){
ran=paste(head(x,-1), tail(x,-1), sep="-")
ran[length(ran)]=">100"
ran
}
# use the base hist() function to calculate BINs ,超过100的数值归类到一起;
data[data>100]=101
MassStat <- with(hist(data, breaks=seq(0, 110, by = 10), plot=FALSE),
data.frame(N=counts, Mass=pairjoin(breaks), PCT=counts/sum(counts)))
MassStat
MassStat$Mass=factor(MassStat$Mass,levels = MassStat$Mass,order=T)
pm=ggplot(data=MassStat, aes(x=Mass, y=PCT*100)) +
geom_bar(fill="#4DAF4A",alpha = .9, stat="identity",width=0.8) +
guides(fill=FALSE)+
theme(legend.key = element_blank(),legend.title = element_blank(),
axis.text.x = element_text(angle=70, vjust=0.5)
)+ xlab("Protein mass (kDa)")+ylab("Percent(%)") +ggtitle("Protein mass distribution")
- 发表于 2018-07-01 15:33
- 阅读 ( 9761 )
- 分类:R
你可能感兴趣的文章
- 多组学联合分析—桑基图 362 浏览
- 一文教你绘制GO富集分析弦图,简单又好看 364 浏览
- R脚本绘制多层环形饼图 2791 浏览
- corrplot包绘制相关性图,简单又好看! 2914 浏览
- 已知集合交集数量绘制韦恩图 3414 浏览
- 绘制Upset 图 5927 浏览
相关问题
- linux 下 R 命令怎么后台运行 2 回答
- rJava 安装包错 1 回答
- R包安装 1 回答
- R语言绘制中国地图,每个省份出现多边形 1 回答
- R 更新后报错 0 回答
- 按照课程里的代码用R分析TCGA数据差异基因的KEGG富集,发生报错 1 回答
0 条评论
请先 登录 后评论
