筛选VCF中指定位置变异位点
筛选VCF中指定位置变异位点
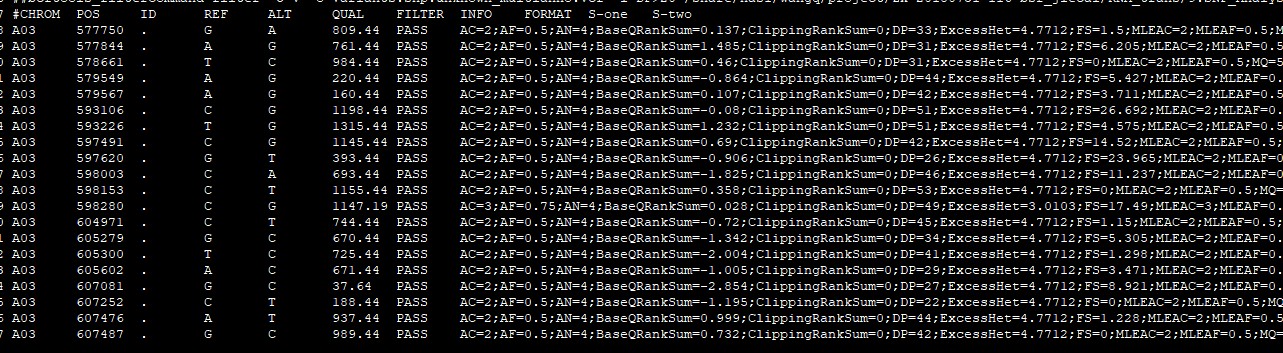
在做DNA重测序,BSA等项目都会遇到VCF文件,有时候会需要挑选出某些区域的变异信息。这里有简单的方法可以实现。
我们需要用到vcftools这款软件,它是专门处理VCF文件的工具,只需要一条命令就可以完成。
vcftools --vcf Variants.snp.unknown_multianno.vcf --chr A03 --from-bp 577700 --to-bp 607700 --out out_prefix --recode --recode-INFO-all
这里解释一下各个参数
--vcf 后面跟的是vcf文件
--chr 后面跟筛选区域所在的染色体
--form-bp 后跟筛选区域的起始位置
--to-bp 后跟筛选区域的终止位置
--out 输出文件的前缀
--recode 没有此参数则不会输出
- 发表于 2018-08-10 14:25
- 阅读 ( 8618 )
- 分类:软件工具
你可能感兴趣的文章
- SNP 过滤标准保证数据质量及参考文献: 3901 浏览
- 提取 VCF 文件中的基因型信息 9781 浏览
- 对vcf文件排序 4157 浏览
- 变异结果 vcf 格式转换成 hapmap格式 11495 浏览
- 看懂变异记录结果文件(VCF) 32879 浏览
0 条评论
请先 登录 后评论
