GFF文件格式不标准,第三列只有mRNA处理方法
GFF文件格式不标准,第三列只有mRNA处理方法
gff文件第三列一般有gene,mRNA,CDS,exon等等信息,但是有时候没有gene信息,只有mRNA信息,这种情况一般是一个基因只注释了一个转录本的基因组,这个时候我们可以批量的处理一下,添加上gene行信息,也就是mRNA信息直接用于基因信息,方便我们的分析流程处理数据:
可以用awk命令批量添加gene行,把mRNA的ID作为基因ID,并且在mRNA行添加Parent信息:
awk -F"[\t=]" 'BEGIN{OFS="\t"}{if($3=="mRNA"){print $1,$2,"gene",$4,$5,$6,$7,$8,$9"="$10 ; print $1,$2,$3,$4,$5,$6,$7,$8,$9"="$10"Parent="$10;}else{print $0}}' PO.gene.gff >PO.gene.gff1
最终结果如下,左边为原始gff文件,右边为更改后的文件,有遇到类似问题的小伙伴可以根据命令来修改一下:
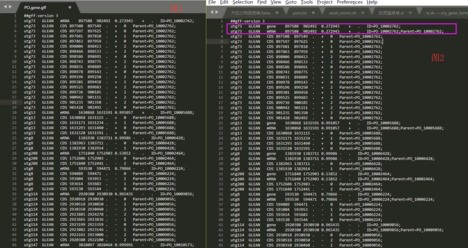
更多AWK命令使用技巧见:linux系统使用
生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高
- 发表于 2019-05-07 15:48
- 阅读 ( 9685 )
- 分类:linux
你可能感兴趣的文章
- linux fail2ban-server 服务 添加信任IP 1201 浏览
- 文件夹名称中存在空格,scp如何正确识别 1671 浏览
- Linux命令处理文件合并 1422 浏览
- GFF或GTF格式转bed 2828 浏览
- awk正则表达式使用外部变量 2721 浏览
- quota 显示的space 与du显示的不一致,导致文件上传失败 1874 浏览
相关问题
- 搜集的gff3文件;第一列不同,有的是chr,有的是id;这个影响吗? 2 回答
- 老师,我在做饭基因家族分析,我搜集的基因组的gff3文件,第一列有的是chr有的是基因id,这个会影响后续结果嘛? 2 回答
- 老师,为什么我的cds和pep文件导不出来呀?导出来报错,并且文件内容无。最后一张照片时我退出重进后的历史命令 2 回答
- 基因家族分析2.0,做基因结构分析时报错,完全按照提供指令做的 1 回答
- 老师,我这个是桑树的fasta和gff文件,不是从官网下载的。假如各个网站都找不到我想要的物种的基因组文件,只有手头这个文件,我要怎么做才能和教程的代码连接上呀?或者说碰到这种目标物种查不到的情况可以怎么做呢? 1 回答
- 老师,下面是报错、染色体信息、gff文件中的染色体和命令行 2 回答
1 条评论
请先 登录 后评论
