PubMed文献按影响因子排序
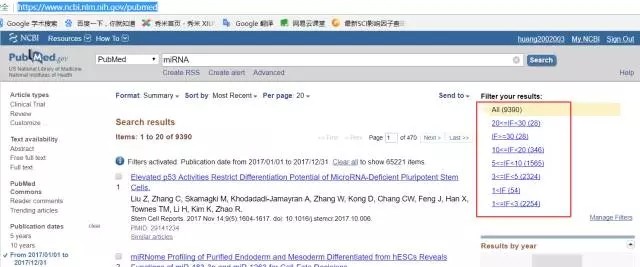
搜素文献,很多人都需要用到PubMed,功能非常强大,绝大多数的文献都能搜索得到,但是pubmed的高级功能可能很多人还不知道->那就是自定义筛选条件:最好的应用就是将文献按照影响因子进行分类,如下图所示:
做到这个只需简单三步就能完成:
注册NCBI账号 :(如果有账号直接登录)https://www.ncbi.nlm.nih.gov/account/
进入PubMed 网页 https://www.ncbi.nlm.nih.gov/pubmed
制作过滤器,保存并激活
最关键的一步就是制作过滤器:
首先,随便在pubmed中搜素一下找到:manager filters 点击进入(在右下角红框框处),当然我的IF已经设置好了。
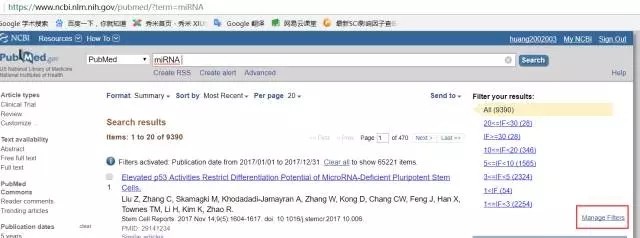
其次,进入之后按下面步骤建立filter,关键为step2 制作过滤条件:
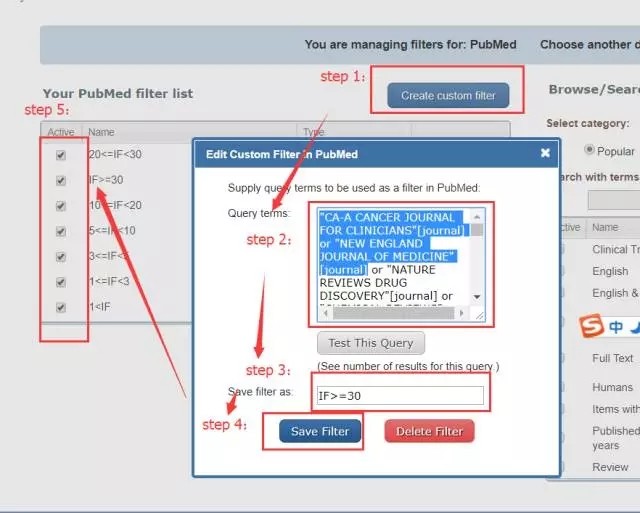
在step2中,我们输入影响因子大于30的杂志的名称,格式为:把杂志名称用双引号引起来,后面跟一个[journal] 然后or 再跟杂志一个杂志,直到把所有影响因子大于30的杂志用完。如下所示:
"CA-A CANCER JOURNAL FOR CLINICIANS"[journal] or "NEW ENGLAND JOURNAL OF MEDICINE"[journal]
添加完影响因子大于30的之后,再重复上述4步把其他影响因子范围的杂志分别建立到不同的filter当中,这样自定义的过滤器就完成了;最后勾上激活过滤器;
大功告成,最麻烦的就是把不同影响因子的杂志归类整理了,这个繁琐的过程既然我们已经做了,就分享给大家,下面有链接分别整理到了不同的TXT文本文件当中,只要复制粘贴就可以,省去制作过滤条件的麻烦;
百度网盘地址:http://pan.baidu.com/s/1kU9aST5
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读、
- 发表于 2018-04-22 14:45
- 阅读 ( 25081 )
- 分类:软件工具
你可能感兴趣的文章
- python代码到pubmed上搜索关键字,统计每年发文章数量 1333 浏览
