ggplot2点图geom_point中aes(shape)映射
此前在相关文章中详细介绍了关于geom_point 在设定shape时可以进行多少设置,每种出现的结果:
https://www.omicsclass.com/article/475
区别与点shape设定,点的shape 映射必须转化成因子,连续变量无法mapped to shape,譬如基于上一文章相同数据dat
dat
X Y shape
1 1 6 0
2 2 6 1
3 3 6 2
4 4 6 3
5 5 6 4
6 1 5 5
7 2 5 6
8 3 5 7
9 4 5 8
10 5 5 9
11 1 4 10
12 2 4 11
13 3 4 12
14 4 4 13
15 5 4 14
16 1 3 15
17 2 3 16
18 3 3 17
19 4 3 18
20 5 3 19
21 1 2 20
22 2 2 21
23 3 2 22
24 4 2 23
25 5 2 24
26 1 1 25
进行绘图将shape列数据映射到aes(shape)
library(ggplot2)
p=ggplot(dat,aes(x=X,y=Y,shape=shape))+
geom_point(size=20)
print(p)
 将直接报错,注意先转化因子:
将直接报错,注意先转化因子:
dat$shape=factor(dat$shape)
p=ggplot(dat,aes(x=X,y=Y,shape=shape))+
geom_point(size=10)
print(p)
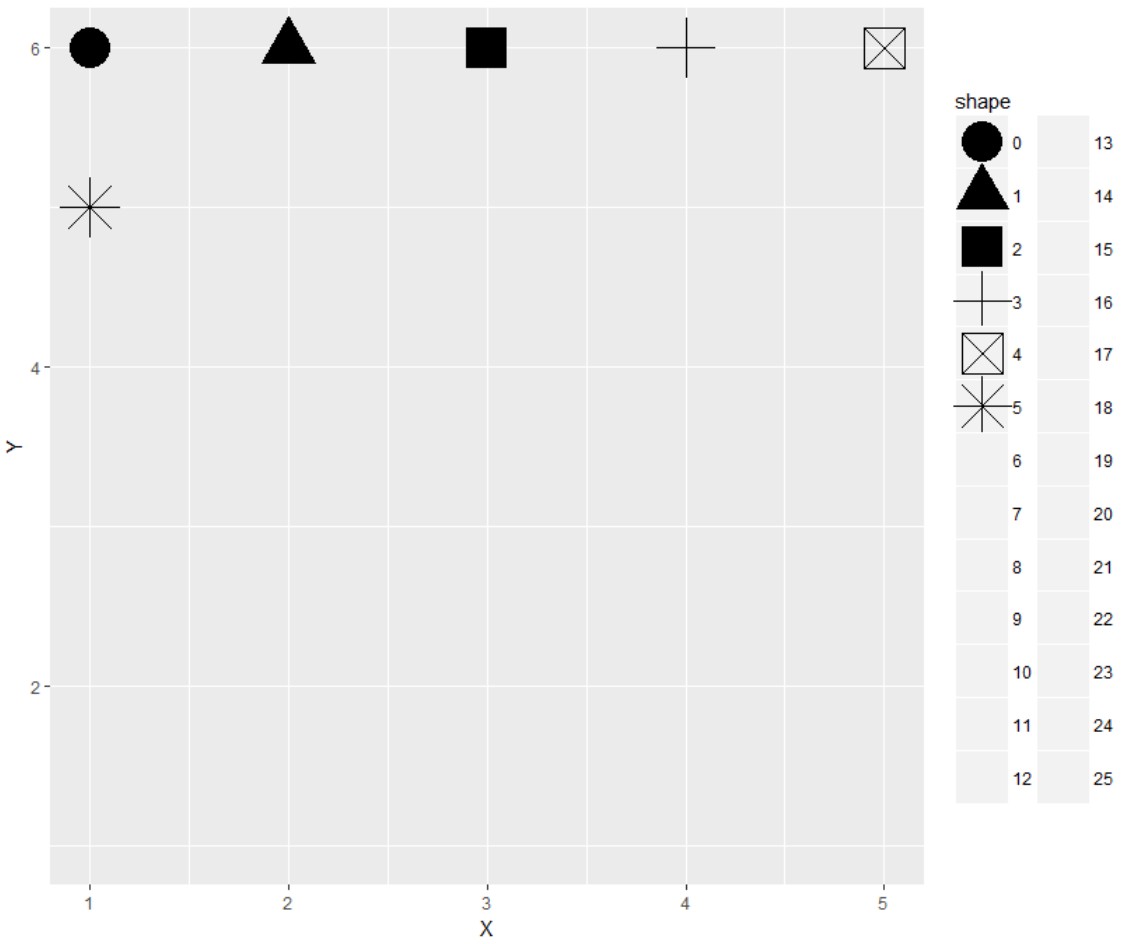
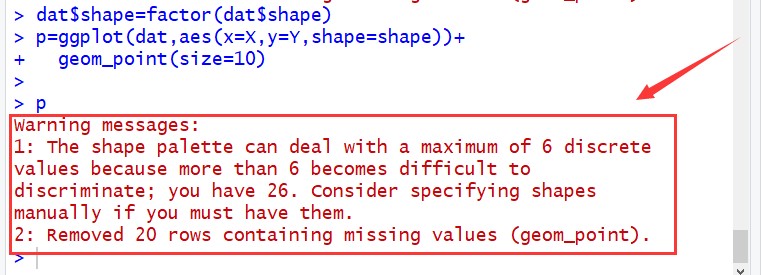
而由结果图片明显能知道shape有效的映射,仅6个,前六个因子有效,而之后的因子并不能出现其他shape(对应因子“0”、“1”,“2”...而不是数字0、1、2...)
故在进行数据整理的过程中需要注意基于shape进行映射对数据进行区分不应超过六个分类
如果想提升自己的绘图技能,我们推荐:R语言绘图基础(ggplot2)
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-10-12 15:07
- 阅读 ( 7738 )
- 分类:R
你可能感兴趣的文章
- 不均等坐标系绘制 2749 浏览
- ggplot 柱状图,y轴从0 开始,最高柱子 留有余地代码: 2974 浏览
- cnv_dumbbell_plot.r 绘制哑铃图ggplot2包 3264 浏览
- 绘制展示基因在样本中表达量与数量的柱状图 4778 浏览
- ggplot中用字符窜传递变量名称列名等 3011 浏览
- ggplot2中各种主题元素修改对应方法函数速查表 5188 浏览
