单细胞转录组数据挖掘流程记录-BCC 基底细胞癌(GSE123813)
单细胞转录组数据挖掘流程记录-BCC 基底细胞癌(GSE123813)
为方便学习我们单细胞课程的学员利用我们提供的脚本快速的分析公共数据做数据挖掘,这里开始记录一下不同公共数据相关分析shell代码。大家有了我们的代码就可以照抄分析,提高分析数据效率,今天是第1个数据,后续还有数据慢慢给大家分享:
数据介绍:
BCC 基底细胞癌(GSE123813),NCBI GEO数据下载地址:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE123813。
这个数据作者已经分析好,有原始的counts 还有注释好的metadata文件,可以使用我们的脚本直接读入:
2.单细胞转录组数据下载:
mkdir -p data/GSE123813 #创建文件夹#数据下载 wget -c "https://ftp.ncbi.nlm.nih.gov/geo/series/GSE123nnn/GSE123813/suppl/GSE123813%5Fbcc%5Fall%5Fmetadata.txt.gz" -O GSE123813_bcc_all_metadata.txt.gz wget -c "https://ftp.ncbi.nlm.nih.gov/geo/series/GSE123nnn/GSE123813/suppl/GSE123813%5Fbcc%5FscRNA%5Fcounts.txt.gz" -O GSE123813_bcc_scRNA_counts.txt.gz
观察数据:
metadata数据:
cell.id patient treatment sort cluster UMAP1 UMAP2bcc.su001.post.tcell_AAACCTGAGCTTCGCG su001 post CD45+ CD3+ CD8_mem_T_cells -8.49122047424316 5.53352499008179bcc.su001.post.tcell_AAACCTGAGGACATTA su001 post CD45+ CD3+ CD8_ex_T_cells 4.62372446060181 6.66859436035156bcc.su001.post.tcell_AAACCTGCACGCATCG su001 post CD45+ CD3+ CD8_mem_T_cells -7.92044115066528 3.19655275344849bcc.su001.post.tcell_AAACCTGCAGATGGGT su001 post CD45+ CD3+ CD8_mem_T_cells -7.87822341918945 4.33330249786377bcc.su001.post.tcell_AAACCTGCAGTGGAGT su001 post CD45+ CD3+ Tregs -2.38209414482117 11.2608156204224bcc.su001.post.tcell_AAACCTGCATGCCTTC su001 post CD45+ CD3+ CD8_ex_T_cells 1.27268886566162 3.15348219871521bcc.su001.post.tcell_AAACCTGCATTGCGGC su001 post CD45+ CD3+ CD8_ex_T_cells 4.33470439910889 5.28554677963257bcc.su001.post.tcell_AAACCTGGTTATCACG su001 post CD45+ CD3+ CD4_T_cells -3.83144402503967 9.61049652099609bcc.su001.post.tcell_AAACCTGTCACCCGAG su001 post CD45+ CD3+ CD8_ex_T_cells 4.10303926467896 6.88553094863892bcc.su001.post.tcell_AAACCTGTCAGGTTCA su001 post CD45+ CD3+ CD8_mem_T_cells -9.1302433013916 3.4471583366394
count数据:
bcc.su001.pre.tcell_AAACCTGCAGATCGGA bcc.su001.pre.tcell_AAACCTGCAGGGATTG bcc.su001.pre.tcell_AAACGGGCATAGACTC bcc.su001.pre.tcell_AAACG>FO538757.2 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0>AP006222.2 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0>RP11-206L10.9 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0>LINC00115 1 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0>FAM41C 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0>NOC2L 0 0 0 0 0 0 0 0 2 1 0 0 0 0 1 0 0 0>KLHL17 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1>PLEKHN1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0>HES4 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0>
观察数据主要注意的地方:
1)metadata列分割是tab还是空格分隔开,第一列的细胞名称和count矩阵的列名也就是细胞名称是否对应。
2)count矩阵的保存格式:表格?10X?h5?等格式,不同格式用不同的参数读入。
3.单细胞数据分析:
这个数据整理的还是挺标准的,可以用我们的R脚本直接读入分析:
scripts=~/single-cell/scripts/ prefix=GSE123813 outdir=BCC_GSE123813 mkdir BCC_GSE123813.1 #数据读入 Rscript $scripts/seurat_sc_qc.r --count GSE123813_bcc_scRNA_counts.txt.gz --project BCC_GSE123813 \ --nUMI.min 500 \ --nUMI.max 40000 \ --nGene.min 250 \ --mito.gene.pattern "^MT.*-" \ --percent_mito 10 \ --log10GenesPerUMI 0.8 \ -o 01.qc -p GSE123813 --metadata GSE123813_bcc_all_metadata.txt.gz #降维聚类分析 Rscript $scripts/seurat_sc_cluster.r --rds 01.qc/GSE123813.afterQC.rds \ -p GSE123813 --resolution 0.1 -d 30 -o 02.cluster \ --vars.to.regress nUMI percent_mito --high.variable.genes 2000
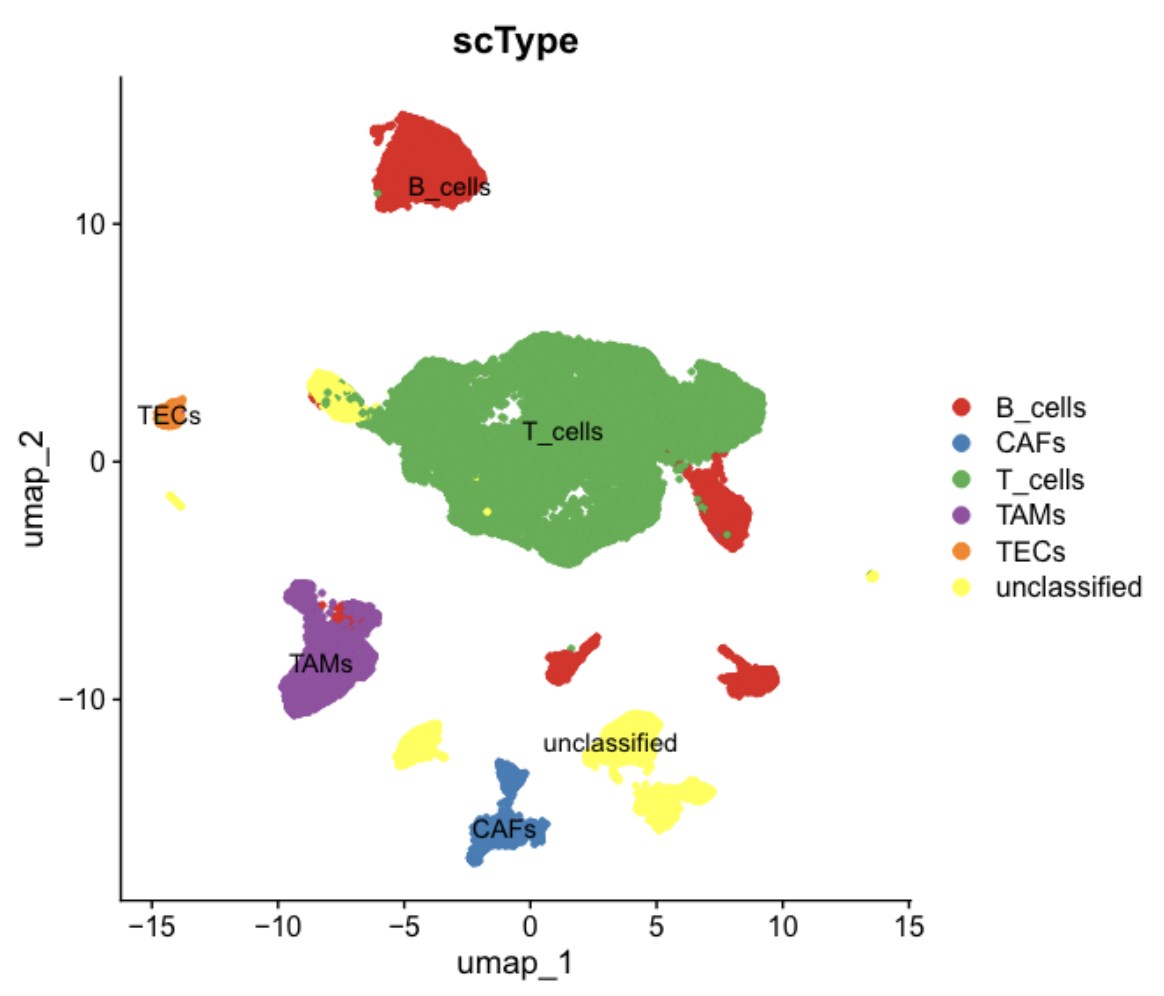
4.scType细胞分群分析结果展示
5.单细胞转录组分析课程推荐:https://bdtcd.xetslk.com/s/4i88K6

- 发表于 2024-06-11 17:24
- 阅读 ( 1648 )
- 分类:转录组
你可能感兴趣的文章
- 植物单细胞转录组文章 1723 浏览
- 拟南芥叶单细胞分析流程 1567 浏览
- 单细胞转录组镜像代码更新4.0 2225 浏览
- 单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919) 2457 浏览
- 非负矩阵分解(cNMF) 4132 浏览
- 单细胞转录组如何将UCell CytoTRACE2 infercnv等结果添加到总的rds中 1774 浏览
相关问题
0 条评论
请先 登录 后评论
