关于CodeML分析中四种常见模型假设
codeml是PAML软件包下的一个程序,在估算蛋白编码序列同义替换和非同义替换速率以及检测序列是否经受正选择分析中受到广泛应用。对于一棵系统发育进化树,在考虑其不同序列间(site)和不同支系间(branch)的ω值不相同,主要分为四种模型假设。
1. 枝模型(Branch model):假设ω在系统发育树上的不同分支上存在差异。
(1)One-ratio model(Model=0):假设系统发育树中所有支系的 ω 值相等;
(2)Free-ratio model(Model=1):假设系统发育树中所有支系的 ω 值不相等;
(3)Two-ratio model(Model=2):假设前景枝和背景枝的ω 值不同;
2. 位点模型 (Site model):假设不同位点(密码子之间或蛋白的氨基酸之间)上的ω存在差异。
该模型主要用于检测正选择( ω >1)作用,共有8个不同假设的模型:
(1)M0(单一比率),即:One-ratio model,假设所有位点具有相同的 ω 值;
(2)M1a(近中性),假设仅有保守位点(0<ω <1)和中性位点( ω =1)而没有正选择位点( ω >1)存在,这两类位点的比率分别为p0和p1,其对应的ω 值分别为ω0、ω1;
(3)M2a(正选择),该模型在M1基础上增加了第三类ω值,即假设除了保守位点和中性位点外,还存在处于正选择压力下的位点( ω >1),这三类位点的比率分别为p0、p1和p2,其对应的ω 值分别为ω0、ω1和ω2;
(4)M3(离散),假设所有的位点ω 值呈简单的离散分布趋势;
(5)M7(beta),假设所有位点的 ω 属于矩阵(0, 1)并呈beta分布;
(6)M8(beta & ω ) ,该模型在M7基础上增加另一类ω 值(ω >1);
(7)M8a(beta & ω =1),与M8模型类似,但将ω 值固定为1(ω =0);
3. 枝位点模型 (Branch site model):假设不同位点的和不同支系间受的ω均存在差异(既考虑位点间也考虑支系间的 ω 值),共有四个模型Model A、Model B、Model C和Model D,主要参数如下:
(1)Model A (Model 2, NSites=2, ncatG=ignored)
(2)Model B (Model 2, NSites=3, ncatG=ignored)
(3)Model C (Model 3, NSites=2, ncatG=ignored)
(4)Model D (Model 3, NSites=3, ncatG=2 or 3)
4. 进化枝模型 (Clade Model):与枝位点模型类似,能同时检测多个进化枝(Clade),但是该模型并没有将背景支的dN/dS值约束在(0,1)。共有CmC和 CmD 两种模型,主要参数如下:
(1)CmC (Model 3, NSites=2, ncatG=2 or 3)
(2)CmD (Model 3, NSites=3, ncatG=ignored)
CodeML分析总流程:
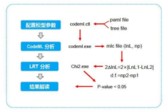 (1)配置零假设模型和备选模型的参数(codeml.ctl);
(1)配置零假设模型和备选模型的参数(codeml.ctl);
(2)运行CodeML程序进行分析获得对应的LnL和np值;
(3)通过似然率检验(LRT)(卡方检验)获得p值确定零假设模型和备选模型之间是否存在差异;
(4)根据结果进行解读
- 发表于 2022-10-10 15:41
- 阅读 ( 5590 )
- 分类:软件工具
你可能感兴趣的文章
- 关于CodeML分析中Branch-Site模型假设 3959 浏览
