胃癌预后signature数据挖掘-自噬基因
背景:自噬是真核细胞中一种高度保守的细胞生物学行为,是细胞内包裹细胞质成分的双层膜结构形成并与溶酶体结合产生自我吞噬性质的一种分解代谢过程。自噬的分解代谢作用可以清除细胞内的毒性物质,维持细胞内环境稳态,同时在这一过程中产生的氨基酸等小分子物质可被细胞代谢循环利用,为细胞的生存提供营养。自噬维持细胞内环境稳态的作用能抑制肿瘤的发生;但在已完成恶性转化的肿瘤细胞中,自噬能在不利于肿瘤生存的微环境内维持肿瘤细胞的基础代谢,保持细胞活性。因此,自噬发挥双重作用,是初始肿瘤发生的抑制剂,然后成为肿瘤进展的促进剂,自噬在癌症的发生、发展和对治疗的抵抗中起着至关重要的作用。然而,很少在胃癌(GC)中分析自噬相关基因(ARG)。本研究的目的是使用生物信息学分析来分析 GC 中的 ARG,筛选 GC 患者与胃癌相关的预后biomarker。
文章:

Front Genet (IF: 3.258; Q1). 2021 Feb22;12:615834. doi:10.3389/fgene.2021.615834.
数据来源:
TCGA数据库:375个胃癌病人基因表达数据和临床数据,包括表型数据,生存数据,年龄,性别,肿瘤大小,TNM分期等。去除OS 时间少于30天的样本,最终339个样本用于后续的分析。TCGA当中的数据作为biomarker筛选与模型构建数据集。
GEO数据库作为验证数据集:数据集GSE62254 有300个病人胃癌样本的基因表达数据和临床数据。
自噬相关基因集:
共搜集到531个基因与自噬相关的基因(ARGs ) :其中232个ARGs基因来自 Human Autophagy Database (HADb) ,另外 394 ARGs 来自 GO enrichment-related autophagy genes (GO_AUTOPHAGY) Molecular Signatures Database (MSigDB v6.24)。
预后基因筛选:
1.自噬相关基因单因素cox回归分析,筛选到42个预后显著相关的基因:
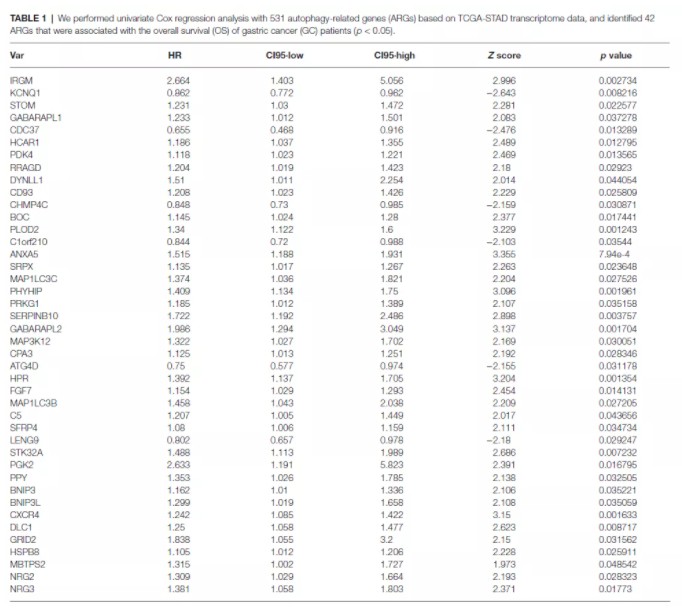
lasso cox 回归分析,进一步筛选预后相关基因,从42个预后基因中筛选到15个基因:
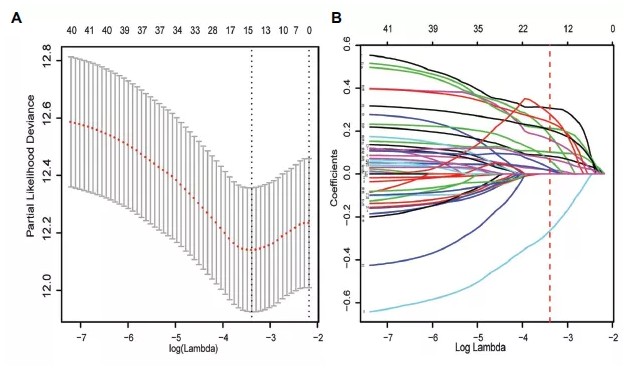
3.多因素cox分析,从15个基因中筛选到6个基因与预后显著相关:
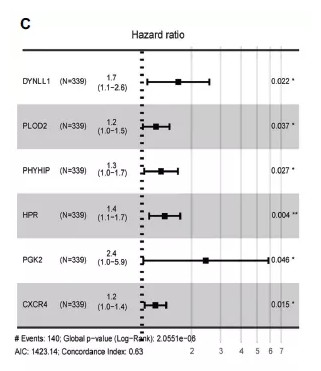
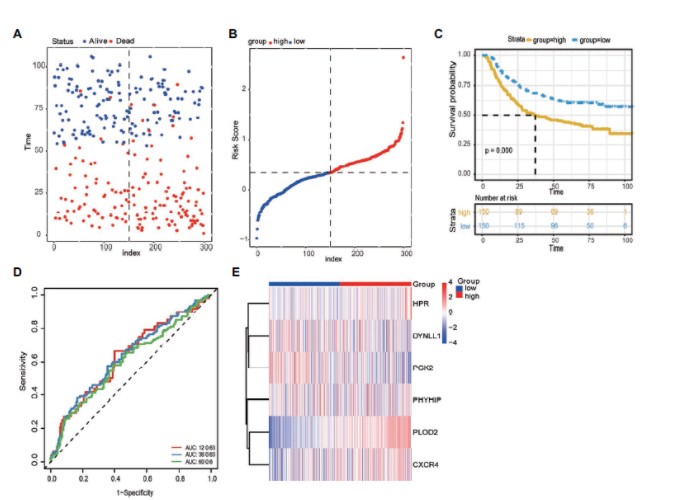
4.利用最终的6个自噬基因构建风险模型,将样本按照风险值分成高低风险组,发现高风险组预后显著差于低风险组,风险预测模型具有很好的预后价值:The risk score for OS = (0.1504 × expression value of DYNLL1) +
(0.1673 × expression value of PLOD2) + (0.0944 × expression value of PHYHIP) + (0.1141 × expression value of HPR) + (0.0727 × expression value of PGK2) + (0.0103 × expression value of CXCR4). ROC曲线分析在 1,3,5年的预测准确性AUC值达到 0.7。
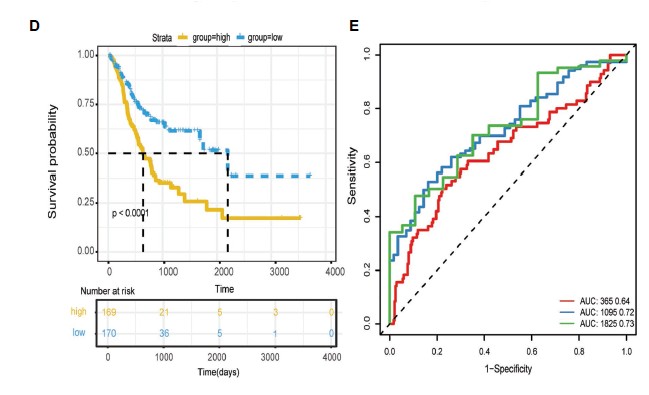
高低风险组比较分析
根据模型计算每一个病人的风险值,按照风险值的中位值划分高低风险组:风险值分布图与对应的病人死亡状态,以及基因表达热图展示。
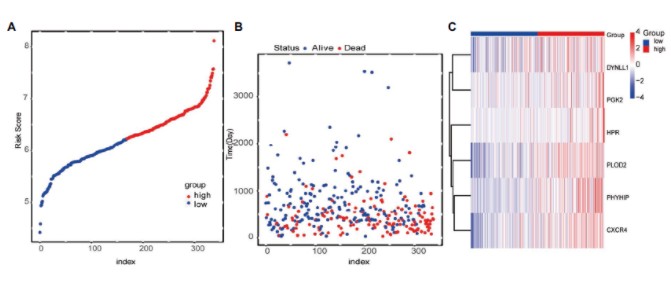
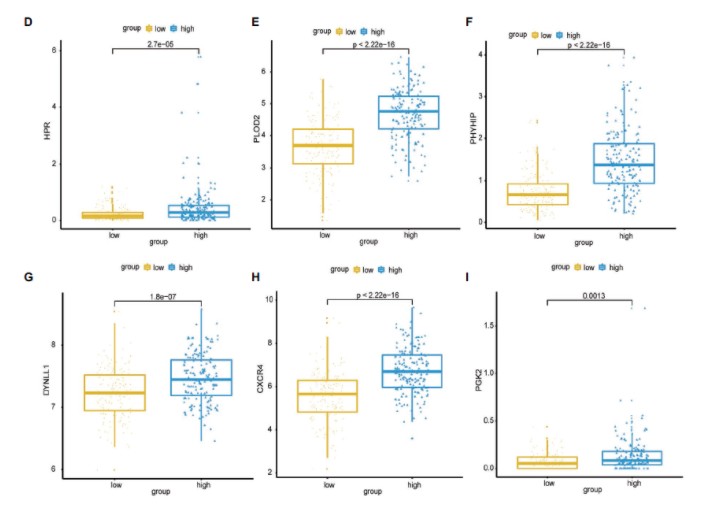
6个signature基因在高低风险组表达差异分析
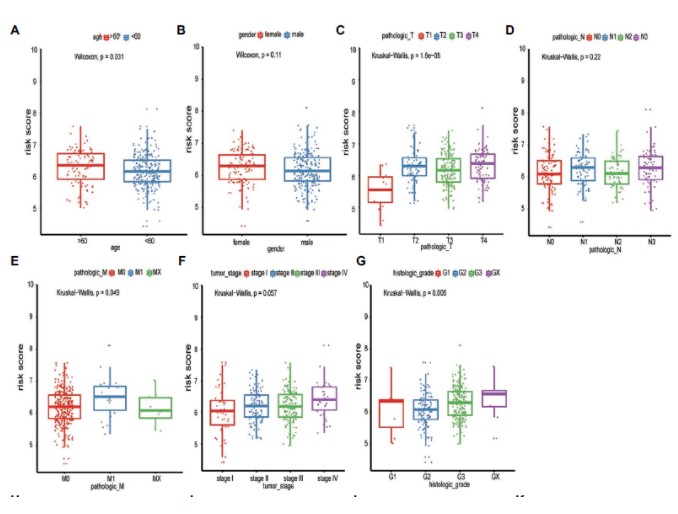
模型预测的风险值与其他临床数据的相关性分析:
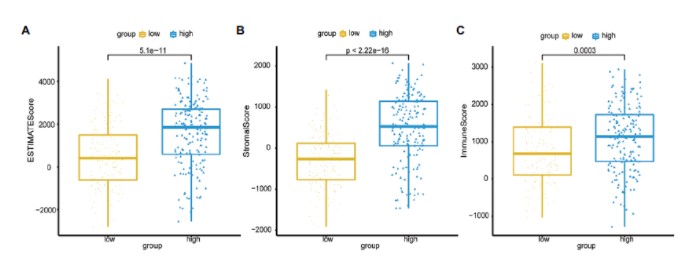
高低风险组,免疫侵润差异分析:ESTIMATE分析基质细胞得分,免疫细胞得分,综合得分。比较发现高低风险组免疫侵润存在显著差异:
模型验证
在GEO数据集中利用模型预测病人的风险值,并划分高低风险组,生存分析发现,高风险组预后显著差于低风险组,说明模型具有普适性具有临床应用价值。
更多数据挖掘文章解析:
- 发表于 2021-06-30 09:51
- 阅读 ( 4782 )
- 分类:TCGA
你可能感兴趣的文章
相关问题
- 请教一下TCGA的蛋白数据怎么理解,它是有正负值的,这个正负值代表什么意思,为什么蛋白表达会有负值?我只想给它分成高表达,低表达和表达缺失组应该怎么处理呀? 0 回答
- TCGA下载 docker报错 0 回答
- TCGA差异分析如何根据临床信息分组? 1 回答
- TCGA用R数据下载错误 1 回答
- 老师,想请教一下TCGA基因表达数据的问题,我从xena.ucsc网页上下载了基因表达数据TCGA-CESC.htseq_counts.tsv;然后发现该数据中只有Ensembl格式的基因ID ,没有SYMBOL格式的。所以接下来进行基因ID格式转换,却发现同一个SYMBOL ID对应的多个Ensembl格式的ID,想问下老师,这种情况该怎么处理?同一个SYMBOL ID所对应的多个Ensembl格式ID的基因表达数据应该留下哪一个? 1 回答
- TCGA的RNA-seq count annotation过滤,有些不知道是否需要删除 0 回答

