群体遗传进化分析用MEGA
MEGA
群体遗传进化分析时,我们往往会对线粒体的一些特定基因进行克隆,然后利用一代测序的方法获得这些克隆的序列信息,那么怎么对这些个体基因序列进行分析,并进行相关的群体遗传进化分析呢?MEGA能满足这一分析的绝大部需求。
MEGA是已经大家都比较了解的一个分子进化遗传分析的软件,所以今天其实是来卖安利的。告诉大家利用它能干群体遗传进化相关的哪些事儿。

第一步:Align
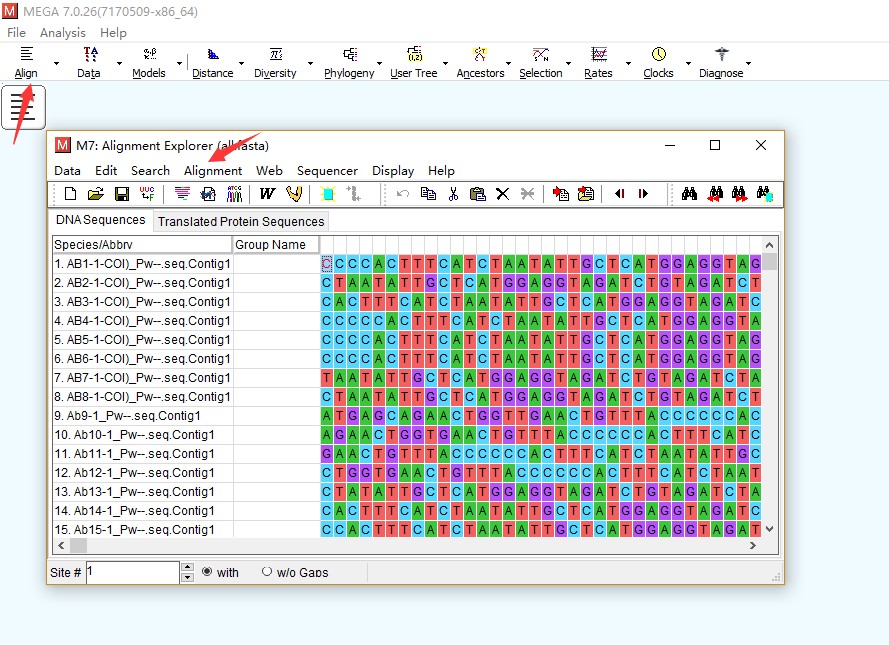 建立一个align是进行多序列比对的第一步,在建立的窗口导入我们通过各种方式获得的多序列的fasta等文件,剪切对齐,又或者直接进行分析。在这里可对基因序列又或者是蛋白质序列进行分析。只有在进行这一步分析之后才可以更好进行后续的分析。这一步分析之后需要将分析的结果导出保存。之后的分析在保存文件的基础上进行即可。
建立一个align是进行多序列比对的第一步,在建立的窗口导入我们通过各种方式获得的多序列的fasta等文件,剪切对齐,又或者直接进行分析。在这里可对基因序列又或者是蛋白质序列进行分析。只有在进行这一步分析之后才可以更好进行后续的分析。这一步分析之后需要将分析的结果导出保存。之后的分析在保存文件的基础上进行即可。
第二步:Stastictics
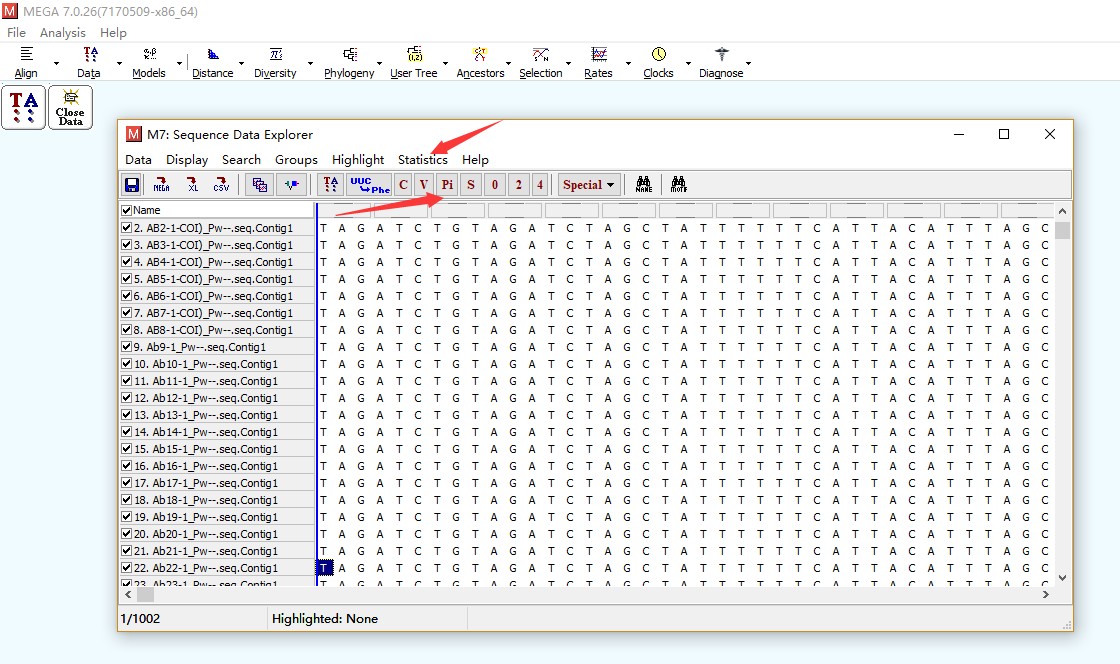
导入上一步分析的结果文件,可以统计一下基本信息,包括基本的变异位点信息,密码子使用信息,转换颠换信息等。后续的一部分计算分析就是基于这些统计的结果进行的。
这些统计结果可以保存成excel或者其他的格式,例如设定保证为excel后统计转换颠换的信息会自动弹出一个excel的表格,保存即可:
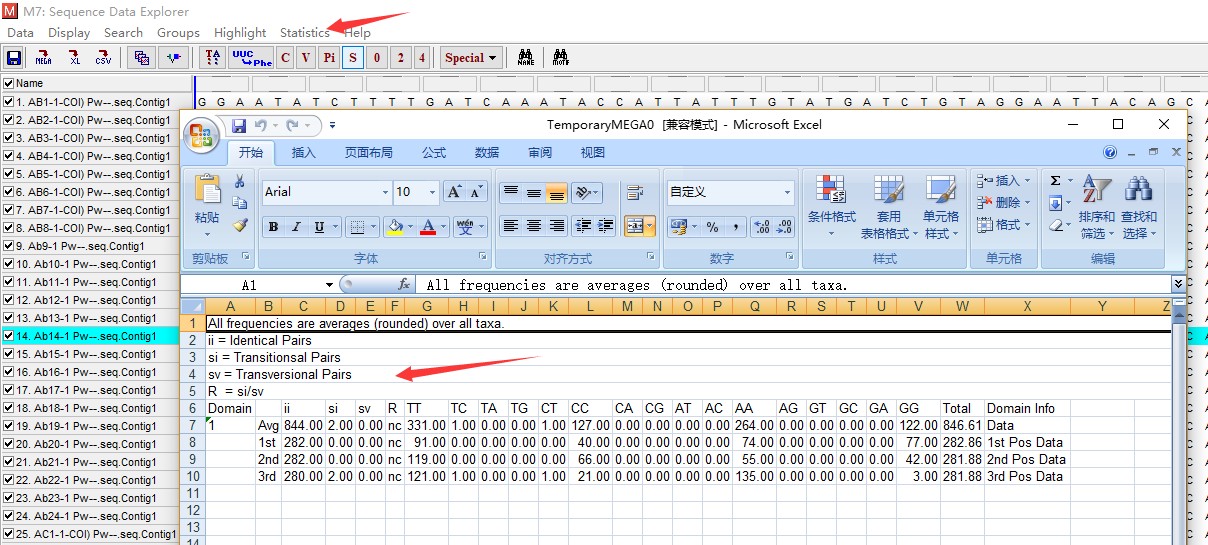
那么这张表格里面能看到的信息就是密码子1st、2nd、3rd上的转换颠换的各种具体信息了,包括转换颠换比和具体的碱基替换类型。类似的其他分析也可获得相应的结果。
第三步:Distance
计算序列的遗传距离也是MEGA的主要功能之一,这里的计算可以是个体之间,也可以是群组(地理种群)之间,故而在利用某个特定序列计算不同的地理种群之间的遗传距离非常有效。
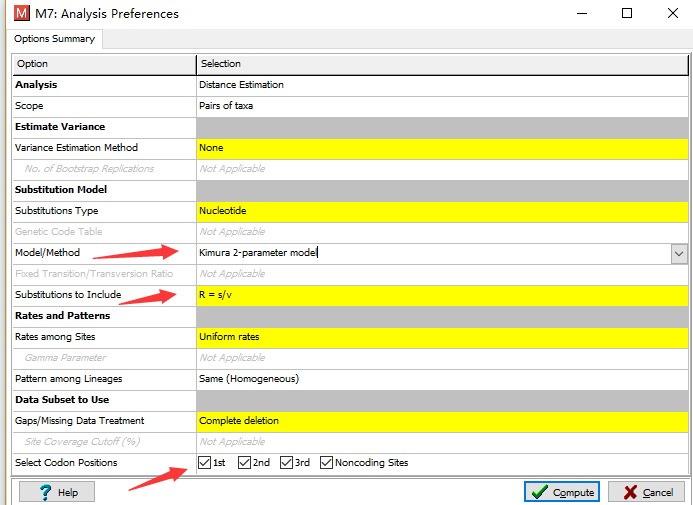
需要注意遗传距离的计算要修改相应的设定,不同的设定结果不同,计算出的结果能保存成文本或excel,方便后续使用。
例如计算群体之间的遗传距离可以获得类似下方的结果(基于K2P,和Ts +Tv):
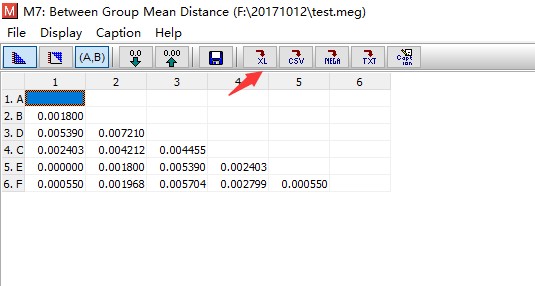
按照图片中指示的位置可以将结果保存为指定格式。
第三步:Phylogeny
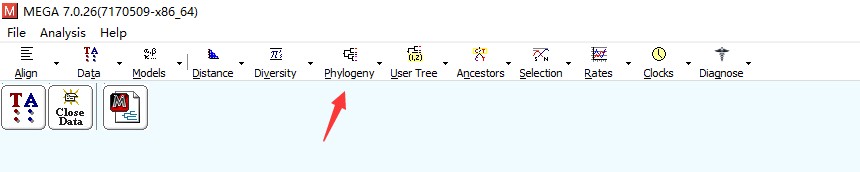
利用这一步分析可以得到序列之间的系统发育树,这里需要选择不同的计算算法,之后要进行distance计算类似的设定,在修改设定之后初步的系统发育树即绘制出来了。
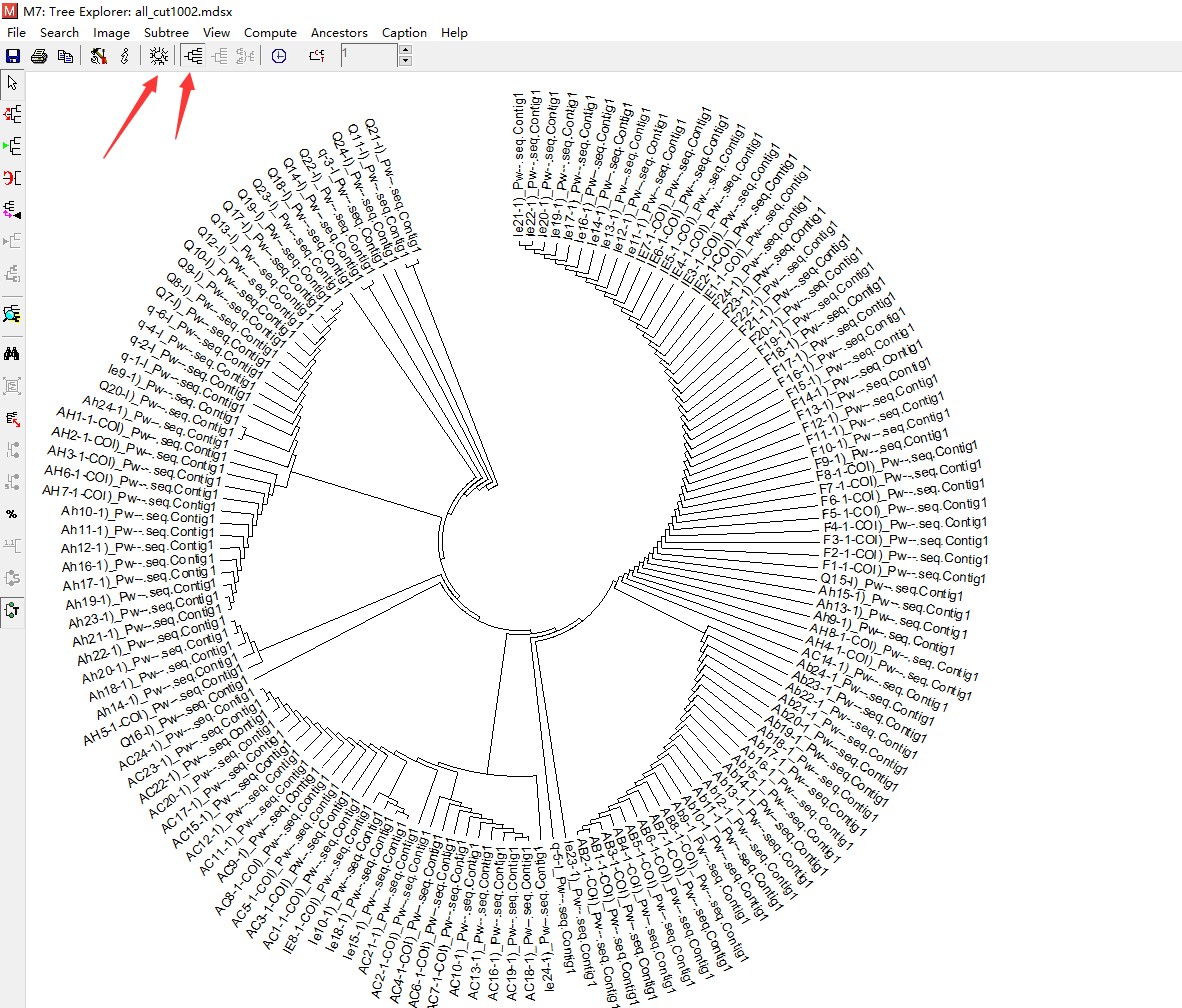
到这一步修改系统树的显示形式就可以了,显示包括了传统类型,放射型还有圆形三大类。选择一个自己比较满意的形式进行保存即可。利用工具栏的image可以将其保存为图片(如果希望获得更漂亮的系统发育树,参考:两分钟做一张让人羡慕的系统进化树图!)。
第四步:other
除以上几个分析之外,MEGA还涉及其他很多种分析,例如核苷酸序列和蛋白质序列相互转换,在线blast等等,相关的使用说明可以在MEGA官方网站获得,目前MEGA版本已经更新至7.0,能满足分子遗传进行化绝大部分分析的需求。
小贴士
给出一个小贴士:出现在MEGA统计分析结果中的“nc”是什么。
“nc”会出现在哪里?给你一个栗子。
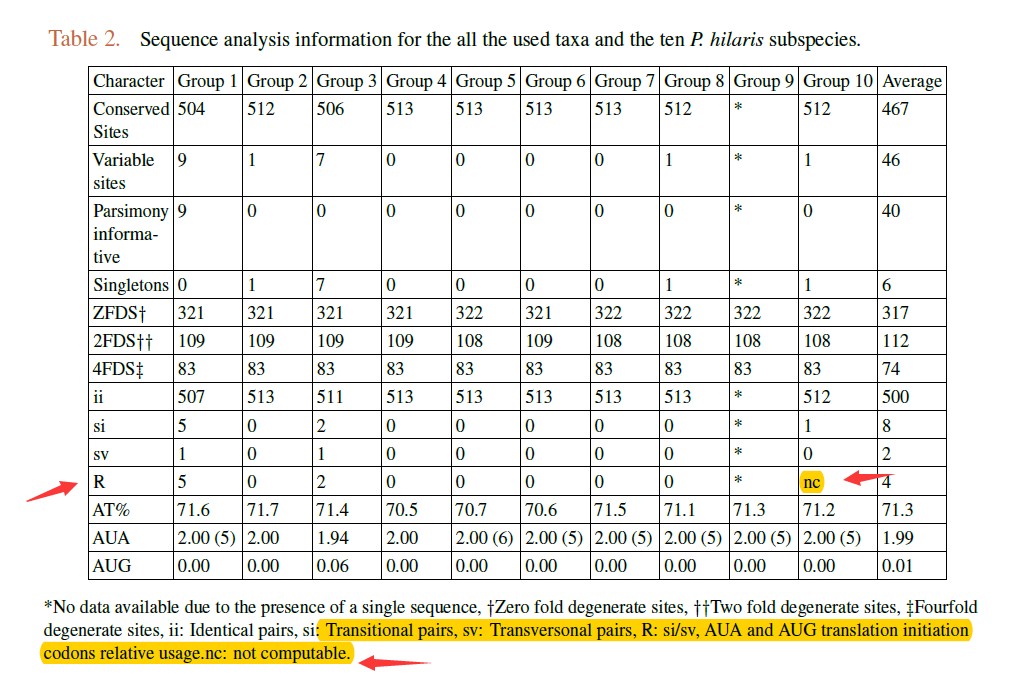
这一表格来源了2014年的一篇文章(Saeb A T M),在计算Ts/Tv(即si/sv),也就是转换比上颠换的时候结果出现了一个“nc”,即表格中红箭头标识的位置。他给出了解释:not computable!
那么就说明了一个问题,MEGA无法计算。why why why……因为R表示转换数比上颠换数,但是颠换没有发生,那么分母…额……(ps: MEGA提到的R有很多种情况,注意差别)
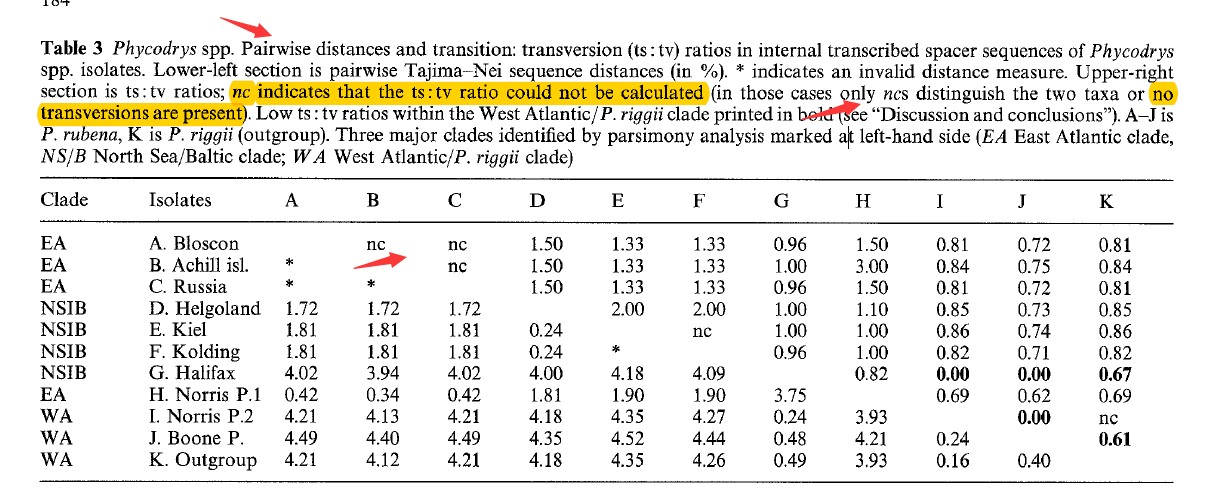
如果我们再基于这样的R值去进行其他的分析,同样会是“nc”,例如下面这张表格的数据就是基于Ts/Tv的结果计算了遗传距离,导致遗传距离的结果也无法计算。(Oppen M J H V 2004)
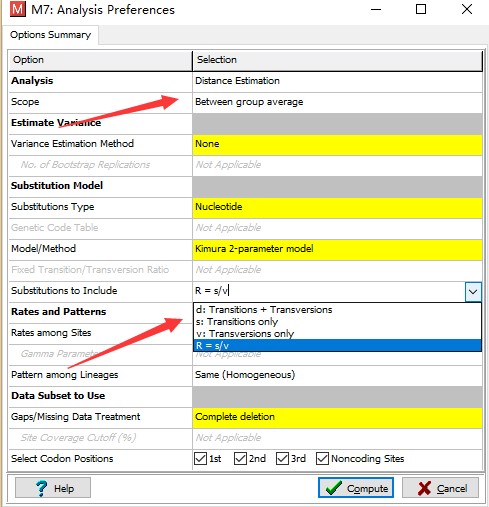
那么我以自己的例子来演示一下,当我的Ts/Tv数据为“nc”,进行一个遗传距离计算,设定Substitution to Include是R=s/v,那么后续的结果还是“nc”。
那么你遇到过类似的情况吗?很明显,这种情况虽然不是常态,但并不是没有。
但是当你使用MEGA进行分析的时候遇见了也不要担心。查看一下你的数据,是不是不符合计算标准?确认之后大胆的写上你的结果就是:NC!
参考文献
Oppen M J H V, Draisma S G A, Olsen J L, et al. Multiple trans-Arctic passages in the red alga Phycodrys rubens: evidence from nuclear rDNA ITS sequences[J]. Marine Biology, 1995, 123(1):179-188.
Saeb A T M, Grewal P S. Phylogenetic and Population Genetic Structure Of the Yellow Spotted Longicorn Beetle Psacothea Hilaris[J]. Advances in Life Sciences & Health, 2015:1-11.
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-04-23 09:39
- 阅读 ( 19056 )
- 分类:软件工具
你可能感兴趣的文章
- MEGA下载 4088 浏览
- 生物信息分析用笔记本推荐-2020年618前夕 10529 浏览
- 如何利用iTOL绘制漂亮的进化树 19118 浏览
- 从MEGA进化树分析结果nwk文件提取基因ID 10419 浏览
- MEGA构建进化树遇到报错信息 40087 浏览
