如何下载TCGA中指定Primary Site的样本数据
如何下载TCGA中指定Primary Site的样本数据
在我们的《TCGA差异表达分析》课程中,我们介绍了采用TCGAbiolinks 去下载GDC上的TCGA数据。但是最近有学员想基于“Primary Site” 筛选一下样本,只对其中的一种类型进行分析。 如下图所示:
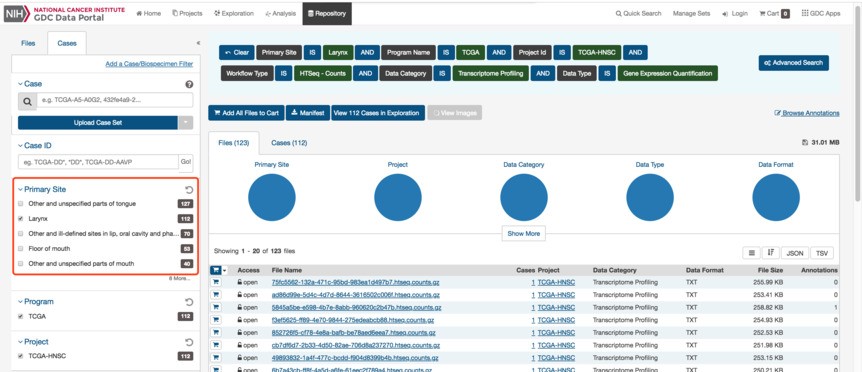
我查看了一下TCGAbiolinks的文档,发现该软件包不支持对Primary Site的筛选。既然GDC官方网站上能显示出Primary Site,那肯定会有一个字段对应这个信息。
我再看了一下GDC的官方API文档,通过调用API,是可以拿到样本的“Primary Site”, 但是采用官方的API比较麻烦。最后找到了Bioconductor中的“GenomicDataCommons ”包,该包是对GDC API 的封装。
简单研究了一下GenomicDataCommons 文档,最后写了一个简陋的代码用于筛选样本:
# 筛选primary_site对应的癌症样本
library(GenomicDataCommons)
resp = cases() %>% filter(~ project.project_id=='TCGA-HNSC' &
primary_site =='Larynx') %>%
GenomicDataCommons::select(c(default_fields(cases()),'samples.sample_type')) %>%
response_all()
resp %>% count()
case_name <- resp$results$submitter_id
# 之后对下载的samplesDown 进行过滤,获得需要下载的sample_download
download_name = substr(samplesDown,1,12)
sample_download <- samplesDown[download_name %in% case_name]
有了sample_download ,就可以采用TCGAbiolinks进行下载了。
把要下载的数据筛选出来之后重新得到query,添加barcode选项指定下载想要的数据:
query <- GDCquery(project = project,
data.category = data_category,
data.type = data_type,
workflow.type = workflow_type,
barcode = sample_download,
# sample.type = sample_type,
legacy = legacy)
如果您想学习TCGA数据挖掘,请学习的我TCGA系列课程:
- 发表于 2019-01-09 15:14
- 阅读 ( 6013 )
- 分类:TCGA
你可能感兴趣的文章
- NCBI-SRA数据下载的3种方法 26137 浏览
- 从TCGA数据库中下载指定类型样本的数据 5619 浏览
- gdc-client 下载TCGA数据 13156 浏览
- GEO数据库之芯片数据下载 13093 浏览
- 从NCBI当中SRA数据库中下载高通量测序数据 22530 浏览
相关问题
- R语言下载GSE数据一直在报错 0 回答
- 老师,下载数据是出现图片这个错误,我该怎么办? 1 回答
- 求助:用Rstudio下载数据,出现remaining后不动了,是不能下载了吗? 2 回答
- SRA数据下载 1 回答
- 高通量下载数据格式 1 回答
- 请问在运行利用exprs()获取表达矩阵 exprSet = exprs(gset)时,报错Error in (function (classes, fdef, mtable) : unable to find an inherited method for function ‘exprs’ for signature ‘"factor"’不知道什么原因? 1 回答
3 条评论
请先 登录 后评论
