快速了解转录调控家族的新贵-circRNAs
做转录组调控研究可用的技术手段逐渐增多,各种数据的联合分析变得越来越重要。转录调控家族在CNS上也不再总是mRNA、LncRNA这些老面孔,近年来circRNAs可谓是风生水起,大家一起来看看这个circRNAs到底是何方神圣吧?说不定可以成为您科研工作的有力武器!
1 环状RNA是什么?
circRNAs(Circular RNAs,环形RNA分子)是一类不具有5' 末端帽子和3' 末端poly(A)尾巴、并以共价键形成环形结构的非编码RNA分子。
2 环状RNA的研究意义?
环状RNA(circRNA)是区别于传统线性RNA的一类新型RNA,具有闭合环状结构,大量存在于真核转录组中。大部分的环状RNA是由外显子序列构成,在不同的物种中具有保守性,同时存在组织及不同发育阶段的表达特异性。由于环状RNA对核酸酶不敏感,所以比线性RNA更为稳定,这使得环状RNA在作为新型临床诊断标记物的开发应用上具有明显优势。
3 环状RNA有哪些类型?
目前发现的circRNA主要来源于基因外显子,但多项研究也表明circRNA的类型比想象的要复杂,可以来自基因组上多种基因结构。
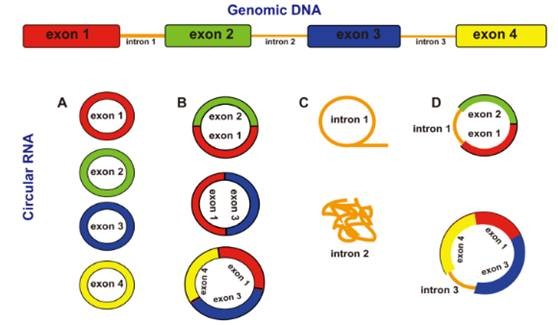
环状RNA的主要类型
1 | annot_exon:环状RNA的剪切点位于基因的一个外显子的起始和另一个外显子的终止(如B) |
2 | one_exon:环状RNA位于基因的某一个外显子内(如A) |
3 | exon_intron:环状RNA的剪切点之间位于基因的一个或多个完整外显子,其序列由剪切点起始点的第一个外显子起始点、剪切点之间的外显子和最后一个外显子的剪切点终止位点的碱基构成(如D) |
4 | intronic:环状RNA位于基因的一个内含子内(如C) |
5 | antisense:环状RNA位于基因的反义链上 |
6 | intergenic:环状RNA位于基因间区 |
4 环状RNA的识别软件?
find_circ
find_circ(如下图),将不能和基因组比对上reads的两端各取20bp作为锚点,再将锚点作为独立的reads往基因组上比对并寻找唯一匹配位点,如果两个锚点的比对位置在线性上方向呈反向,那么就延长锚点的reads,直至找到环状RNA的接合位置(junction),若此时两侧的序列分别为GT/AG剪接信号,则判断为潜在的环状RNA。
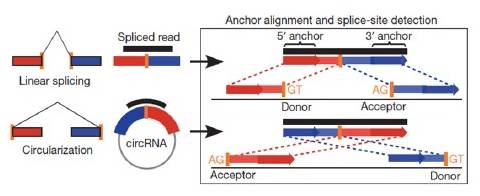
CIRI预测
这款工具是中科院北京生命科学院的赵老师组发表在genome biology(详情查看参考文献部分),是一篇工具类的文章。
它考虑了经典的环状RNA以及一些短外显子成环状RNA的情况,同样以GT-AG剪接信号和外显子边界得到环状RNA。
这个就是今天我们讲的重点,我们先来看一下它的流程图(E):
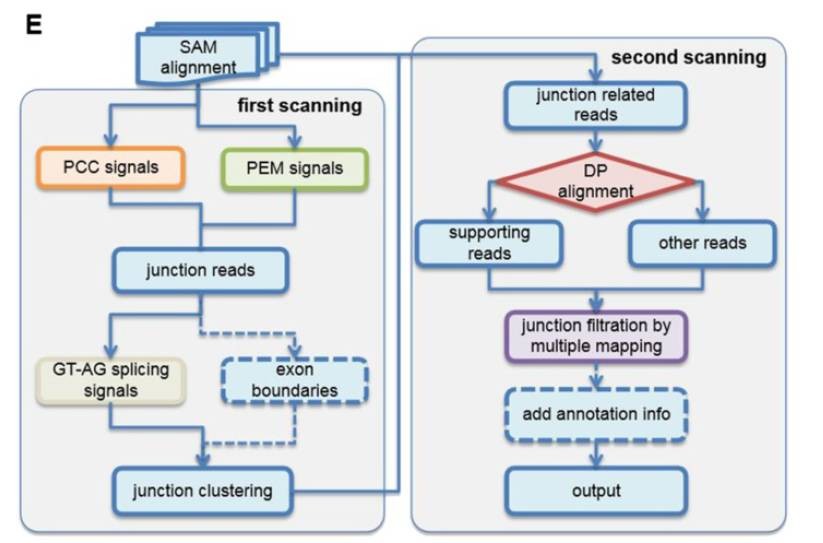
首先把bwa比对产生的所有bam文件合并,经过第一步筛选:还有PCC(paired chiastic clipping)信号和PEM(paired-end mapping)信号的junction reads留下;然后在使用AT-AG原则筛选(GT-AG法则:真核生物基因的内含子5'端大多数是以GT开始,3'端大多数是以AG结束,构成RNA剪接的识别信号);最后形成的junction reads集合进行第二步筛选,使用dynamic programming algorithm确认是否是环状RNA,最后加入注释信息,输出结果。
它的优点在于:1、采用了sam格式中的CIGAR值进行分析,从sam文件中扫描PCC信号。2、对于单外显子成环,或者“长外显子1-短外显子-长外显子2”形成的环形结构,CIRI可以很好的讲这两种情况分开。3、对于paired-end reads,CIRI会考虑一对reads,其中一条可以比对到环状RNA上,另一条也需要比对到环状RNA的区间内。4、对于splicing信号,CIRI也会考虑其他弱splicing信息,算法会从GTF/GFF文件中抽取外显子边界位置,用已知的边界过滤假阳性。
两款软件的比较
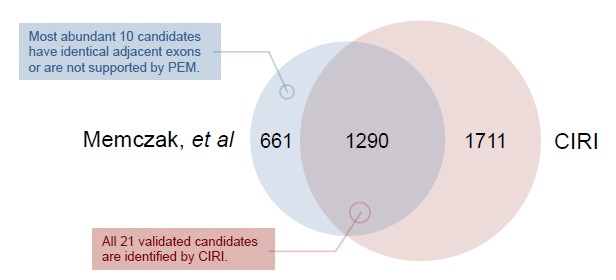
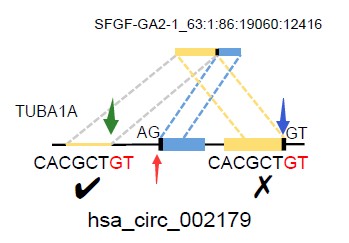
两种方法识别的数量如上图所示,共同识别的有1290个,有661个环状RNA被find_circ识别但是没有被CIRI识别,研究发现其中这661个当中,很大一部分的结果并非环状RNA。例如hsa_circ_002179这种模式(如下图),既有可能是线性RNA但是这个RNA有很大可能是线性reads(如果红色箭头处和绿色箭头处结合是线性RNA,如果红色箭头处和蓝色箭头处结合为环状RNA),这种情况有很高的假阳性,而CIRI把这种情况判断为线性RNA,降低了假阳性。
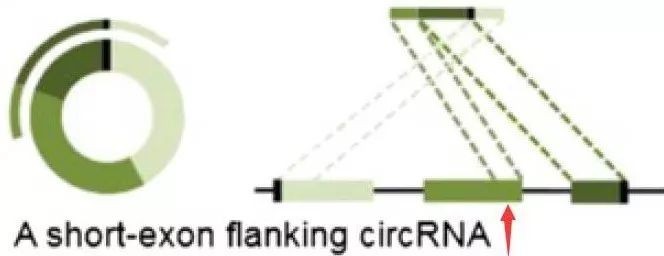
在只有CIRI识别而find_circ没有识别到的1711个环状RNA里边,它们都包含一个短的外显子(红色箭头处),如下图所示,find_circ对于这种环状RNA无法识别,所以CIRI可以更有效的检测识别环状RNA。
所以小编推荐大家在预测环状RNA的时候可以使用这个好用的软件CIRI。
5 参考文献
Yuan Gao, Jinfeng Wang, and Fangqing Zhao.CIRI: an efficient and unbiased algorithm for de novo circular RNA identification.Genome Biol. 2015; 16(1): 4.
Chen G, Cui J, Jin B al.Genome-Wide Identification of Circular RNAs in Arabidopsis thaliana.Front Plant Sci. 2017 Sep 27;8:1678.
- 发表于 2018-04-21 22:43
- 阅读 ( 5351 )
- 分类:转录组
你可能感兴趣的文章
相关问题
- 全转录组到底能不能测到circRNA? 1 回答
