你不知道的微生物互作分析方法
处理16s测序数据时,相信很多同学已经看倦了常见的Alpha、Beta多样性分析,PCA分析之类,那么除了这些标准内容,我们还能做些什么呢?
此前,我们介绍了如何进行网站在线OTU互作网络分析——OTU互作网络分析用MENA,而今天我们又来推荐一个新方法——CoNet。CoNet全称是Co-occurence network inference,也可以称为共现性网络分析,可用于探究微生物之间的互作关系,例如探究土壤微生物之间的交流模式[1],再比如探究人体微生物的共现与互斥关系[2]。目前针对这一分析方法开发出了Cytoscaep相应的分析插件CoNet,利用这个插件可以直接进行分析,操作简单上手快!
数据准备
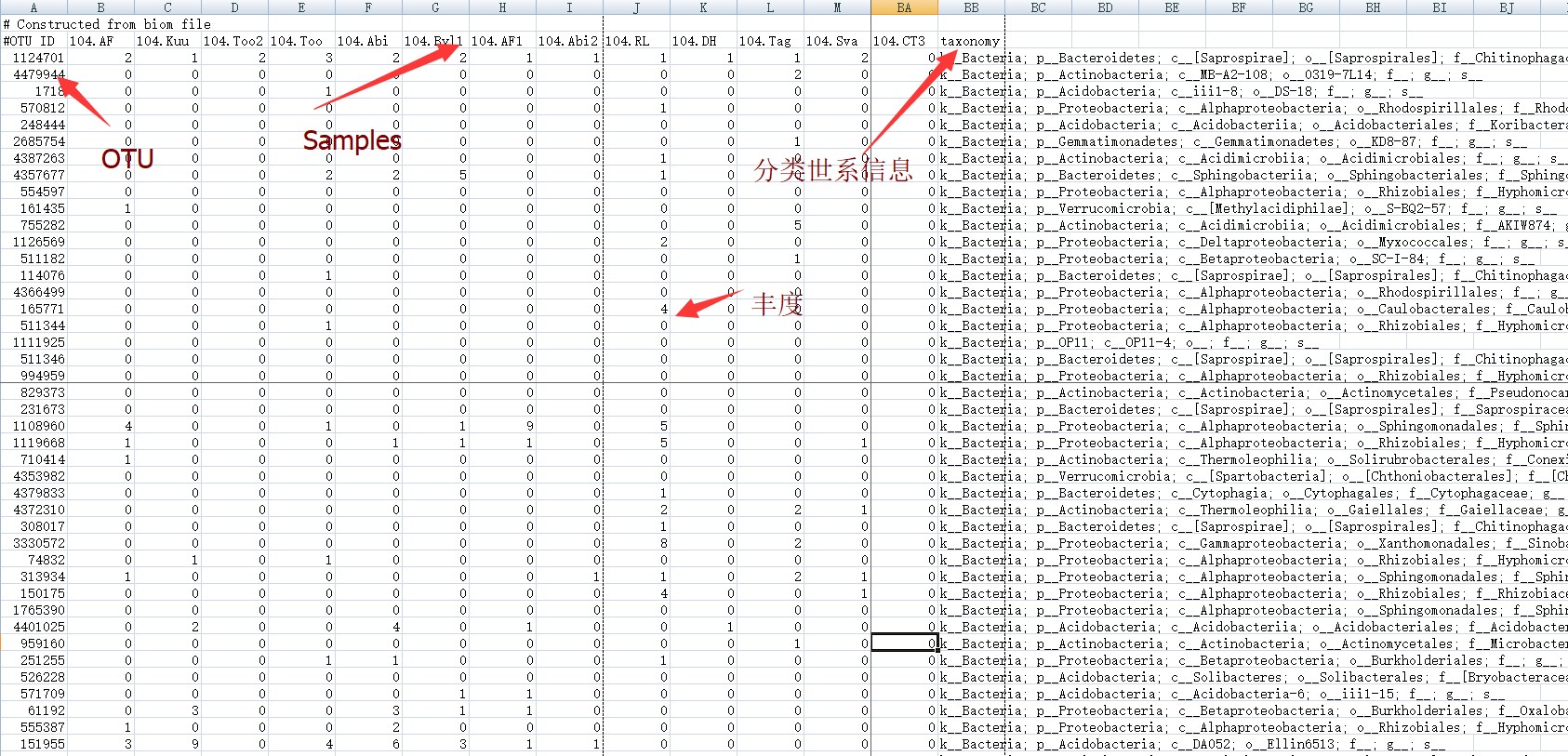
CoNet分析应用的文件可以是BIOM文件,也可以是由它转换而来的OTU丰度矩阵文件。格式上的要求(如下图):第一列是OTU名称ID, 之后的每一列的列名是样品名称,每一行数据表示OTU在每个样品中的丰度。而最后一列是OTU的分类世系信息,涉及门纲目科属种等。OTU数据也可以只是具体到属水平的分类。
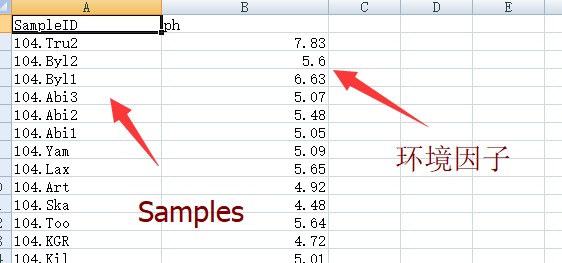
也可以准备环境因子数据进行联合分析,如下:
插件安装
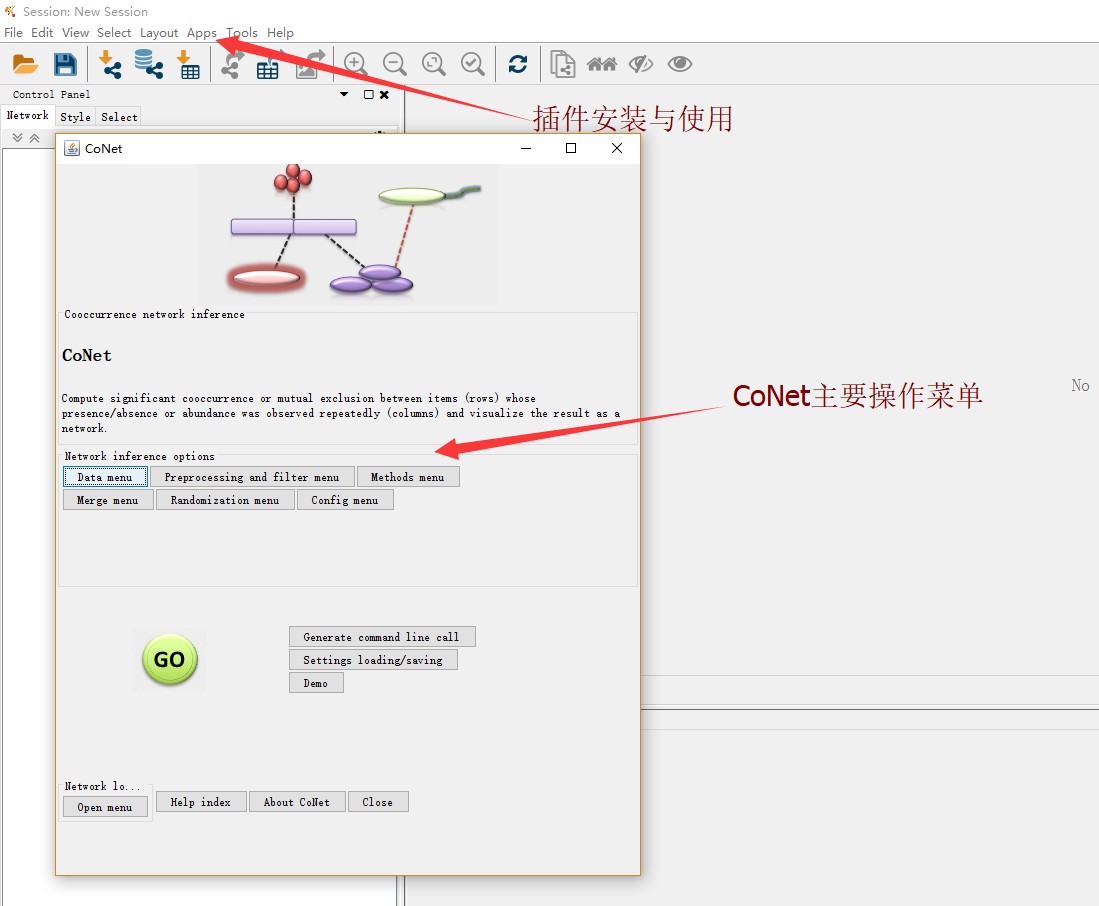
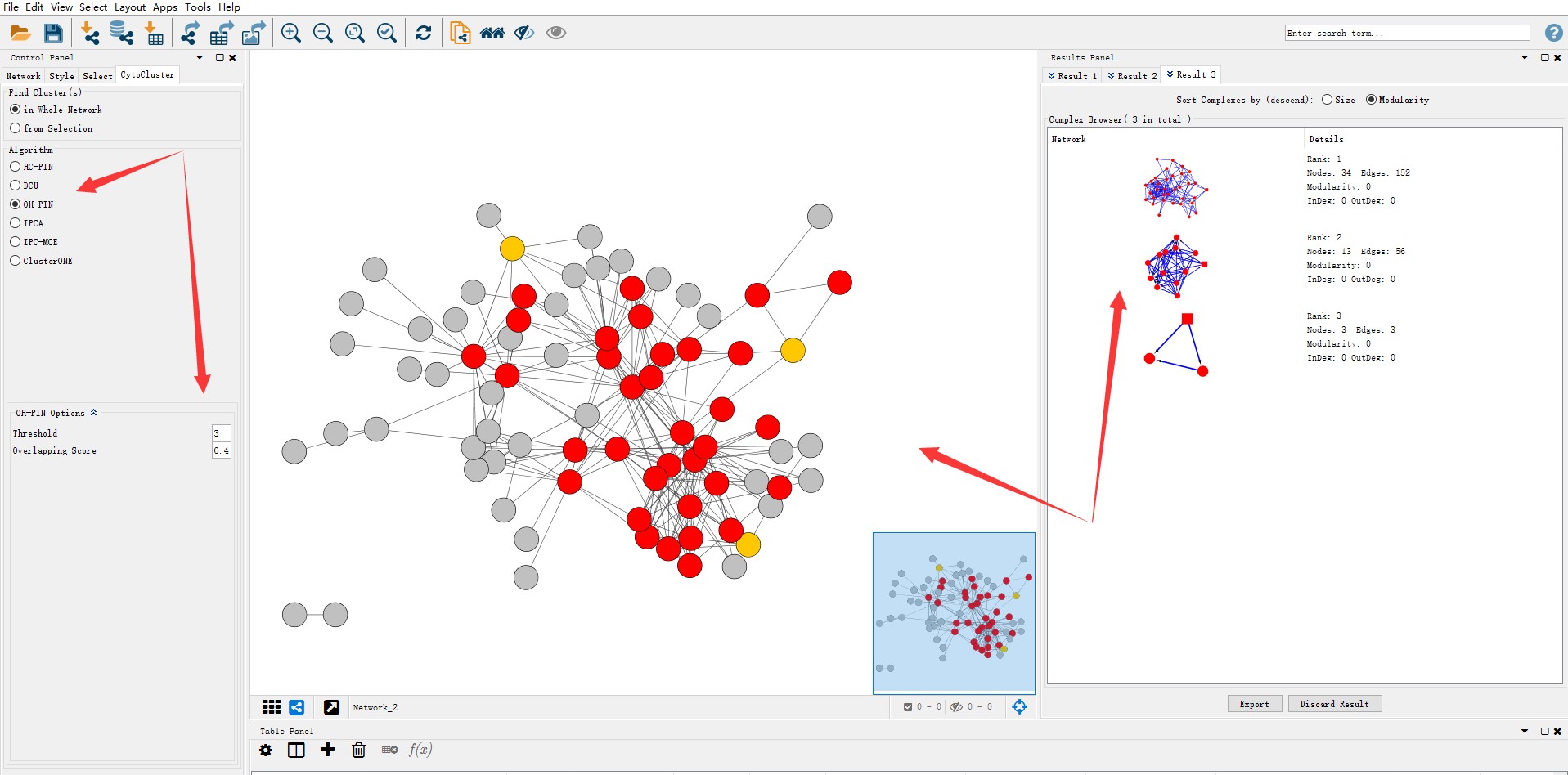
Cyroscape的安装可以参考:Cytoscape-网络图绘图技巧。而进行CoNet之前需要在Cytoscape上安装CoNet插件,同时需要安装CytocCluster,方便对生成的网络进行聚类分析,从而划分模块。而开始分析的页面也就是下图显示的页面。
导入数据与设置
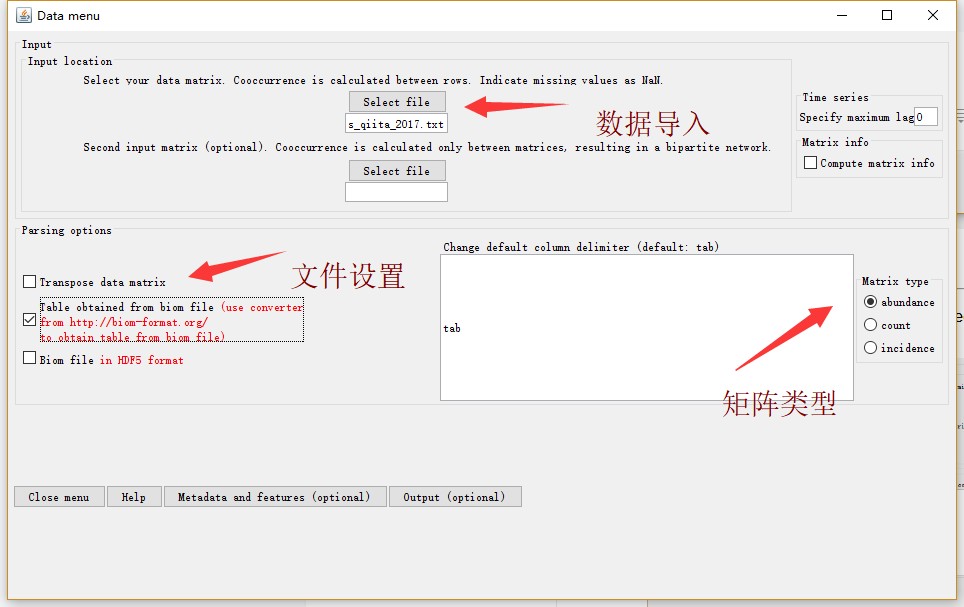
点击Data menu。在弹出的窗口选项——Select file中导入丰度矩阵,并进行相关设置(如下图)。
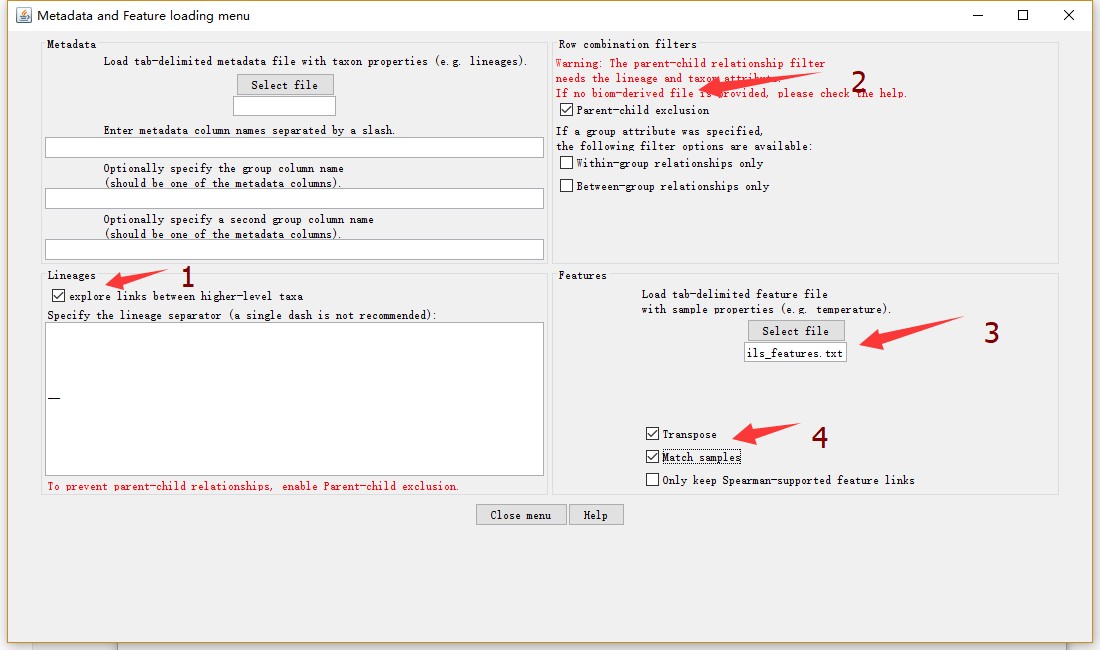
完成初步数据设置之后,选择该窗口的Metadata and features(optional),开始其他设置,并导入环境因子数据,包括勾选explore links between higher-level taxa,Parent-child exclusion,以及对环境因子数据进行设置(如下图)。
数据过滤
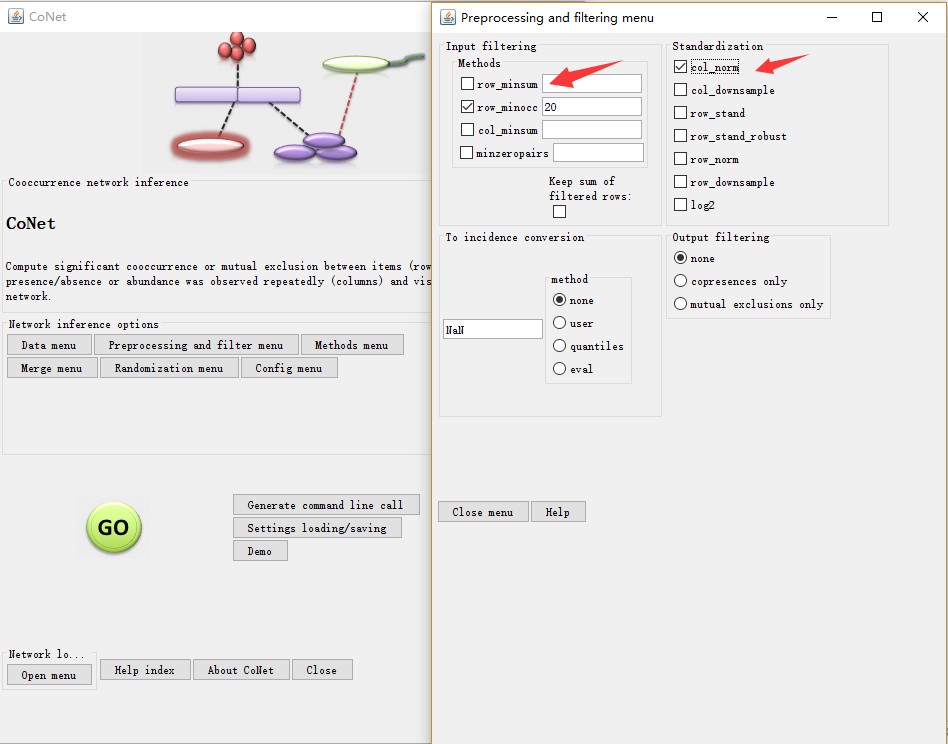
完成上一步之后,回到主菜单,选择Preprocessing and filter menu,开始对数据进行过滤,同时进行数据的标准化。
计算方法
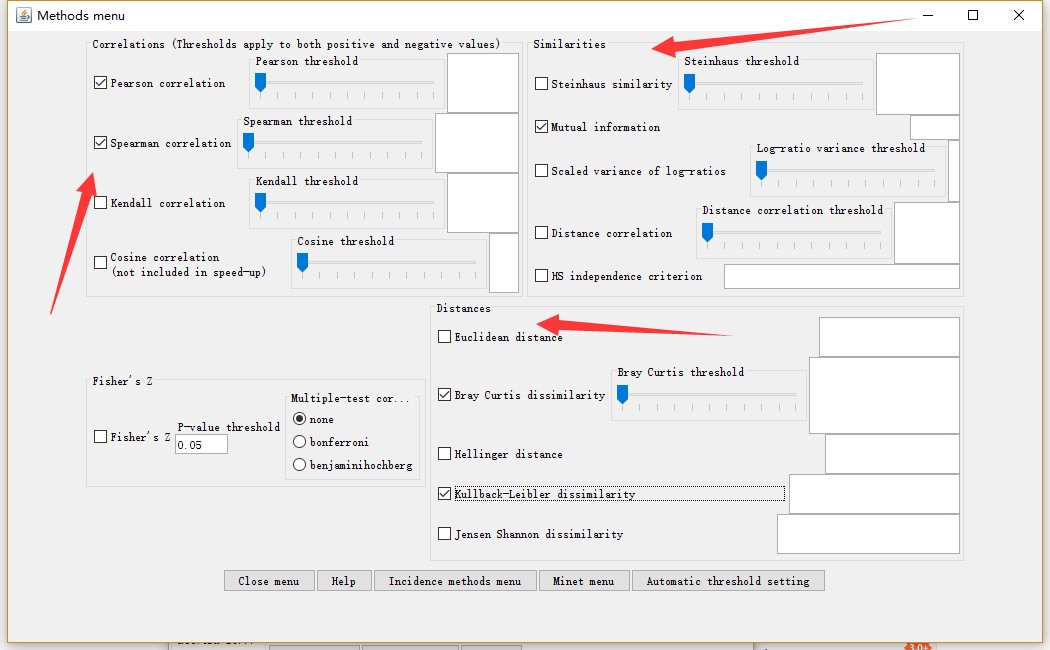
在Method menu进行相关性、相似性以及距离计算方法的选择。例如下图的组合方式:Pearson、Spearman、Mutual information、BC以及KLD。
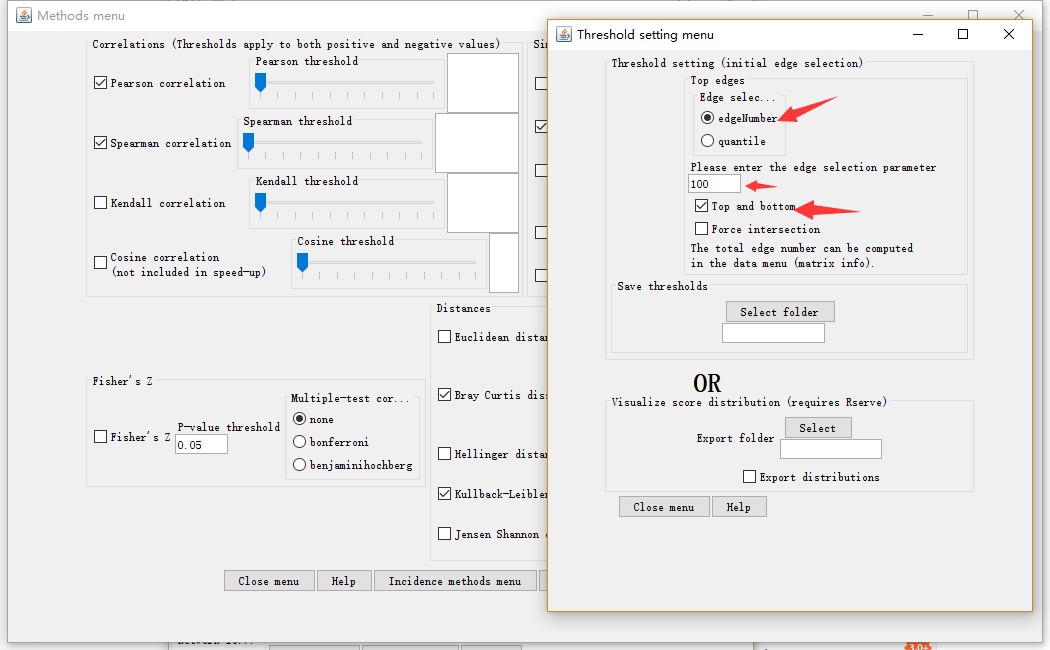
通过该窗口选择Automatic threshold setting,设置网络阈值,选择edgeNumber 和Top and bottom,并填写上对应的数值100,可以按照实际填写。
ReBoot
进入Randomization menu,先进行Permutaion,设置Routine 、Resampling,重新标准化Renormalize,以及进行空分布文件的的保存等等(如下图)。设置完成之后,进入主菜单点击GO。
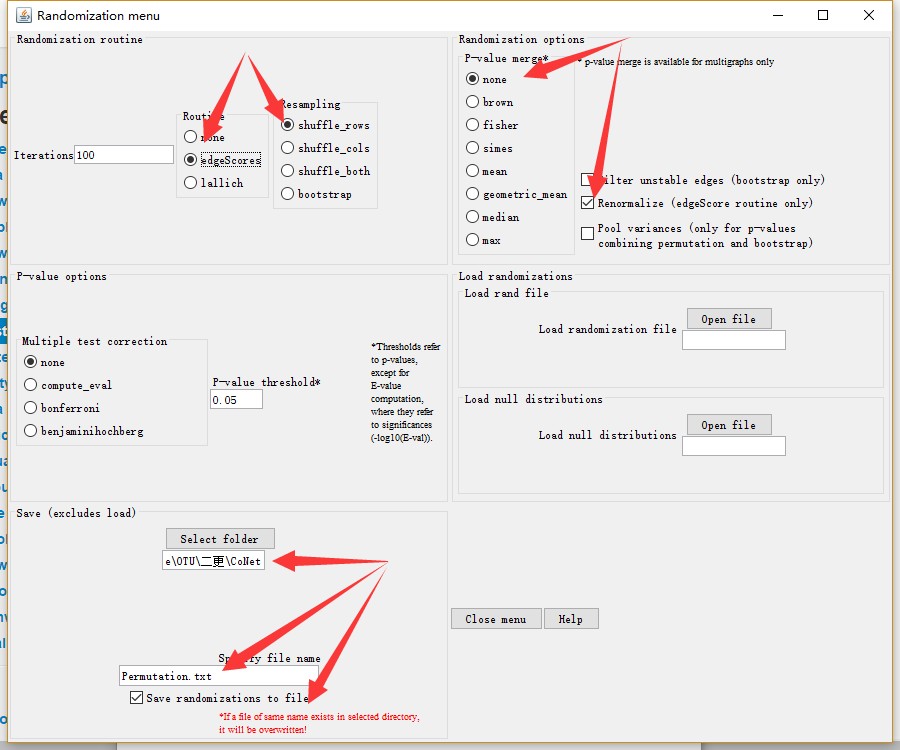
完成Permutation,开始进行Bootstrap,在相同窗口中进行设置,修改Resampling,取消重新标准化Renormalize,设置P-value merge方法为Brown,多重检验benjaminihochberg,同样保存随机化文件,并导入刚刚计算的空分布文件(如下图),进入主菜单点击GO。
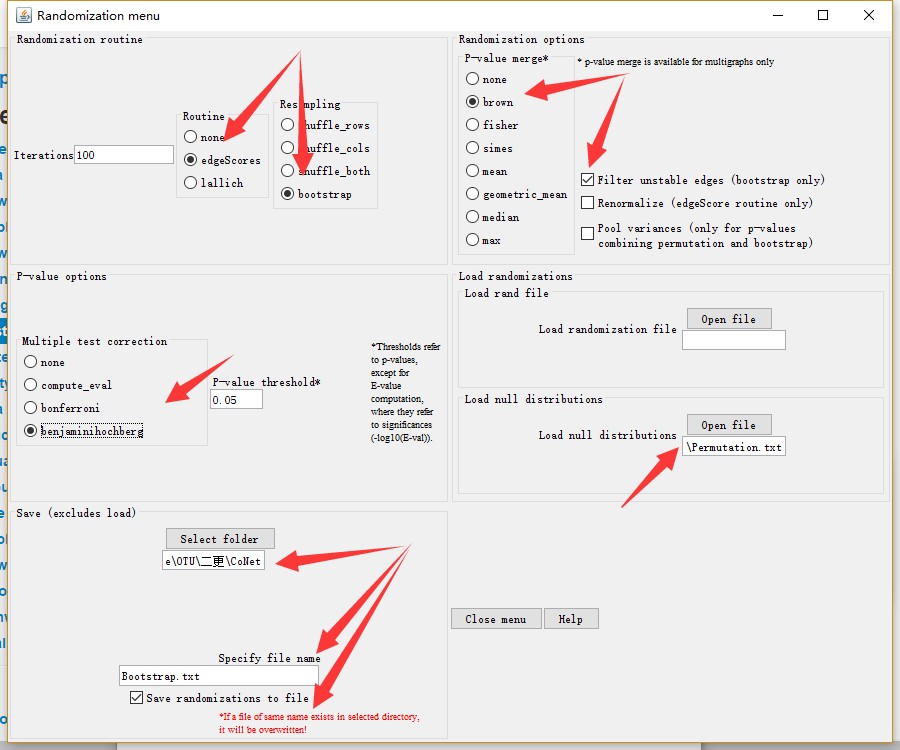
恢复网络(可选)
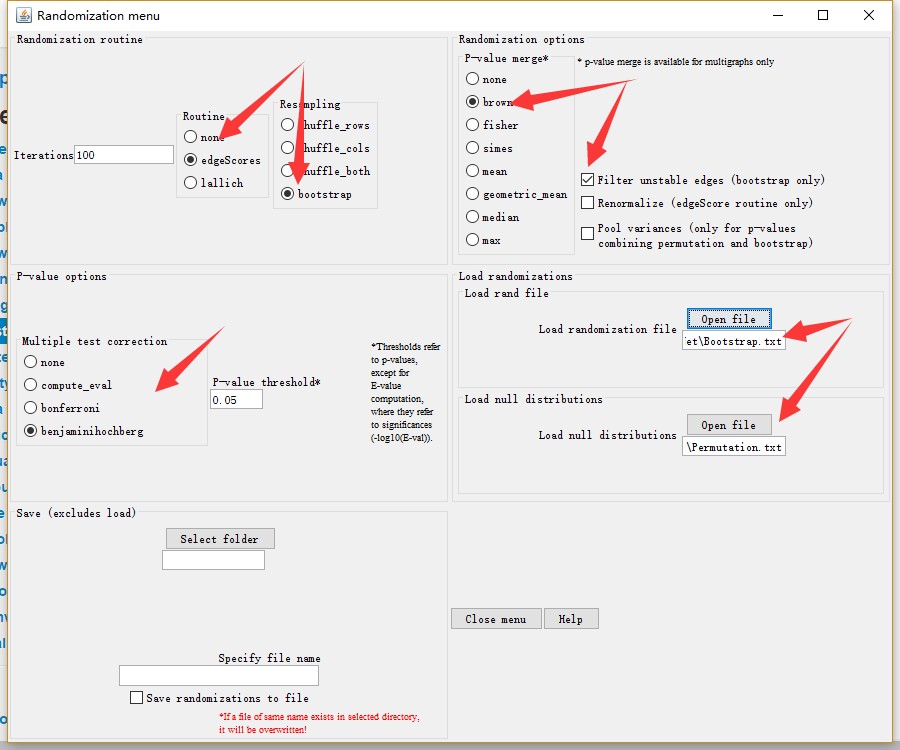
基于前一步生成的文件恢复CoNet网络,在前一步的设置上基础上,取消文件保存,并导入生成的随机文件(如下图),之后点击主菜单GO。
网络图基本设置与聚类分析
参考Cytoscape-网络图绘图技巧进行网络图点线设置,可以获得如下结果。
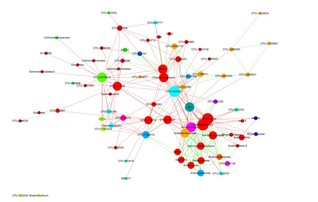
此外,结合CytoCluster对网络进行聚类分析,对网络进行模块划分,部分结果如下:
愿大家get多多技能 ,学习快乐!
参考文献:
[1] Durrer A, Gumiere T, Taketani R G, et al. The drivers underlying biogeographical patterns of bacterial communities in soils under sugarcane cultivation[J]. Applied Soil Ecology, 2016, 110.
[2] Faust K, Sathirapongsasuti J F, Izard J, et al. Microbial Co-occurrence Relationships in the Human Microbiome[J]. Plos Computational Biology, 2012, 8(7):e1002606.
相关推荐:微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制
课程推荐:微生物扩增子分析课程实操 微生物16S/ITS/18S分析原理及结果解读
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-04-20 19:07
- 阅读 ( 21793 )
- 分类:宏基因组
你可能感兴趣的文章
- cytoscape软件的安装与使用_v3.10.2 8802 浏览
- 微生物与环境因子,代谢物,功能等相关性分析linkET 3269 浏览
- 微生物多样性扩增子(16S/18S/ITS)课程更新v2.0 2718 浏览
- PICRUSt2扩增子功能预测与差异比较分析 6317 浏览
- 微生物多样性分类柱状图按照分组进行排序脚本升级;tax_bar_plot.r 2702 浏览
- cytoscape-cytoHubba插件的安装与使用 25797 浏览
