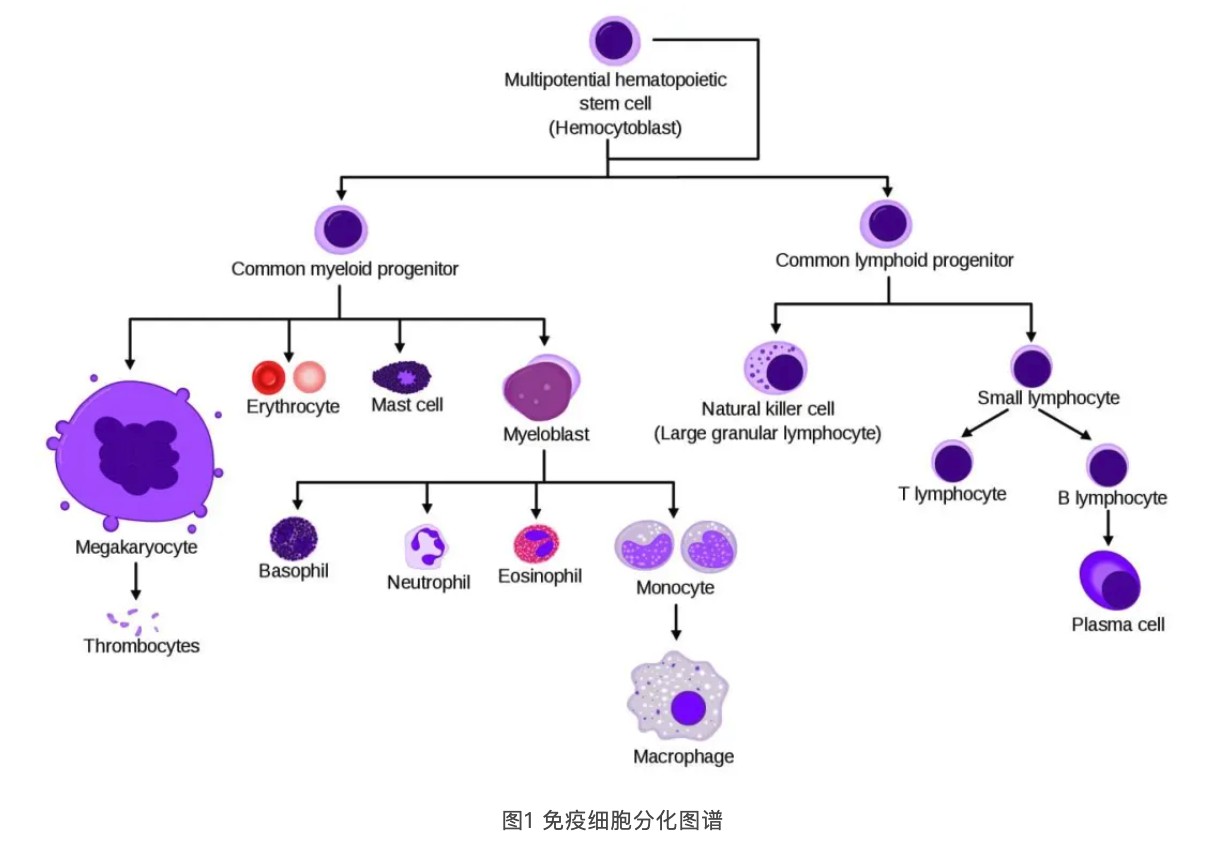
人类免疫细胞marker基因
理解各种免疫细胞的详细分类以及与之相关的Marker Gene 是一个复杂且持续发展的领域。以下是一些常见的免疫细胞亚型及其相关的Marker Gene 示例。请注意,这个列表只涵盖了一些基本的免疫细胞类型,而不是详尽的分类。细胞类型和Marker Gene 可能会因研究背景和分析流程而有所不同。
免疫细胞亚型 | Marker Gene 示例 |
CD4+ T Helper 细胞 | CD4、IL2RA、FOXP3(调节性T细胞,Treg) |
CD8+ 细胞毒性 T细胞 | CD8A、CD8B、GZMB(GZMH,Perforin) |
B细胞 | CD19、CD20、CD79A、MS4A1(CD20) |
自然杀伤细胞 | CD56(NCAM1)、CD16(FCGR3A)、NCR1(NKp46) |
单核细胞 | CD14、CD16(FCGR3A)、CD163 |
浆细胞 | CD38、CD138(SDC1)、XBP1 |
树突状细胞 | CD11c(ITGAX)、CD11b(ITGAM)、CD1c(BDCA1) |
巨噬细胞 | CD68、CD163、CD206(MRC1) |
嗜碱性粒细胞 | CCR3、IL5RA |
Th1 细胞 | T-bet(TBX21)、IFNG(IFN-γ)、CXCR3 |
Th2 细胞 | GATA3、IL4、IL13、CCR4 |
Th17 细胞 | RORC(RORγt)、IL17A、IL17F、CCR6 |
调节性 T细胞(Treg) | FOXP3、CD25(IL2RA)、CTLA4 |
感染性单核细胞(Monocyte-Derived Dendritic Cell) | CD1c(BDCA1)、CD14、CD11c(ITGAX) |
单核细胞(Monocytes)可以进一步分为不同的亚型,主要分为三个主要亚型:经典单核细胞(Classical Monocytes,也称为CD14++CD16-)、非经典单核细胞(Non-Classical Monocytes,也称为CD14+CD16++)、中间单核细胞(Intermediate Monocytes,也称为CD14++CD16+)。每个亚型在免疫功能和表观特征上都有所不同,因此它们具有不同的Marker Gene。
以下是每个单核细胞亚型及其相关Marker Gene 的示例:
单核细胞亚型 | Marker Gene 示例 |
经典单核细胞 | CD14、CD64(FCGR1A)、CCR2、LYZ(溶酶体酶) |
非经典单核细胞 | CD16(FCGR3A)、CD163、CD33、CX3CR1(Fraktalkine受体) |
中间单核细胞 | CD14、CD16(FCGR3A)、CD163、CD11c(ITGAX) |
CD4+ T Helper 细胞(CD4+ T Helper Cells)是一类免疫细胞,它们可以进一步分为不同的亚型,每个亚型在免疫功能和细胞特性上都有所不同。以下是一些常见的 CD4+ T Helper 细胞亚型及其相关的 Marker Gene 示例:
CD4+ T Helper 细胞亚型 | Marker Gene 示例 |
Th1 细胞 | T-bet(TBX21)、IFNG(IFN-γ)、CXCR3 |
Th2 细胞 | GATA3、IL4、IL5、IL13、CCR4 |
Th17 细胞 | RORC(RORγt)、IL17A、IL17F、CCR6 |
调节性 T细胞(Treg) | FOXP3、CD25(IL2RA)、CTLA4 |
Th9 细胞 | PU.1(SPI1)、IL9、IL10 |
Th22 细胞 | AHR、IL22、CCR10 |
Th25 细胞 | IL25、IL17RB |
CD8+ 细胞毒性 T细胞(Cytotoxic T Cells)可以根据其功能和表达的分子特征进一步分为几个亚型。以下是一些主要的CD8+ 细胞毒性 T细胞亚型及其相关的Marker Gene 的示例:
CD8+ T细胞毒性 T细胞亚型 | Marker Gene 示例 |
原始幼稚(Naïve)CD8+ T细胞 | CD8A、CD45RA(PTPRC)、CCR7、LEF1 |
中间记忆(Central Memory,TCM)CD8+ T细胞 | CD8A、CD45RO(PTPRC)、CCR7、IL7R |
效应记忆(Effector Memory,TEM)CD8+ T细胞 | CD8A、CD45RO(PTPRC)、CCR7、CX3CR1、CD57 |
末端效应(Terminal Effector,TE)CD8+ T细胞 | CD8A、CD45RO(PTPRC)、GZMB(Granzyme B)、PRF1(Perforin)、KLRG1 |
B细胞可以进一步分为不同的亚型,这些亚型在免疫功能和表观特征上有所不同。以下是一些常见的B细胞亚型及其相关的Marker Gene 的示例:
B细胞亚型 | Marker Gene 示例 |
B-1a 细胞(B-1a Cells) | CD5、CD11b(ITGAM)、CD43(SPN) |
B-1b 细胞(B-1b Cells) | CD5(较低表达)、CD11b、CD20 |
B-2 细胞(B-2 Cells) | CD20、CD19、CD22、CD79A、CD79B |
调节性 B 细胞 | CD24、CD38、CD1d、IL10、CD5(一些亚型) |
自然杀伤细胞(Natural Killer Cells,NK细胞)是一类免疫细胞,通常可进一步分为两个主要亚型:经典自然杀伤细胞(Classical NK Cells)和非经典自然杀伤细胞(Non-Classical NK Cells)。每个亚型在免疫功能和表观特征上都有所不同,因此它们具有不同的Marker Gene。
以下是每个自然杀伤细胞亚型及其相关Marker Gene 的示例:
自然杀伤细胞亚型 | Marker Gene 示例 |
经典自然杀伤细胞 | CD56(NCAM1)、CD16(FCGR3A)、NCR1(NKp46) |
非经典自然杀伤细胞 | CD56(NCAM1)、CD16(FCGR3A)、NKG2D(KLRK1) |
浆细胞(Plasma Cells)也可以进一步分为不同的亚型,其中两个主要亚型是成熟浆细胞(Mature Plasma Cells)和浆细胞前体(Plasma Cell Precursors)。这两种亚型在免疫功能和表观特征上有所不同,因此它们具有不同的Marker Gene。
以下是成熟浆细胞和浆细胞前体的一些Marker Gene 示例:
浆细胞亚型 | Marker Gene 示例 |
成熟浆细胞 | CD38、CD138(SDC1)、XBP1、PRDM1(Blimp-1) |
浆细胞前体 | CD19、CD20、CD27、CD319(SLAMF7) |
树突状细胞(Dendritic Cells,DCs)是一类免疫细胞,具有高度专业化的抗原提呈功能。它们可以进一步分为不同的亚型,包括经典树突状细胞(Classical Dendritic Cells,cDCs)和非经典树突状细胞(Non-Classical Dendritic Cells,ncDCs)。每个亚型在免疫功能和表观特征上都有所不同,因此它们具有不同的Marker Gene。
以下是每个树突状细胞亚型及其相关Marker Gene 的示例:
树突状细胞亚型 | Marker Gene 示例 |
经典树突状细胞(cDCs) | CD11c(ITGAX)、CD11b(ITGAM)、CD1c(BDCA1) |
非经典树突状细胞(ncDCs) | CD123(IL3RA)、BDCA2(CD303)、SLAN(MDC8) |
巨噬细胞(Macrophages)是一类免疫细胞,它们在免疫系统中具有多种功能,可以根据其活性状态和功能分为不同的亚型。以下是一些巨噬细胞亚型及其相关的Marker Gene 示例。请注意,巨噬细胞的分类和Marker Gene 可能会因研究和分析方法的不同而有所不同。
巨噬细胞亚型 | Marker Gene 示例 |
经典激活型巨噬细胞(M1型) | iNOS(NOS2)、IL12B、CXCL9、CXCL10、STAT1 |
替代激活型巨噬细胞(M2型) | CD206(MRC1)、CD163、IL10、Arg1(ARG1)、CD209(DC-SIGN) |
胶质细胞(Microglia) | TMEM119、P2RY12、CX3CR1(Fraktalkine受体)、TREM2 |
巨噬细胞的其他亚型 | 还有其他不同状态和功能的巨噬细胞亚型,Marker Gene 可能会有所不同。 |
Th1(T-helper 1)细胞是一类免疫细胞,通常与细胞介导的免疫应答和抗细胞感染有关。Th1 细胞通常被分为不同的亚型,这些亚型在功能和表观特征上可能略有不同。以下是一些Th1 细胞的亚型及其相关Marker Gene 示例:
Th1 细胞亚型 | Marker Gene 示例 |
经典 Th1 细胞 | T-bet(TBX21)、IFNG(IFN-γ) |
植入 Th1 细胞 | CXCR3、CCR5、CD103(ITGAE) |
非经典 Th1 细胞 | Blimp-1(PRDM1)、CXCL10(IP-10) |
Th2细胞(T Helper 2 细胞)通常用于描述一类免疫细胞,其主要功能包括产生和释放抗炎细胞因子,调节免疫应答,并在过敏反应和免疫调节中发挥重要作用。尽管Th2细胞有一些通用的Marker Gene,但它们也可以进一步细分成不同的亚型,这些亚型在免疫功能和表观特征上可能有所不同。以下是一些常见的Th2细胞亚型及其相关的Marker Gene 示例:
Th2细胞亚型 | Marker Gene 示例 |
经典Th2细胞 | GATA3、IL4、IL5、IL13、CCR4(C-C趋化因子受体4) |
Treg相关Th2细胞 | FOXP3、IL10、TGF-β、CD25(IL2RA)、CTLA4(CD152) |
Mucosal Th2 细胞 | CRTH2(GPR44)、IL17RB、IL25R、IL33R、CD161(KLRB1) |
非经典Th2细胞 | IL9、IL13RA1、CCR8、IL18R1、IL21R |
Th17细胞是一类重要的T细胞亚型,它们在免疫系统中发挥关键作用,但在不同的研究中,Th17细胞也可以进一步分为不同的亚型。以下是一些Th17细胞的可能亚型及其相关的Marker Gene 示例:
Th17细胞亚型 | Marker Gene 示例 |
经典Th17细胞 | RORC(RORγt)、IL17A、IL17F、IL23R、CCR6 |
趋化性Th17细胞(Th17.1细胞) | IL17A、CCR6、CCR4、CXCR3、IFN-γ、T-bet |
产生IL-10的Th17细胞(Th17.10细胞) | IL17A、IL10、CCR6、CD161、IL23R、CCR4 |
稳定型Th17细胞 | RORC(RORγt)、IL17A、IL17F、CCR6、IL23R |
调节性T细胞(Treg)是一类免疫细胞,它们在调节免疫应答和维护免疫平衡方面发挥重要作用。Treg细胞本身也可以进一步细分为不同的亚型,其中包括自然调节性T细胞(nTreg)和诱导性调节性T细胞(iTreg)等。不同的Treg亚型可能在功能和标记基因上有所不同。
以下是一些Treg亚型及其相关Marker Gene 的示例:
Treg亚型 | Marker Gene 示例 |
自然调节性T细胞(nTreg) | FOXP3、CD25(IL2RA)、CTLA4、GITR(TNFRSF18) |
诱导性调节性T细胞(iTreg) | FOXP3、CD25(IL2RA)、CTLA4、IKZF2(HELIOS) |
这些表格列出了各种免疫细胞的一些Marker Gene 示例。请注意,这只是一个基本的示例,也可能有些地方不太准确。细胞的精确分类和Marker Gene 可能会因研究和分析方法的不同而有所不同。对于精确的细胞类型注释,通常需要使用单细胞转录组数据进行聚类和差异基因分析,以鉴定特定亚型的细胞群并确定其Marker Gene。此外,免疫细胞的功能和特性仍在研究中不断发展,细胞亚型的定义和分类也在不断演变,因此您可能需要参考最新的研究和领域专业知识,以获得更详细和准确的信息,最好根据您的数据和研究问题来选择适当的Marker Gene 和分析方法。
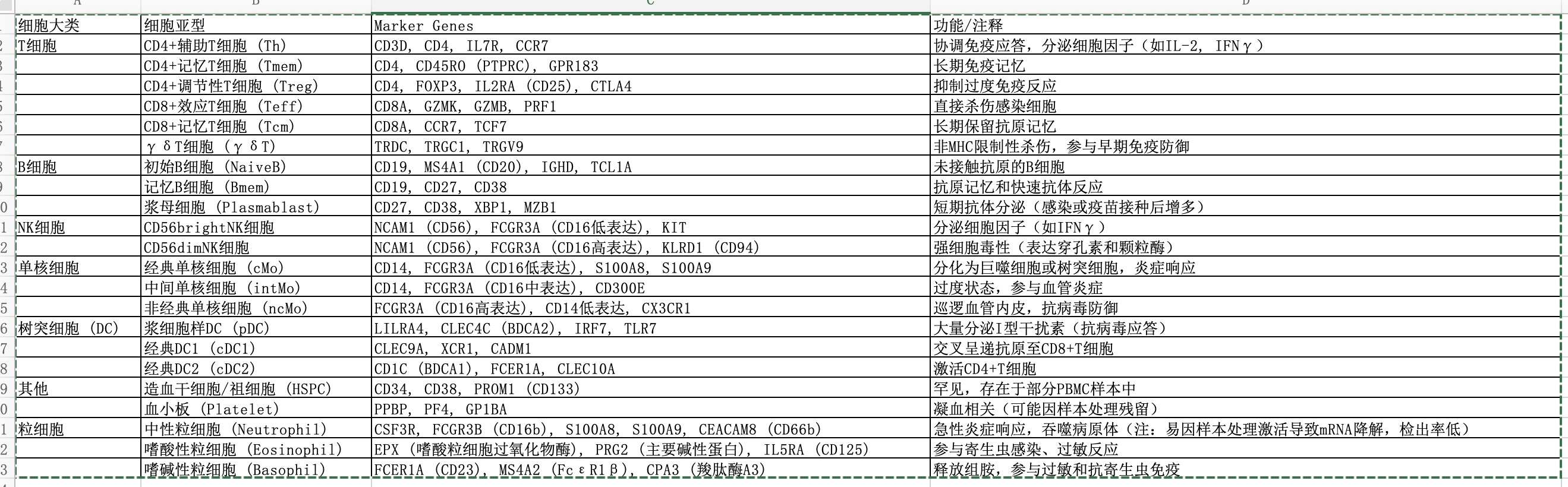
| 细胞大类 | 细胞亚型 | Marker Genes | 功能/注释 |
| T细胞 | CD4+辅助T细胞 (Th) | CD3D, CD4, IL7R, CCR7 | 协调免疫应答,分泌细胞因子(如IL-2, IFNγ) |
| CD4+记忆T细胞 (Tmem) | CD4, CD45RO (PTPRC), GPR183 | 长期免疫记忆 | |
| CD4+调节性T细胞 (Treg) | CD4, FOXP3, IL2RA (CD25), CTLA4 | 抑制过度免疫反应 | |
| CD8+效应T细胞 (Teff) | CD8A, GZMK, GZMB, PRF1 | 直接杀伤感染细胞 | |
| CD8+记忆T细胞 (Tcm) | CD8A, CCR7, TCF7 | 长期保留抗原记忆 | |
| γδT细胞 (γδT) | TRDC, TRGC1, TRGV9 | 非MHC限制性杀伤,参与早期免疫防御 | |
| B细胞 | 初始B细胞 (NaiveB) | CD19, MS4A1 (CD20), IGHD, TCL1A | 未接触抗原的B细胞 |
| 记忆B细胞 (Bmem) | CD19, CD27, CD38 | 抗原记忆和快速抗体反应 | |
| 浆母细胞 (Plasmablast) | CD27, CD38, XBP1, MZB1 | 短期抗体分泌(感染或疫苗接种后增多) | |
| NK细胞 | CD56brightNK细胞 | NCAM1 (CD56), FCGR3A (CD16低表达), KIT | 分泌细胞因子(如IFNγ) |
| CD56dimNK细胞 | NCAM1 (CD56), FCGR3A (CD16高表达), KLRD1 (CD94) | 强细胞毒性(表达穿孔素和颗粒酶) | |
| 单核细胞 | 经典单核细胞 (cMo) | CD14, FCGR3A (CD16低表达), S100A8, S100A9 | 分化为巨噬细胞或树突细胞,炎症响应 |
| 中间单核细胞 (intMo) | CD14, FCGR3A (CD16中表达), CD300E | 过度状态,参与血管炎症 | |
| 非经典单核细胞 (ncMo) | FCGR3A (CD16高表达), CD14低表达, CX3CR1 | 巡逻血管内皮,抗病毒防御 | |
| 树突细胞 (DC) | 浆细胞样DC (pDC) | LILRA4, CLEC4C (BDCA2), IRF7, TLR7 | 大量分泌I型干扰素(抗病毒应答) |
| 经典DC1 (cDC1) | CLEC9A, XCR1, CADM1 | 交叉呈递抗原至CD8+T细胞 | |
| 经典DC2 (cDC2) | CD1C (BDCA1), FCER1A, CLEC10A | 激活CD4+T细胞 | |
| 其他 | 造血干细胞/祖细胞 (HSPC) | CD34, CD38, PROM1 (CD133) | 罕见,存在于部分PBMC样本中 |
| 血小板 (Platelet) | PPBP, PF4, GP1BA | 凝血相关(可能因样本处理残留) | |
| 粒细胞 | 中性粒细胞 (Neutrophil) | CSF3R, FCGR3B (CD16b), S100A8, S100A9, CEACAM8 (CD66b) | 急性炎症响应,吞噬病原体(注:易因样本处理激活导致mRNA降解,检出率低) |
| 嗜酸性粒细胞 (Eosinophil) | EPX (嗜酸粒细胞过氧化物酶), PRG2 (主要碱性蛋白), IL5RA (CD125) | 参与寄生虫感染、过敏反应 | |
| 嗜碱性粒细胞 (Basophil) | FCER1A (CD23), MS4A2 (FcεR1β), CPA3 (羧肽酶A3) | 释放组胺,参与过敏和抗寄生虫免疫 |
HSC造血干细胞:CYTL1 GATA2,CD34,CD117
T细胞:CD3D,CD3E,CD3G,CD4,CD8
B细胞:CD79A,CD79B,CD19,MS4A1
Naïve B cell:CD19,MS4A1,IGHD,IGHM,IL4R,TCL1A
Memory B cell:CD27,CD38,IGHG
B浆细胞Plasma_B:MZB1,SDC1,JCHAIN,XBP1
NK细胞:
KLRF1,NCAM1,NCR1,GNLY,NKG7,FCGR3A
NK1(CD56+ CD16-):NCAM1,GNLY,NKG7
NK2(CD56- CD16+):FCGR3A KLRF1
髓系细胞Myeloid:
LYZ,CD68,MS4A6A,CD1E,IL3RA,LAMP3
巨噬细胞Macrophages:CSF1R,CD68,CD163,CD14
单核细胞Monocytes:S100A8,S100A9,CD68,LYZ
CD14单核细胞CD14_Mono:CD14,LYZ
CD16单核细胞CD16_Mono:LYZ,FCGR3A
树突状细胞Dendritic:
IL3RA,THBD,CD1C,CD68,CD83,FCER1A,CST3
髓源树突状细胞mDC:CD1C,FCER1A
浆样树突状细胞pDC:KLRD1,LILRA4,CLEC4C,IL3RA
中性粒细胞Neutrophil:FCGR3B,CXCR2
肥大细胞Mast:TPSAB1,KIT,CPA3,ENPP3
血小板Platelets:PPBP,GP9
血红细胞Erythrocyte:HBA1,HBD,GYPA
T细胞分类marker:
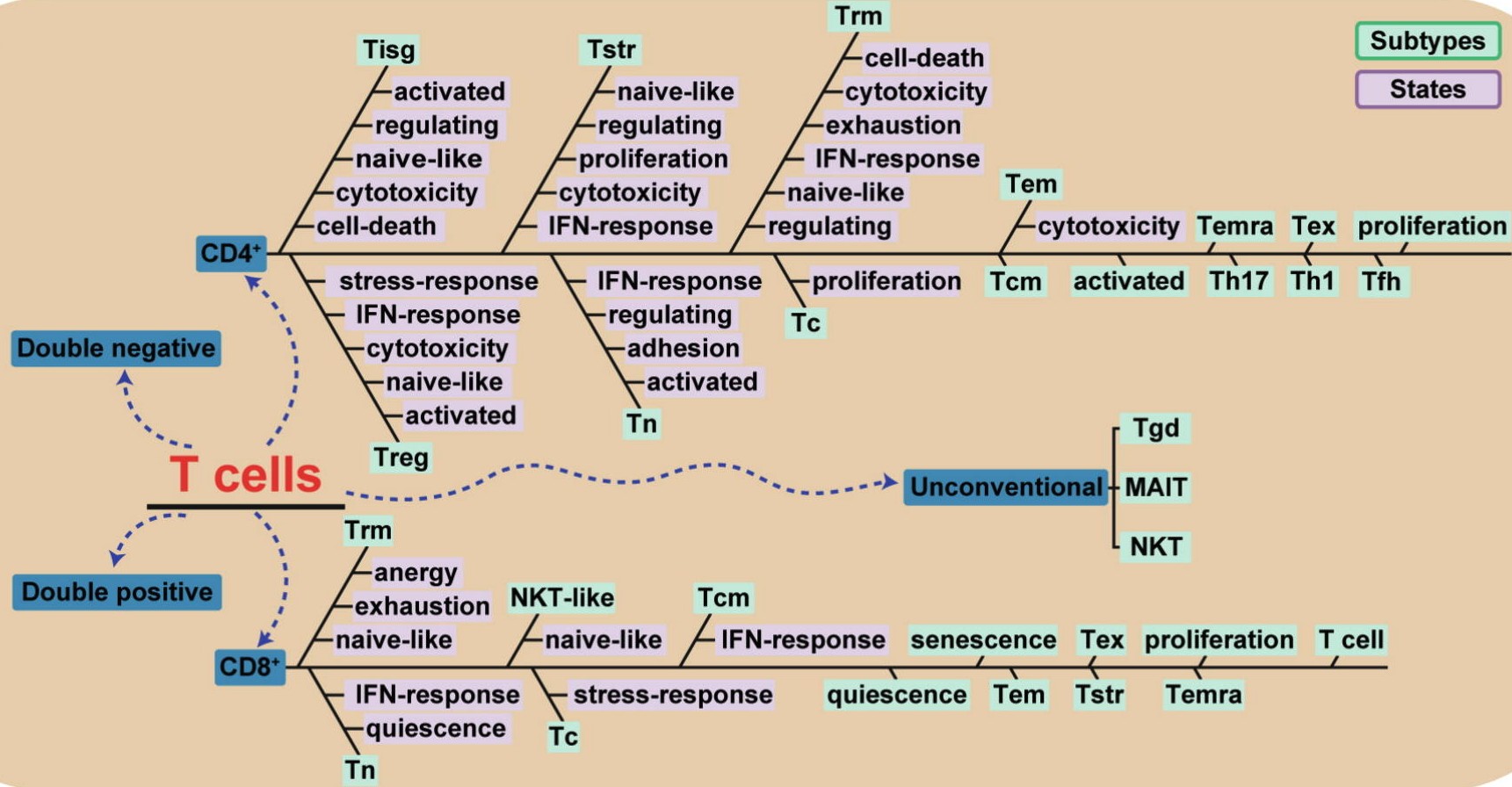
We divided T cells into CD4+ T cells, CD8+ T cells, double negative T cells, double positive T cells, and unconventional T cells. Unconventional T cells include γδ T (Tgd), natural killer T (NKT), and mucosal-associated invariant T (MAIT) cells. In total, we identified 15 subtypes of CD4+ T cells, including naive T (Tn), Treg, effector memory T (Tem), central memory T (Tcm), terminally differentiated effector memory T (Temra), exhausted T (Tex), follicular helper T (Tfh), type 1 helper T (Th1), type 17 helper T (Th17), IFN response T (Tisg), resident memory T (Trm), stress response T (Tstr), cytotoxicity T (Tc), proliferation T and activated T cells (Fig. 1c). Some of these subtypes can be further divided into multiple states, such as activated, regulating, naive-like and cell-death, in CD4+ Tisg. CD8+ T cells were divided into 13 subtypes: Tn, Trm, Tcm, Tem, Temra, Tex, Tstr, Tc, quiescence T, senescence T, proliferation T, NKT-like and CD8+ T cells (Fig. 1c). Similarly, we also identified several states in some T cell subtypes, such as anergy, naive-like and exhaustion within CD8+ Trm cells. In total, we identified 68 different subtypes/states as a high-confidence reference of T cell subsets, which highly expressed their corresponding marker genes. All marker genes are available in the TCellAtlas database and Table S5 (online).
Tisg: IFN response T;
Tstr: stress response T;
Trm: resident memory T;
Tem: effector memory T;
Temra: terminally differentiated effector memory T;
Tex: exhausted T;
Treg: regulatory T;
Tn: naive T;
Tc: cytotoxicity T;
Tcm: central memory T;
Th17: type 17 helper T;
Th1: type 1 helper T;
Tfh: follicular helper T;
Tgd: γδ T;
NKT: natural killer T;
MAIT: mucosal-associated invariant T.
An automatic annotation tool and reference database for T cell subtypes and states at single-cell resolution. Wen-Kang Shen, Chu-Yu Zhang, Yi-Min Gu, Tao Luo, Si-Yi Chen, Tao Yue, Gui-Yan Xie, Yu Liao, Yong Yuan, Qian Lei, and An-Yuan Guo, Science Bulletin. 2025 Mar. https://doi.org/10.1016/j.scib.2025.02.043
| CD4 | Tn | ['CCR7', 'SELL', 'TCF7', 'LEF1','IL7R'] |
| CD4 | Treg | ['FOXP3', 'CTLA4', 'IL2RA', 'IKZF2', 'TNFRSF9', 'TIGIT', 'CCR8', 'RTKN2', 'BATF', 'TNFRSF4', 'IL32', 'TNFRSF18', 'RGS1', 'CD74', 'LAYN', 'IL1R2', 'DUSP4', 'TBC1D4'] |
| CD4 | Th1 | ['IFNG', 'CCL4', 'CCL5', 'ID2', 'CXCL13', 'DUSP2', 'HAVCR2', 'PDE7B', 'PDCD1', 'GZMA', 'ZBED2', 'MYO7A', 'GZMB', 'BHLHE40', 'RBPJ'] |
| CD4 | Th17 | ['KLRB1', 'CCR6', 'CAPG', 'CCL20', 'IL7R', 'RORA', 'IL4I1', 'ANXA1', 'CXCR6', 'ANKRD28', 'CEBPD', 'IL23R', 'TNFSF13B', 'MGAT4A', 'RORC', 'CTSH', 'CD40LG'] |
| CD4 | Tfh | ['CXCR5', 'PDCD1', 'TNFSF8', 'IL6ST', 'TOX2', 'TOX', 'CXCL13', 'NR3C1', 'IL21'] |
| CD4 | Tcm | ['KLF2', 'TRADD', 'ICAM2', 'PTGER2', 'GPR183', 'LTB', 'S100A10', 'FLT3LG', 'SORL1'] |
| CD4 | Tem | ['GZMK', 'GZMA', 'KLRB1', 'CCL5', 'CCL4', 'CST7', 'EOMES', 'ITM2C', 'CD4', 'F2R', 'IFNG'] |
| CD4 | Tex | ['TNFRSF18', 'CTLA4', 'RGS1', 'DUSP4', 'PDCD1', 'TIGIT', 'SRGN', 'CXCL13', 'GAPDH', 'DNPH1'] |
| CD4 | Tc | ['GZMA', 'GZMB', 'GZMK', 'CD4', 'GZMH', 'NKG7', 'CCL5', 'CST7'] |
| CD4 | Temra | ['PLEK', 'NKG7', 'GZMH', 'FGFBP2', 'GNLY', 'PRF1', 'CX3CR1', 'S1PR5', 'CTSW'] |
| CD8 | Tn | ['CCR7', 'SELL', 'TCF7', 'LEF1', 'TRABD2A', 'ACTN1', 'LDLRAP1', 'NOSIP', 'TXK', 'MAL'] |
| CD8 | Tex | ['HAVCR2', 'CCL3', 'CXCL13', 'CTLA4', 'TIGIT', 'ENTPD1', 'PDCD1', 'CXCR6', 'TNFRSF9'] |
| CD8 | Trm | ['ITGA1', 'XCL1', 'CCL5', 'CAPG', 'XCL2', 'JUN', 'CXCR3', 'ID2', 'CXCR6', 'ZNF683', 'CD8A', 'GZMA', 'CKLF', 'JAML'] |
| CD8 | Tem | ['GZMK', 'CX3CR1', 'GZMH', 'CST7', 'DUSP2', 'CD74', 'CXCR4', 'EOMES', 'SH2D1A', 'GZMA', 'CD8B', 'CCL5', 'CMC1', 'GZMB', 'KLRD1', 'GNLY'] |
| CD8 | Tcm | ['CREM', 'FAM177A1', 'LDHA', 'CCR7', 'OAZ1', 'CMC1', 'CLDND1', 'SARAF', 'FTH1', 'TRAT1', 'MLLT3', 'GGA2', 'CYSTM1', 'DUSP2', 'CST7', 'IL7R', 'DKK3', 'ITM2C', 'GPR183', 'TRMO', 'ATP6V0C'] |
| CD8 | Tc | ['PLEK', 'KLRG1', 'GNLY', 'FCRL6', 'GZMH', 'NKG7', 'FGFBP2', 'CX3CR1', 'ADGRG1', 'S1PR5', 'FGR', 'FCGR3A', 'HOPX', 'GZMK', 'EEF1A1P5', 'TMIGD2', 'CCR6', 'ZEB2', 'CD8B', 'KLRD1', 'TMSB4XP8', 'KLRK1', 'PTPRCAP', 'SPON2', 'CD8A', 'SLC9A3R1', 'PRF1', 'PLAC8'] |
| CD8 | Temra | ['CX3CR1', 'GZMH', 'FCGR3A', 'GZMB', 'S1PR5', 'NKG7', 'KLRD1', 'FGR', 'PLEK', 'FGFBP2', 'GNLY'] |
| Unconventional | MAIT | ["KLRB1", "SLC4A10", "NCR3", "IFNGR1", "LST1", "ZBTB16", "DPP4", "IL23R", "DUSP1", "ME1", "JAML", "IL18RAP", "RORC", "COLQ", "RORA", "CCR6", "SLAMF1"] |
| Unconventional | Tgd | ["TRDC", "TRDV2", "GZMB", "TRDV1", "CD3D", "TRGC1"] |
| Unconventional | NKT | ["KIR2DL4", "NKG7", "GZMM", "ZMAT4", "CD122", "NKG2D", "GZMB", "GNLY", "COX6A2", "GZMA", "CD314", "AC022126.1", "IKZF2", "TYROBP", "XCL2", "XCL1", "KIR3DL2", "KIR2DL3", "CD69", "IL2RB", "CD161", "CD3D", "CD11b", "CD56", "CD16", "CD94"] |
| States | stress-response | ['ATM', 'ATR', 'BAG3', 'CCAR2', 'DNAJB1', 'DNAJB6', 'DNAJC2', 'DNAJC7', 'GSK3B', 'HIKESHI', 'HSF1', 'HSPA1A', 'HSPA1B', 'HSPA5', 'HSPA6', 'HSPA8', 'MAPK1', 'MAPK3', 'MAPKAPK2', 'NUP62', 'RPA1', 'RPA2', 'RPA3', 'SEH1L', 'SIRT1', 'TPR', 'YWHAE'] |
| States | anergy | ['NR4A1', 'LAG3', 'NFATC2', 'CBLB', 'RNF128', 'DGKA', 'EGR3', 'NT5E', 'EGR2', 'NRP1', 'TOB1', 'ITCH', 'IZUMO1R'] |
| States | cell-death | ['CASP8', 'RPS27A', 'TNFRSF10B', 'RIPK1', 'TNFSF10', 'UBA52', 'FASLG', 'YWHAE', 'BIRC3', 'BIRC2', 'UBB', 'UBC', 'XIAP', 'RIPK3', 'CFLAR', 'TRAF2', 'AKT1', 'TNFRSF1A', 'MAPK8', 'MAGED1', 'TNFRSF10A', 'TRADD', 'CASP3', 'BID', 'BAD', 'FADD', 'BCL2L11', 'FAS'] |
| States | naive-like | ['BTG2', 'FOXP1', 'IL7R', 'TCF7', 'BTG1', 'ACTN1', 'SELL', 'KLF2', 'KLF3', 'LEF1', 'CCR7', 'TOB1', 'FOXO1', 'LTB'] |
| States | adhesion | ['ITGA4', 'ITGA6', 'ITGA8', 'ITGA9', 'ITGAL', 'ITGAM', 'ITGAV', 'ITGB1', 'ITGB2', 'ITGB7', 'ITGB8', 'ITGA1'] |
| States | senescence | ['CDKN2B', 'KLRK1', 'IFNA1', 'ATM', 'TAB1', 'TSC2', 'MAPK8', 'MAP3K5', 'TP53', 'IFNA2', 'IFNAR2', 'MAPK9', 'TERF2', 'IFNAR1', 'CDKN2A', 'CDKN1A', 'MAPK14'] |
| States | exhaustion | ['NR4A1', 'IRF4', 'EOMES', 'BATF', 'PDCD1', 'PTPN2', 'PTPN6', 'TIM3', 'PTPN11', 'PRDM1', 'ENTPD1', 'LAG3', 'GATA3', 'LAYN', 'CTLA4', 'HAVCR2', 'TIGIT', 'TOX'] |
| States | regulating | ['FOXP3', 'IKZF2', 'IKZF4', 'IL2RA', 'ENTPD1', 'CCR4', 'ICOS', 'IL10RA', 'TGFB1', 'TIGIT', 'CTLA4', 'LAG3', 'HAVCR2', 'PDCD1'] |
| States | cytotoxicity | ['PRF1', 'GZMA', 'GZMB', 'GZMH', 'GNLY', 'FGFBP2', 'GZMK', 'GZMM', 'NKG7', 'IFNG', 'CTSW', 'CST7'] |
| States | proliferation | ['BAX', 'CCND1', 'CCNB1', 'HMGB2', 'IGFBP2', 'MKI67', 'TOP2A', 'CD24', 'IGF1', 'PTPRC', 'ABL1', 'CD40LG', 'RIPK2', 'EBI3', 'IL2', 'IL23A', 'IL4', 'CD86', 'SLAMF1', 'TNFSF4', 'TNFSF9', 'IL18', 'SYK', 'STAT5B', 'TNFRSF4', 'HMGB1', 'AGER', 'CCL5'] |
| States | quiescence | ['BTG2', 'FOXP1', 'TGFBR1', 'VSIR', 'BTG1', 'TSC2', 'TSC1', 'KLF2', 'TOB1', 'FOXO1', 'RUNX1'] |
| States | IFN-response | ['IFITM3', 'IFITM2', 'IFNAR2', 'IFNGR1', 'IRF1', 'IRF7', 'JAK1', 'JAK2', 'OAS1', 'OAS2', 'OAS3', 'PARP14', 'IFIH1', 'STAT1', 'STAT2', 'PARP9', 'IFITM1', 'ISG15'] |
| States | activated | ['TNFRSF10B', 'TNFRSF10C', 'TNFRSF10D', 'TNFRSF11A', 'TNFRSF12A', 'TNFRSF13B', 'TNFRSF13C', 'TNFRSF14', 'TNFRSF17', 'TNFRSF18', 'TNFRSF19', 'TNFRSF1A', 'TNFRSF1B', 'TNFRSF21', 'TNFRSF25', 'TNFRSF4', 'TNFRSF6B', 'TNFRSF8', 'TNFRSF9'] |
References:
Miao, Y.-R. et al. ImmuCellAI: A Unique Method for Comprehensive T‐Cell Subsets Abundance Prediction and its Application in Cancer Immunotherapy. Adv Sci 10 (2020).
Yang, J. et al. TCellSI: A novel method for T cell state assessment and its applications in immune environment prediction. iMeta e231 (2024) doi:10.1002/imt2.231.
Ianevski, A., Giri, A. K. & Aittokallio, T. Fully-automated and ultra-fast cell-type identification using specific marker combinations from single-cell transcriptomic data. Nat. Commun. 13, 1246 (2022).
Domínguez Conde, C. et al. Cross-tissue immune cell analysis reveals tissue-specific features in humans. Science 376, eabl5197 (2022).
Zheng, L. et al. Pan-cancer single-cell landscape of tumor-infiltrating T cells. Science 374, abe6474.
Chu, Y. et al. Pan-cancer T cell atlas links a cellular stress response state to immunotherapy resistance. Nat. Med. (2023) doi:10.1038/s41591-023-02371-y.
Zheng, C. et al. Landscape of Infiltrating T Cells in Liver Cancer Revealed by Single-Cell Sequencing. Cell 169, 1342-1356.e16 (2017).
Guo, X. et al. Global characterization of T cells in non-small-cell lung cancer by single-cell sequencing. Nat. Med. 24, 978–985 (2018).
Zhang, L. et al. Lineage tracking reveals dynamic relationships of T cells in colorectal cancer. Nature 564, 268–272 (2018).
Wang, X. et al. Single‐Cell RNA‐Seq of T Cells in B‐ALL Patients Reveals an Exhausted Subset with Remarkable Heterogeneity. Adv. Sci. 8, 2101447 (2021).
Tacutu, R. et al. Human Ageing Genomic Resources: new and updated databases. Nucleic Acids Res. 46, D1083–D1090 (2018).
ElTanbouly, M. A. & Noelle, R. J. Rethinking peripheral T cell tolerance: checkpoints across a T cell’s journey. Nat. Rev. Immunol. 21, 257–267 (2021).
Yuan, H. et al. CancerSEA: a cancer single-cell state atlas. Nucleic Acids Res. 47, D900–D908 (2019).
van der Leun, A. M., Thommen, D. S. & Schumacher, T. N. CD8+ T cell states in human cancer: insights from single-cell analysis. Nat. Rev. Cancer 20, 218–232 (2020).
Bhattacharya, S. et al. ImmPort, toward repurposing of open access immunological assay data for translational and clinical research. Sci. Data 5, 180015 (2018).
Yu, Y. et al. Single-cell sequencing of immune cells after marathon and symptom-limited cardiopulmonary exercise. iScience 26, (2023).
Kanehisa, M., Furumichi, M., Sato, Y., Ishiguro-Watanabe, M. & Tanabe, M. KEGG: integrating viruses and cellular organisms. Nucleic Acids Res. 49, D545–D551 (2021).
Agrawal, A. et al. WikiPathways 2024: next generation pathway database. Nucleic Acids Res. 52, D679–D689 (2024).
Milacic, M. et al. The Reactome Pathway Knowledgebase 2024. Nucleic Acids Res. 52, D672–D678 (2024).
The Gene Ontology Consortium et al. The Gene Ontology knowledgebase in 2023. GENETICS 224, iyad031 (2023).
- 发表于 2026-01-19 17:48
- 阅读 ( 548 )
- 分类:转录组