deepseek绘图之--多组学瀑布图
在多组学分析中,经常需要绘制多组学瀑布图,用于展示不同组学数据(如转录组学、蛋白组学、代谢组学等)在同一通路内不同类型分子的贡献差异。这种瀑布图可以直观呈现同一通路内各差异特征对组间差异的贡献强度,为快速锁定核心调控基因提供高效可视化方案。然而,传统工具如R、Python的绘图流程往往涉及繁琐的文件处理、颜色搭配调试和图例优化等,让研究者耗费大量时间,更是令生信新手望而生畏。AI大模型的介入彻底改变了这一局面。只需提供您的不同组学数据,并用自然语言描述定制需求,AI即可自动生成绘图脚本,真正实现"专业绘图,一键生成"。本文将以转录组与代谢组为例,带您体验如何通过AI协作,在短短5分钟内完成一幅专业的多组学瀑布图,助您在科研工作中以极致的效率和专业度获得竞争优势!
初步构建绘图提示词
要绘制转录组与代谢组瀑布图,为了让他更高效的出图,我们尽量描述的详细一点,这样能够减少我们后面修改的次数,更容易得到想要的结果。首先确认自己的路径和绘图需要的文件,分别是:
通路富集的差异基因矩阵

通路富集的差异代谢物矩阵
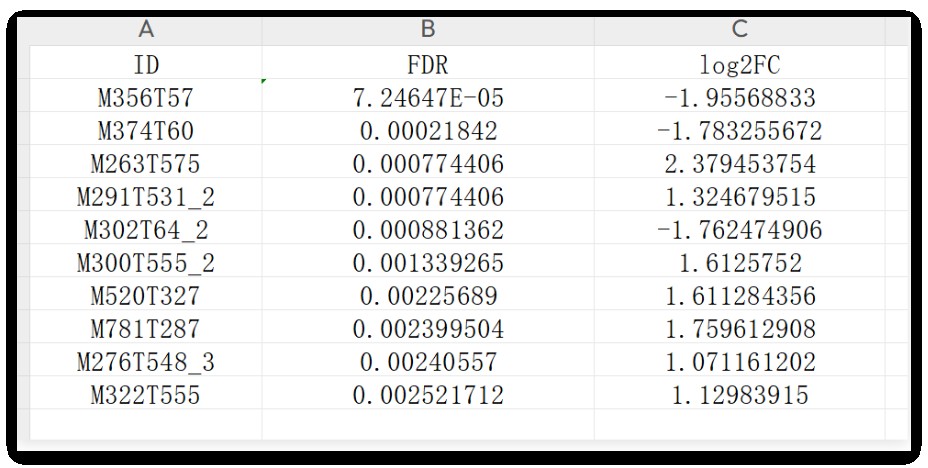
这里只截取了一小部分,类似的文件大家肯定见过,这里就不多赘述了,
写完了的提示词如下,大家可以参考一下格式:
------------------------------------------
读取两个文件(用read.table读取)
工作路径: /share/nas1/wangq/project/test
差异基因矩阵: deg.txt(第一列基因ID,第二列FDR值,第三列log2FC值)
差异代谢物矩阵:dem.txt (第一列代谢物ID,第二列FDR值,第三列log2FC值)
绘制差异基因与差异代谢组同一通路基因和代谢物瀑布图,横坐标使用|log2FC|*(-log10(p值))作为综合得分,衡量特征对组间差异的贡献,纵坐标为该通路所有差异基因和差异代谢物。上调为红色,下调为蓝色
绘图工具: ggplot2
图形类型:瀑布图
------------------------------------------
在deepseek输出上面写好的提示词,如果问题复杂可以打开deepthink,
这样简单的就不需要了。
初步生成绘图代码

AI给出了详细的代码,我们来到RStudio运行一下,结果如下:
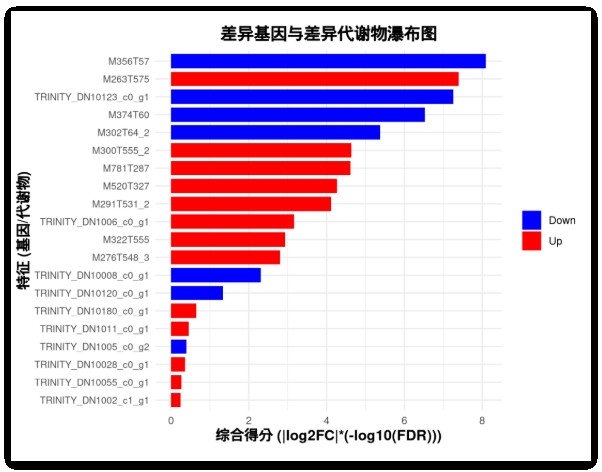
可以看到生成的图片没有问题,只是颜色主题并不好看,我们让他修改一下。
让AI帮助我修改可视化效果

修改后的绘图如下所示,比较符合我们的要求,如果有其他需求,也可以让他再调。
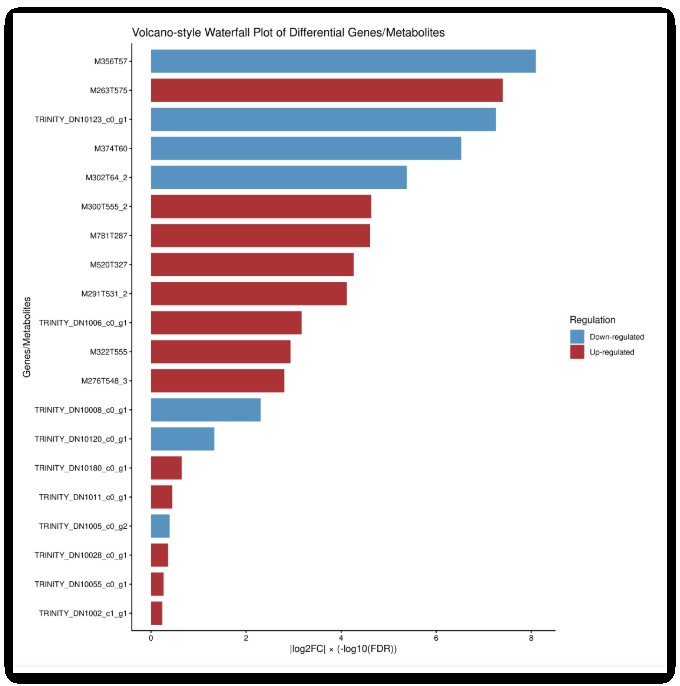
最终绘图代码
# 设置工作路径 setwd("/share/nas1/wangq/project/test") # 加载所需库 library(ggplot2) library(dplyr) # 读取数据 deg <- read.table("deg.txt", header = TRUE, sep = "\t", stringsAsFactors = FALSE) dem <- read.table("dem.txt", header = TRUE, sep = "\t", stringsAsFactors = FALSE) # 为数据添加类型标识并规范列名 deg <- deg %>% mutate(type = "Gene") %>% rename(ID = 1, FDR = 2, log2FC = 3) dem <- dem %>% mutate(type = "Metabolite") %>% rename(ID = 1, FDR = 2, log2FC = 3) # 合并数据 combined_data <- rbind(deg, dem) # 计算综合得分并判断上调/下调 combined_data <- combined_data %>% mutate(score = abs(log2FC) * (-log10(FDR)), direction = ifelse(log2FC > 0, "Up", "Down")) # 按综合得分排序(确保瀑布图顺序) combined_data <- combined_data %>% arrange(score) %>% mutate(ID = factor(ID, levels = ID)) # 锁定排序 # 绘制瀑布图(使用theme_classic()主题) ggplot(combined_data, aes(x = score, y = ID, fill = direction)) + geom_col(width = 0.8) + scale_fill_manual(values = c("Up" = "#AA3538", "Down" = "#5891BF")) + # 指定颜色 labs( x = "|log2FC| × (-log10(FDR))", # x轴标签 y = "Genes/Metabolites", # y轴标签 title = "Volcano-style Waterfall Plot of Differential Genes/Metabolites" # 标题 ) + theme_classic() + # 改为classic主题 theme( plot.title = element_text(hjust = 0.5, size = 14, face = "bold"), # 标题居中加粗 axis.text.y = element_text(size = 8), # 调整y轴文本大小 axis.title = element_text(size = 12, face = "bold"), # 坐标轴标题加粗 legend.title = element_blank() # 隐藏图例标题 ) # 保存图形 ggsave("volcano_style_waterfall_classic.png", width = 10, height = 8, dpi = 300)
- 发表于 2025-10-31 17:03
- 阅读 ( 1408 )
- 分类:R
