生信分析-小提琴图绘制-脚本使用
小提琴图(Violin plot)可以用来展示多组数据的分布状态以及概率密度,这种图表结合了箱线图和密度图的特征,常用于生信分析中。
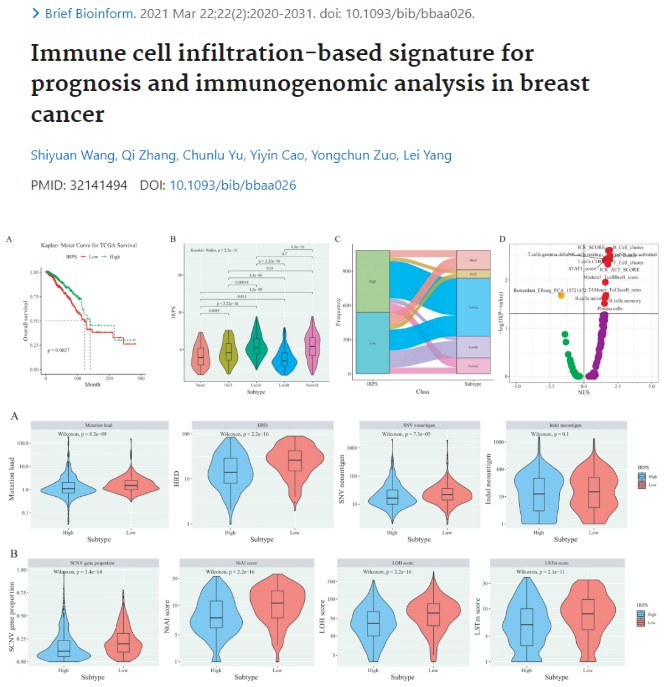
例如:2021年发表在Briefings in Bioinformatics上的一篇影响因子为11.62的文献为例,该文献中大量运用了小提琴图来展示多组数据的分布状态、概率密度以及它们之间的差异。
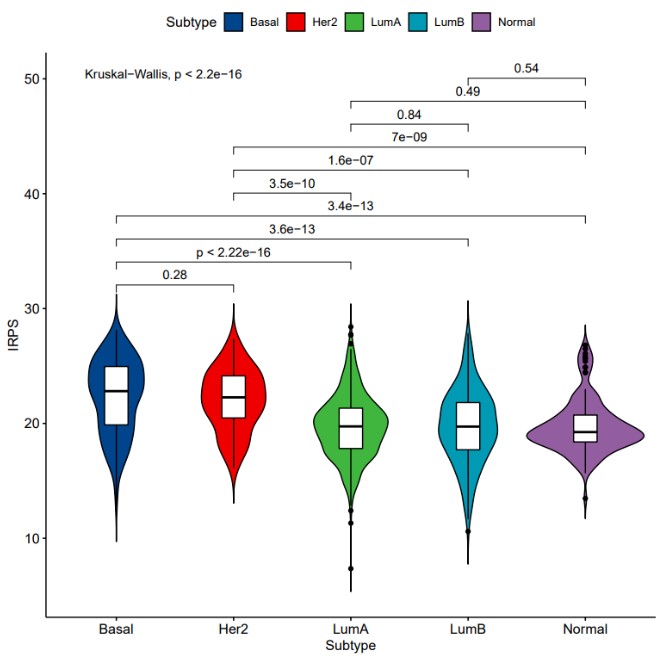
从小提琴图中我们不仅可以直观地了解数据的中位数、四分位数、异常值等信息,还可以进行差异分析,通过显著性P值来判断两组数据的差异程度。例如第一张图B中,作者将样本分为五组,绘制小提琴图展示了免疫相关预后评分(IRPS)在不同分组的分布状态,并通过显著性P值直观地说明了两两分组之间IRPS的差异。
为了使大家更简便快捷地绘制出精美的小提琴图,这里我们给大家提供一个绘制小提琴图的R脚本,这个脚本只需要准备好相应的输入文件,再进行简单的命令行操作即可绘制出两组或多组数据的小提琴图,并可在两两之间进行差异分析。
使用命令示例如下
#当分组数目为两组时,合适的标签高度需根据数据值的大小调试
Rscript compare_violin_boxplot.r -m metadata_gene.tsv \
-n PDCD1 -g IRPS_group -s 2 -t wilcox.test -l 8 -o boxplot -p test

#当分组数目大于两组时,合适的标签高度需根据数据值的大小调试
Rscript /share/work/fangs/scripts/tcga/compare_violin_boxplot.r -m metadata_group.tsv \
-n IRPS -g Subtype -s 5 -t kruskal.test \
-f 31 -b 2 -l 50 -o boxplot -p IRPS
输入文件准备
这个脚本所必需的输入文件只有一个,通过-m参数指定(metadata_group.tsv),文件中每一行为一个样本,列必须包含基因表达量等数值型数据以及样本的分组信息,如下列表格所示:
| barcode | PDCD1 | Subtype |
| TCGA-A8-A07B-01A-11R-A00Z-07 | 1.50899055035449 | LumA |
| TCGA-A8-A08B-01A-11R-A00Z-07 | 2.078678925608 | Her2 |
| TCGA-A8-A08P-01A-11R-A00Z-07 | 2.91142321338723 | LumB |
更多脚本参数设置及说明
初次使用脚本时,可以通过-h参数获得以下帮助信息。
Rscript compare_violin_boxplot.r -h
usage: compare_violin_boxplot.r
[-h] -m meta [-n name] [-g group_name] [-s group_num] [--log2]
[-t method] [-f from] [-b by] [-l lab_height] [-H HEIGHT] [-W WIDTH]
[-o OUTDIR] [-p PREFIX]
Difference comparison plot(violin_box)
optional arguments:
-h, --help show this help message and exit
-m meta, --meta meta Input the clinical information file path that contains
the grouping[required]
-n name, --name name The column name of the value[optional,default:IRPS]
-g group_name, --group_name group_name
Specifies the column name that contains grouping
information[optional,default:TCGA_Subtype]
-s group_num, --group_num group_num
Number of groups[optional,default:5]
--log2 Whether to perform log2
processing[optional,default:False]
-t method, --method method
A method used to compare differences[optional:t.test/w
ilcox.test/anova/kruskal.test,default:kruskal.test]
-f from, --from from Base height of the label[optional,default:17]
-b by, --by by The difference between two tag
heights[optional,default:2]
-l lab_height, --lab_height lab_height
Label height[optional,default:35]
-H HEIGHT, --height HEIGHT
the height of dumbbell plot[optional,default 8]
-W WIDTH, --width WIDTH
the width of dumbbell plot[optional,default 8]
-o OUTDIR, --outdir OUTDIR
output file directory[optional,default cwd]
-p PREFIX, --prefix PREFIX
out file name prefix[optional,default kegg]
根据自身需要调整必要的参数:
-n 输入列名,指定用于绘制小提琴图的一列数据;
-g 输入列名,指定根据哪一列分组信息对样本进行分组;
-s 指定分组的数目,最小为2,最大为5;
-t 指定差异分析的方法,如t.test(t检验)、wilcox.test(秩和检验)等;
-l 指定显著性P值标签的高度,需根据数据的值的大小进行调整。
其中需要注意的是,当绘制仅分为两组的小提琴图时,指定以上参数即可,但如果绘制两组以上的小提琴图时,必须还要指定-f和-b参数。这两个参数调整的是两两分组之间显著性P值标签的位置,-f参数指定的是两两分组之间显著性P值标签的最低高度,-b参数指定的是每个标签的间隔大小。
其他参数说明
--log2 加上该参数即要对连续变量进行log2转化;
-H 指定输出图片的高度,默认高为8英寸;
-W 指定输出图片的宽度,默认宽为8英寸;
-o 指定结果输出的路径,默认为当前路径;
-p 指定输出文件名字的前缀。
脚本获取方法
在“生信博士“公众号回复kmplot即可获得网盘链接以及提取码。
如何使用命令行的方法分析数据
可能有的人没有用过命令的形式分析数据, 可以学习下面的课程入门一下:

- 发表于 2022-06-10 15:48
- 阅读 ( 6428 )
- 分类:R
你可能感兴趣的文章
- 芯片差异分析分组矩阵 6700 浏览
- 如何绘制一份韦恩图 11678 浏览
- p值还是 FDR ? 13387 浏览
