R语言对基因表达量数据RNA-seq做PCA分析
PCA(principal component analysis )主成分分析,可以分析样品之间相关性,确定样品总体上的差异,或者查看是否有批次效应等
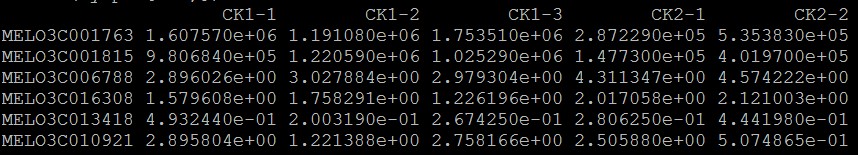
输入数据:
代码部分,筛选基因也可以参照另一篇文章,而不一定是选取200个变化最大的基因,R筛选基因:
myfpkm<-read.table("All_gene_fpkm.xls",header=TRUE,comment.char="",sep = "\t",check.names=FALSE,row.names=1)
probesetvar = apply(myfpkm, 1, var) #表达变化大的基因
ord = order(probesetvar, decreasing=TRUE)[1:200] #前200个基因,或者更多
pca = prcomp(t(myfpkm[ord,]), scale=TRUE)
ss=summary(pca)
#绘图:
plot(pca$x[,1:2],col=rep(c(1,2,3,4,1,2,3,4),each=3),pch=rep(c(16,17),each=12))
#或者3D:
library(scatterplot3d)
scatterplot3d(pca$x[,1:3],color=rep(c(1,2,3,4,1,2,3,4),each=3),pch=rep(c(16,17),each=12))
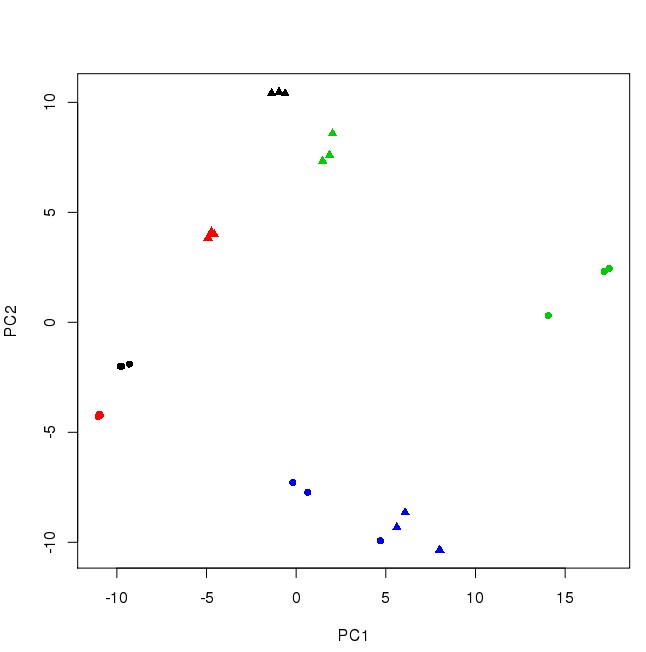
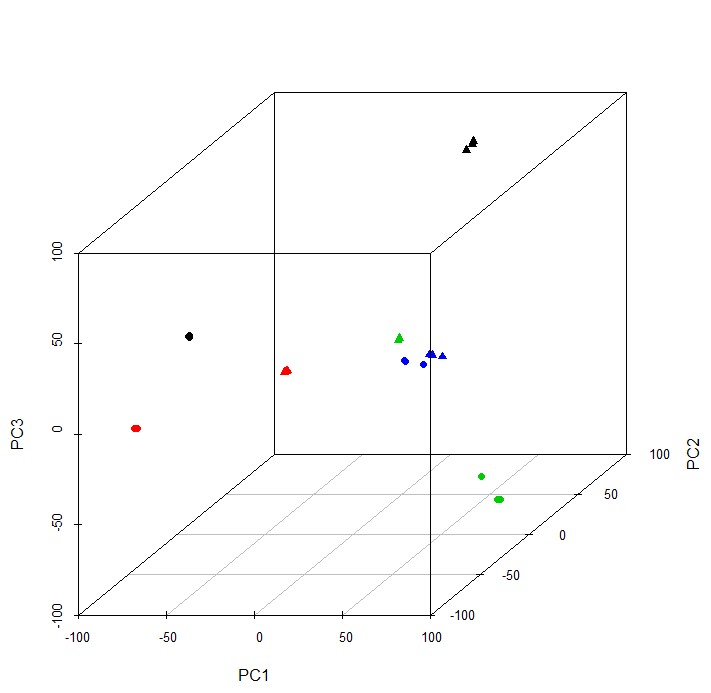
参考文献:https://www.nature.com/articles/nprot.2009.97
更多生物信息课程:https://study.omicsclass.com/index
- 发表于 2018-06-04 15:21
- 阅读 ( 33204 )
- 分类:转录组
你可能感兴趣的文章
- 多组学联合分析—桑基图 450 浏览
- 一文教你绘制GO富集分析弦图,简单又好看 433 浏览
- R脚本绘制多层环形饼图 3011 浏览
- corrplot包绘制相关性图,简单又好看! 3131 浏览
- 已知集合交集数量绘制韦恩图 3642 浏览
- 绘制Upset 图 6197 浏览
0 条评论
请先 登录 后评论
