如何利用NCBI提取特定位置的基因组序列?
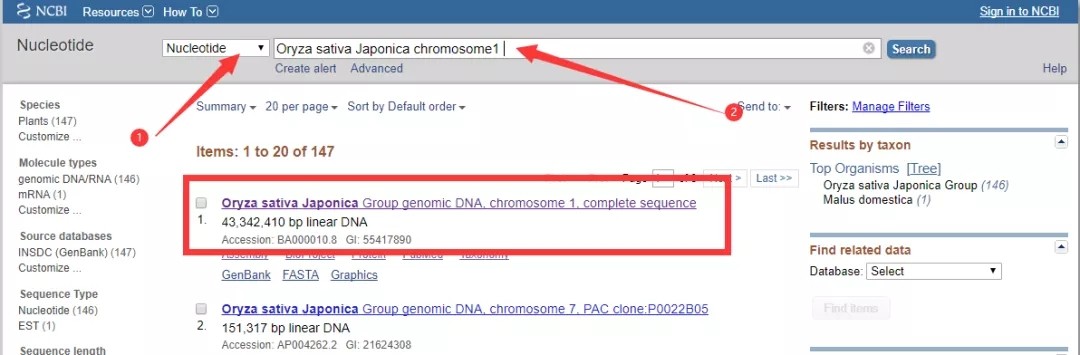
小编经常遇到这样的一些个性化分析,例如,下载某物种某染色体XXXX-XXXX碱基区域内的全部基因信息,尤其是BSA、QTL定位、GWAS等项目客户,该类需求是很多的;之前给大家分享过一个利用Ensembl数据库网站提供的biomart工具进行基因组特定区间基因及注释信息的提取,详见链接:[原创]-如何下载特定区间内基因及注释信息?但由于ensembl收录基因组数量有限,对于未收录物种就只能另寻他法了。今天我再给大家演示一个利用NCBI数据库进行基因组特定序列提取的方法,希望对您的科研学习有所帮助。我们以水稻(日本晴)1号染色体正义链1234-4321位置序列为例进行介绍。1. 打开NCBI,类目选择Nucleotide,检索框输入日本晴拉丁名及染色体号(Oryza sativa Japonica chromosome1 ),点search检索到日本晴1号染色体序列,如下图:
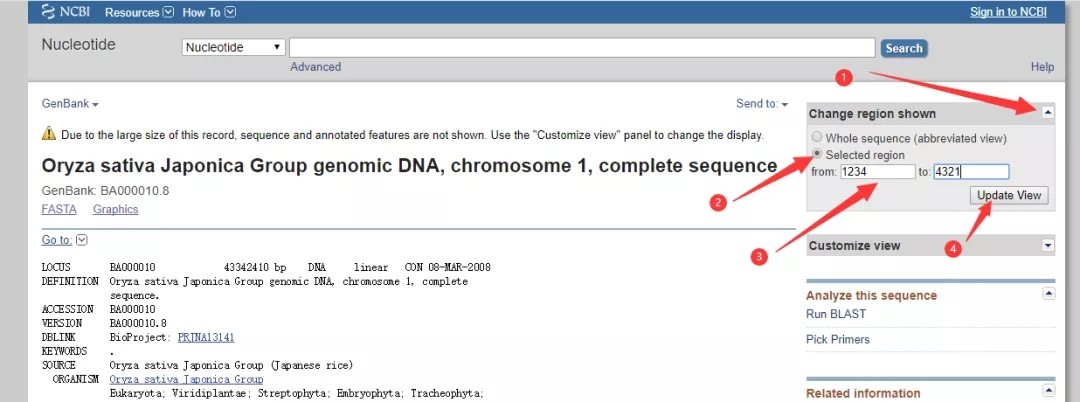
2. 点击上图检索到的日本晴1号染色体序列进入下一步,如下图,在页面右侧有Change region shown选项,展开后选择Selected region 并输入1234-4321位置信息,点击Update view进入下一步。
 3. 点击页面左上角gene bank,格式更改为 FASTA,然后页面就自动展示该区域序列信息了。
3. 点击页面左上角gene bank,格式更改为 FASTA,然后页面就自动展示该区域序列信息了。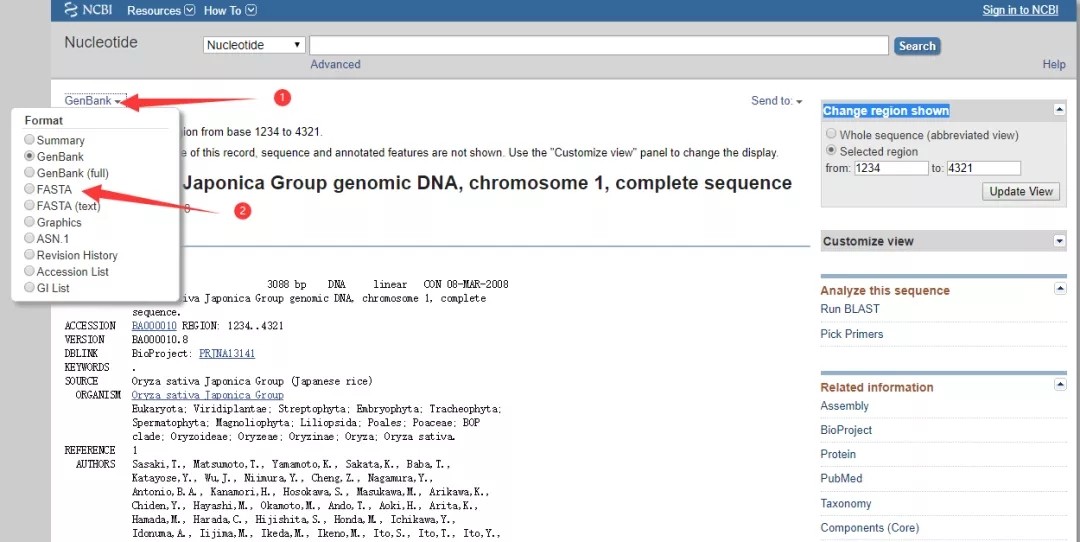 4. 假如想知道该区域的反义链序列则可进行下方操作:在步骤3的基础上,点击页面右侧的Customize view选项,勾选Show reverse complement即可。
4. 假如想知道该区域的反义链序列则可进行下方操作:在步骤3的基础上,点击页面右侧的Customize view选项,勾选Show reverse complement即可。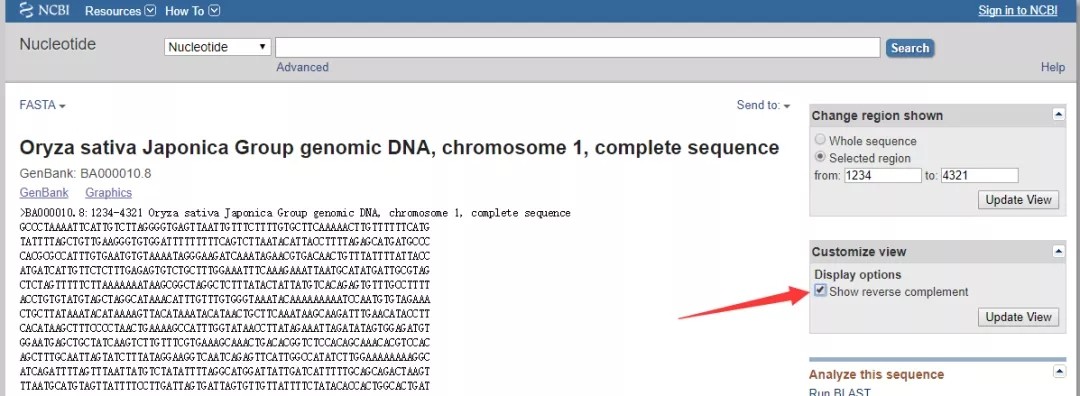 好了,今天的生信小技能就介绍到这里,更多生信小技能大家可以学习下下方课程:
好了,今天的生信小技能就介绍到这里,更多生信小技能大家可以学习下下方课程:日常科研中你我经会常遇到看不懂的图表,不会挖掘的数据,没有思路的文章,沟通不畅的个性化分析,求人不如求己,一切痛点都能解决:
1. 单细胞/空间转录组正在大火,高分文章必备,0基础学单细胞/空间转录组分析,做更牛的个性化分析,绘更漂亮的图,点这个链接:单细胞/空间转录组分析实操
2. 2区文章发文新思路,0实验就能发文章,省钱省时间,套路化内容好写易发表,0基础学习最新版基因家族分析/泛基因家族分析链接:最新基因家族分析 ;最新版泛基因家族分析
3. 转录组越做越普遍,实验必备,看不懂结果?不会深入分析?自学都可以搞定,学习链接:有参转录组自主分析实操 ;转录组与代谢组结果解读/个性化数据分析 ;
4. 代谢组分析硬件要求不高,个人电脑就可以分析,0基础学习链接:代谢组分析实操
3. 现在才是发表T2T基因组/泛基因组文章的好时机,成本低,好发表,做好高级个性化分析发表高分1区不是梦!T2T基因组/比较基因组/泛基因组分析学习链接:T2T基因组组装与注释分析;动植物泛基因组分析 ;比较基因组分析
4. 群体重测序遗传进化分析+GWAS文章,篇篇10+分,缩短分析周期,提升文章亮点,勇敢冲击NGS顶刊,学习链接:群体遗传进化GWAS分析 ;
5. 传统图位克隆研究方法经久不衰,有遗传群体,有性状,自己就可以做,学习链接:遗传图谱构建与QTL分析
6. 微生物16S/18S/ITS多样性分析和宏基因组分析,学习链接:宏基因组分析 ;微生物16S/18s/ITS多样性分析
7. 细胞器基因组与比较基因组分析是真正的无需实验就可以发表2区期刊的文章思路,成本低,文章好发,性价比极高,学习链接:线粒体基因组分析实操 ;叶绿体基因组分析实操
8. 数据分析硬件利器,个人分析能力的倍增器,好用便宜的云生信服务器,专门为高通量测序数据分析而生,了解链接:云生信服务器
9. 免费好课,个人能力提升,生信入门,不可错过,内容涵盖:生信语言与工具基础、R语言绘图、常见科研绘图、数据处理技能等,免费好课总入口:
10. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击进入组学大讲堂网校学习
- 发表于 2020-03-17 14:20
- 阅读 ( 8637 )
- 分类:基因组学
