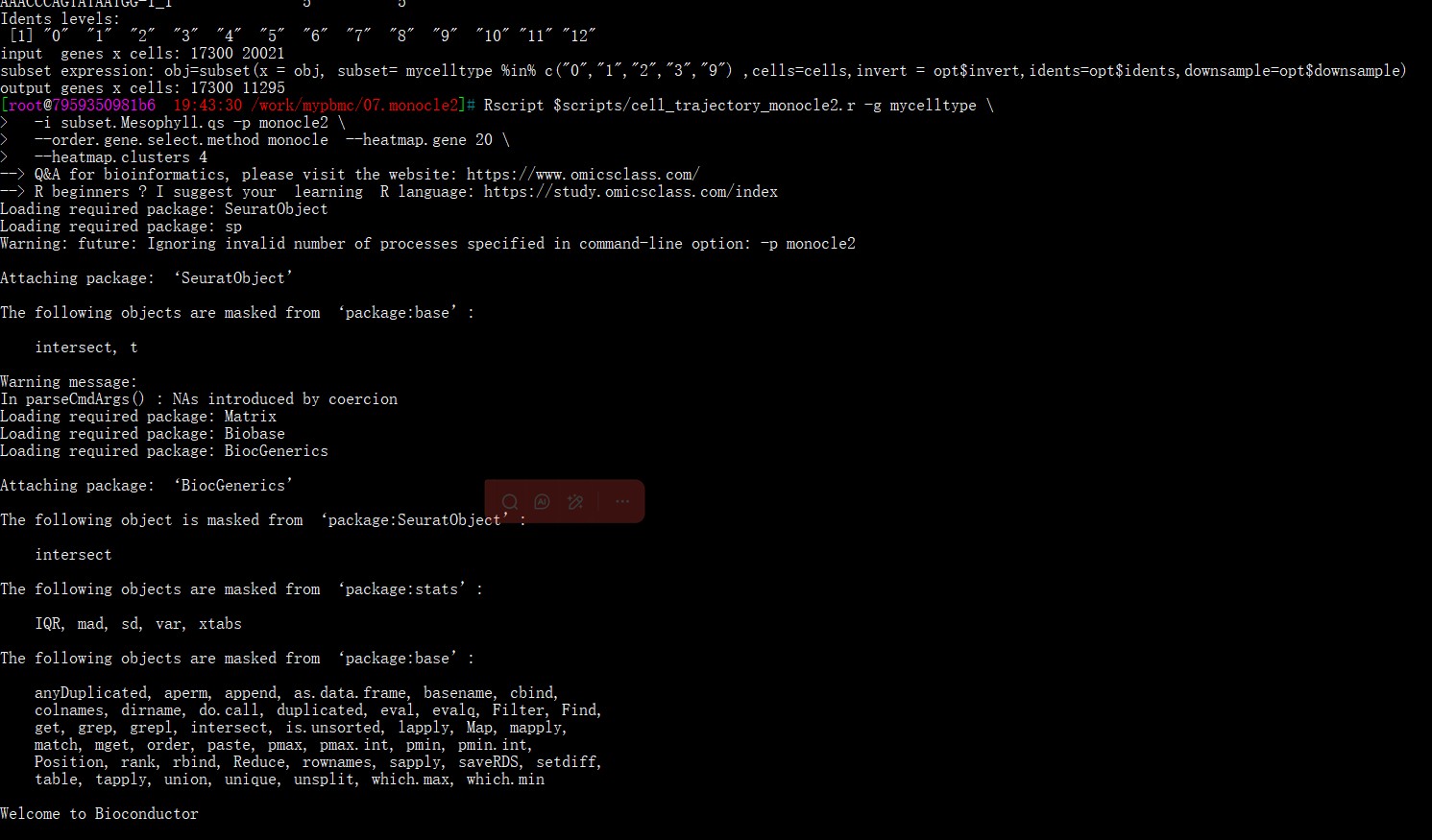
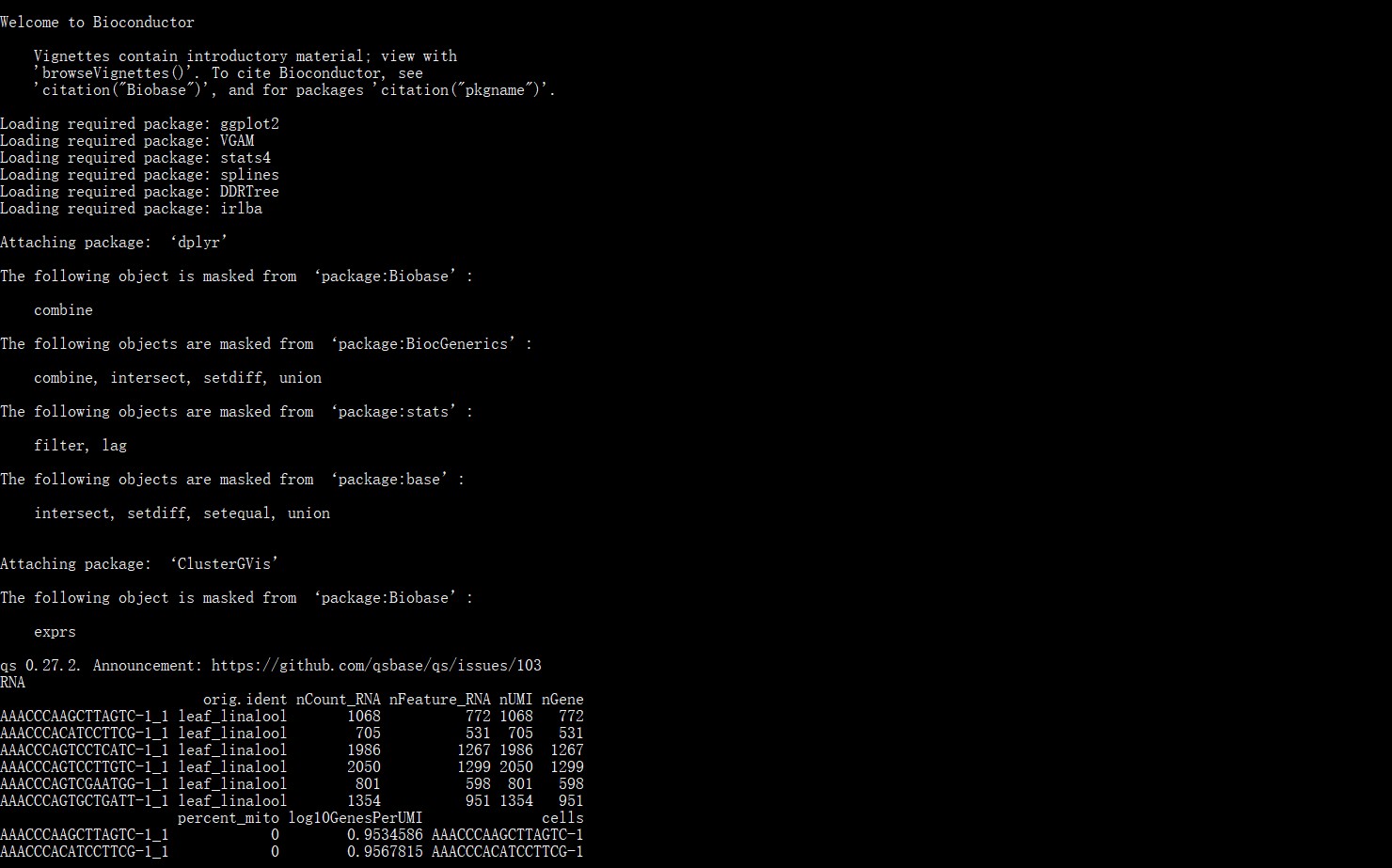
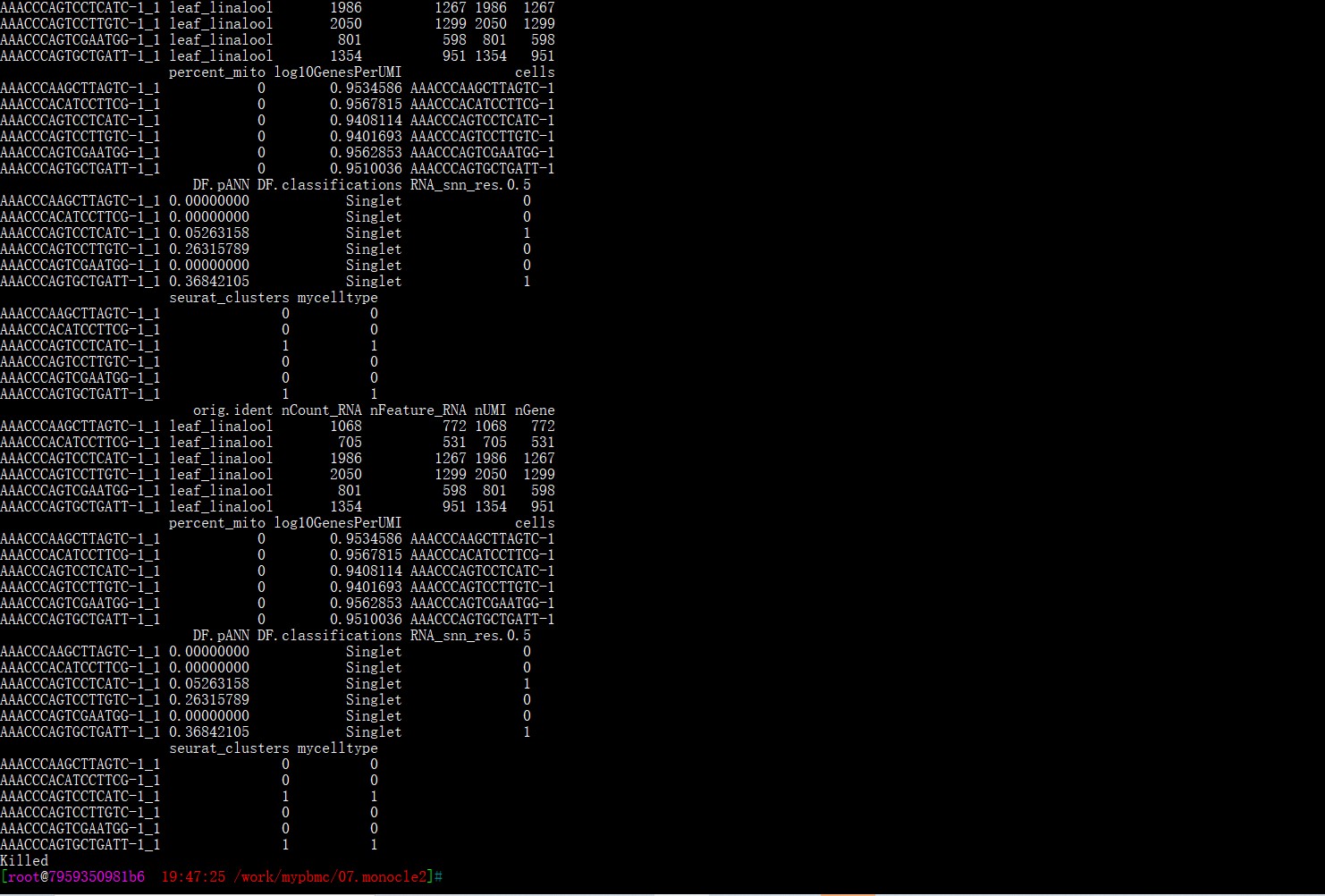
轨迹分析出现killed报错
1 个回答
- 1 关注
- 0 收藏,633 浏览
- HHHHHH 提出于 2025-12-02 19:54
相似问题
- 提供感兴趣基因进行分支分析时,出现Error in simple_vs_index(x, rlang::eval_tidy(args[[1]]), na_ok) : Unknown vertex selected报错 1 回答
- 老师,拟时序基础初步分析时,运行以下代码时Rscript $scripts/cell_trajectory_monocle3.r --rds ../04.cell_type_ann_leaf/leaf.added.celltype.qs \ --use.seurat.umap --groupby mycelltype -o monocle3.1 --cpu 6,出现图中报错,是因为聚类分群的时候resolution值太低了吗 2 回答
- 猪单细胞测序数据拟时序分析时已知细胞分化起源,怎么修改代码? 1 回答