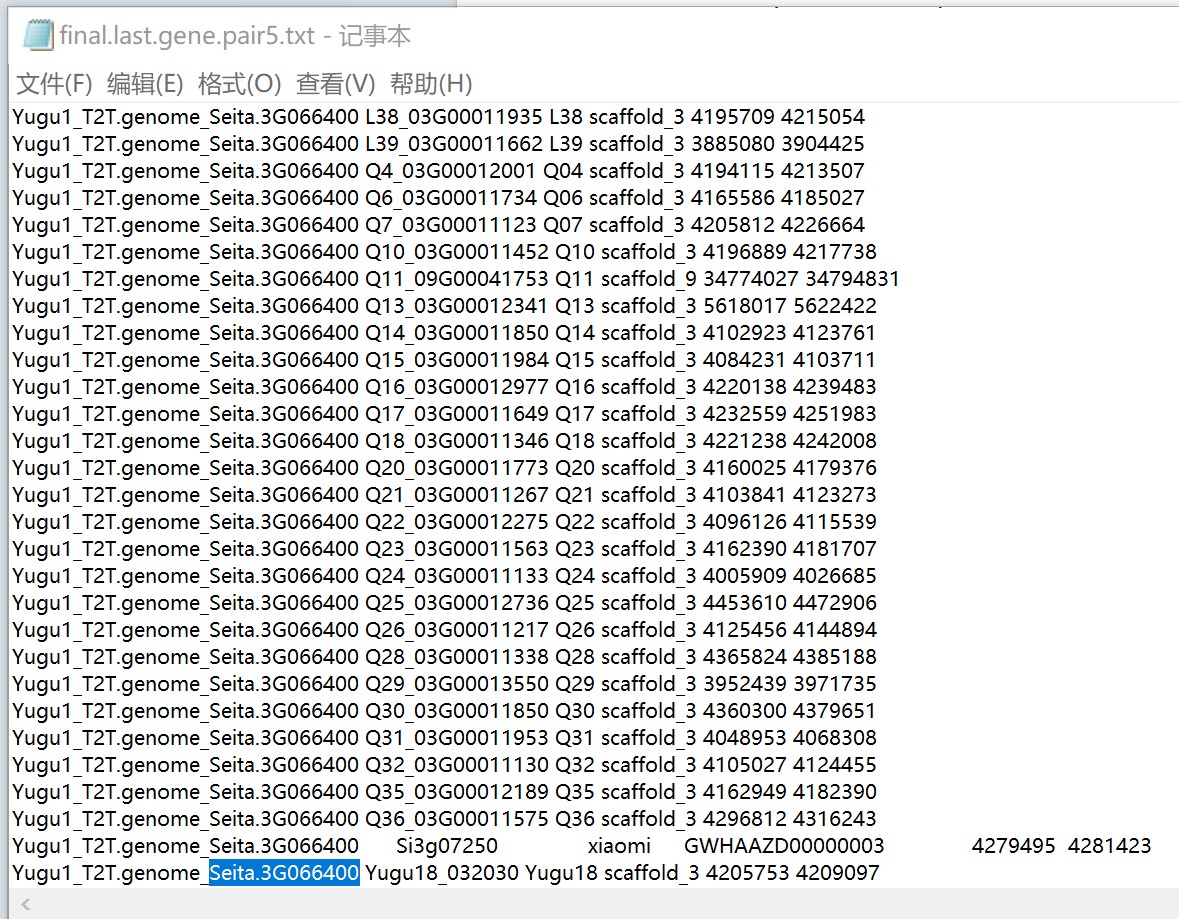
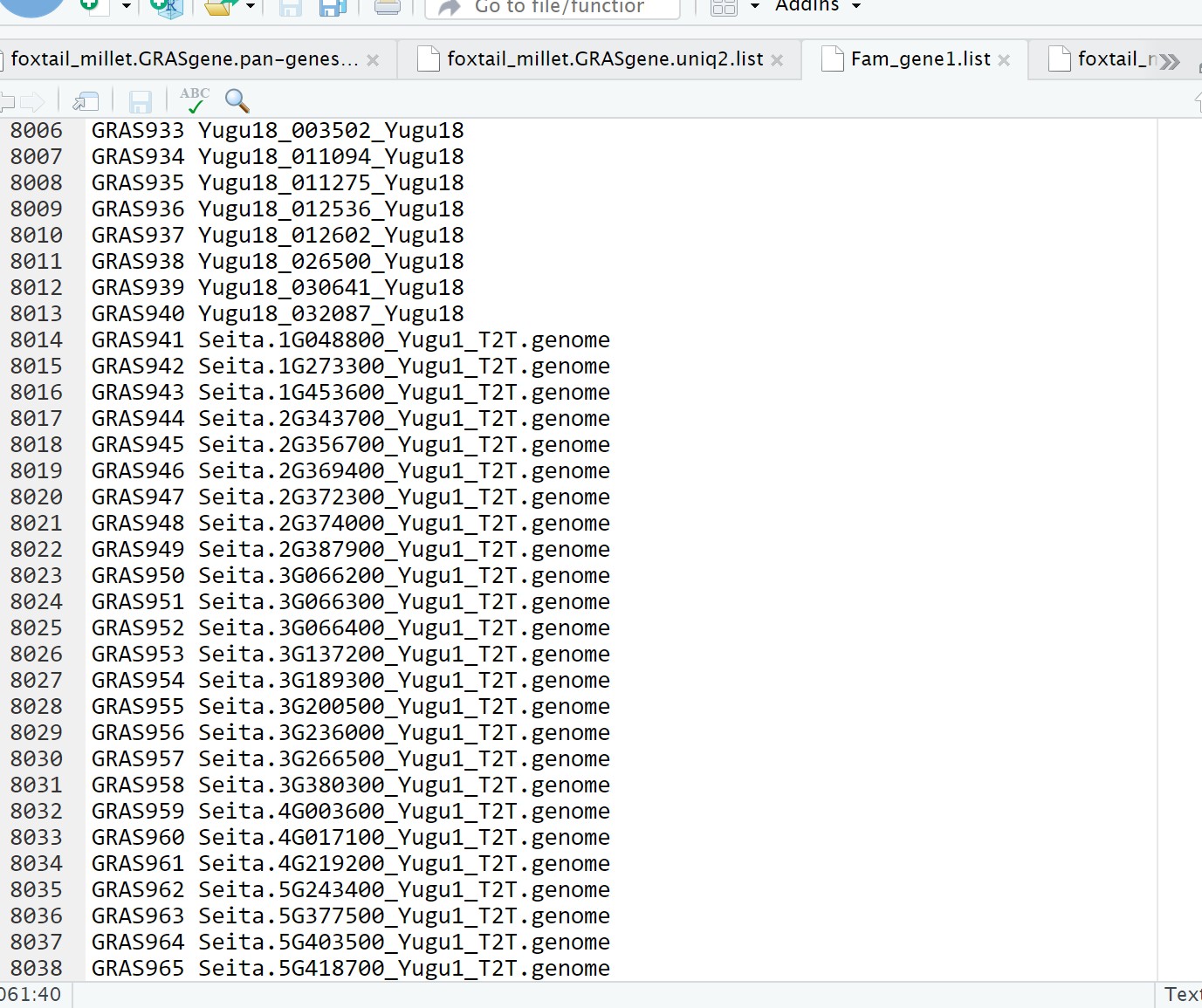
Seita.3G066400是属于GRAS家族基因,并且在结构变异与基因表达分析时,为最显著性差异表达基因,所以我需要以它做结构变异与基因结构和保守结构域分析,但Fam_gene.list文件里却没有鉴定到同源
老师,为什么我得到的Fam_gene.list中大多都是独有的,特别是参考基因组,全部都没有同源基因,那么我接下来结构变异与基因结果和保守结构域分析时,无法比对,该怎么往下做呢?
老师,为什么我得到的Fam_gene.list中大多都是独有的,特别是参考基因组,全部都没有同源基因,那么我接下来结构变异与基因结果和保守结构域分析时,无法比对,该怎么往下做呢?
另外,下面泛基因构建部分得到的列表final.last.gene.pair.txt,例如基因Seita.3G066400明明是有同源基因的,为什么最后得到Fam_gene.list却没有?我按照老师的指令awk -F"\t" '{if($0!~/^Fam_N/){for(i=2;i<=113;i++){print $1"\t"$i}}}' PAV_gene.list |sed "s/;$//"|awk -F"\t" '{gsub(";","\n"$1"\t",$0);print}'|awk -F"\t" '{if($2!=""){print}}' > Fam_gene.list得到的这个表,请问是不是哪里需要修改呢?
请先 登录 后评论
2 个回答
- 2 关注
- 0 收藏,1017 浏览
- Connie 提出于 2025-06-09 17:35
相似问题
- 泛基因家族分析,核心基因等数量统计 1 回答
- SV与家族基因的保守结构域分析 1 回答
- 泛基因家族分析之基因家族成员选择压力分析 1 回答
- 在PAV分析绘制热图的时候,运行R脚本出现如下错误,请问怎么解决?这个aplot安装包安装也一直安装不了 0 回答
- 泛基因家族分析数据准备里面,运行命令保留最长转录本结果都会报错,说是找不到文件,但文件存在在data目录下, ./.gff3.gz does not exist,Please verify the input file name/path 0 回答
- 客服说泛基因组分析中26Pan-genes.list对应泛基因集分析章节中的final.last.gene.pair.txt文件,但是两者之间有差异,像是缺了几列是怎么回事,如下面详述,请解答 1 回答