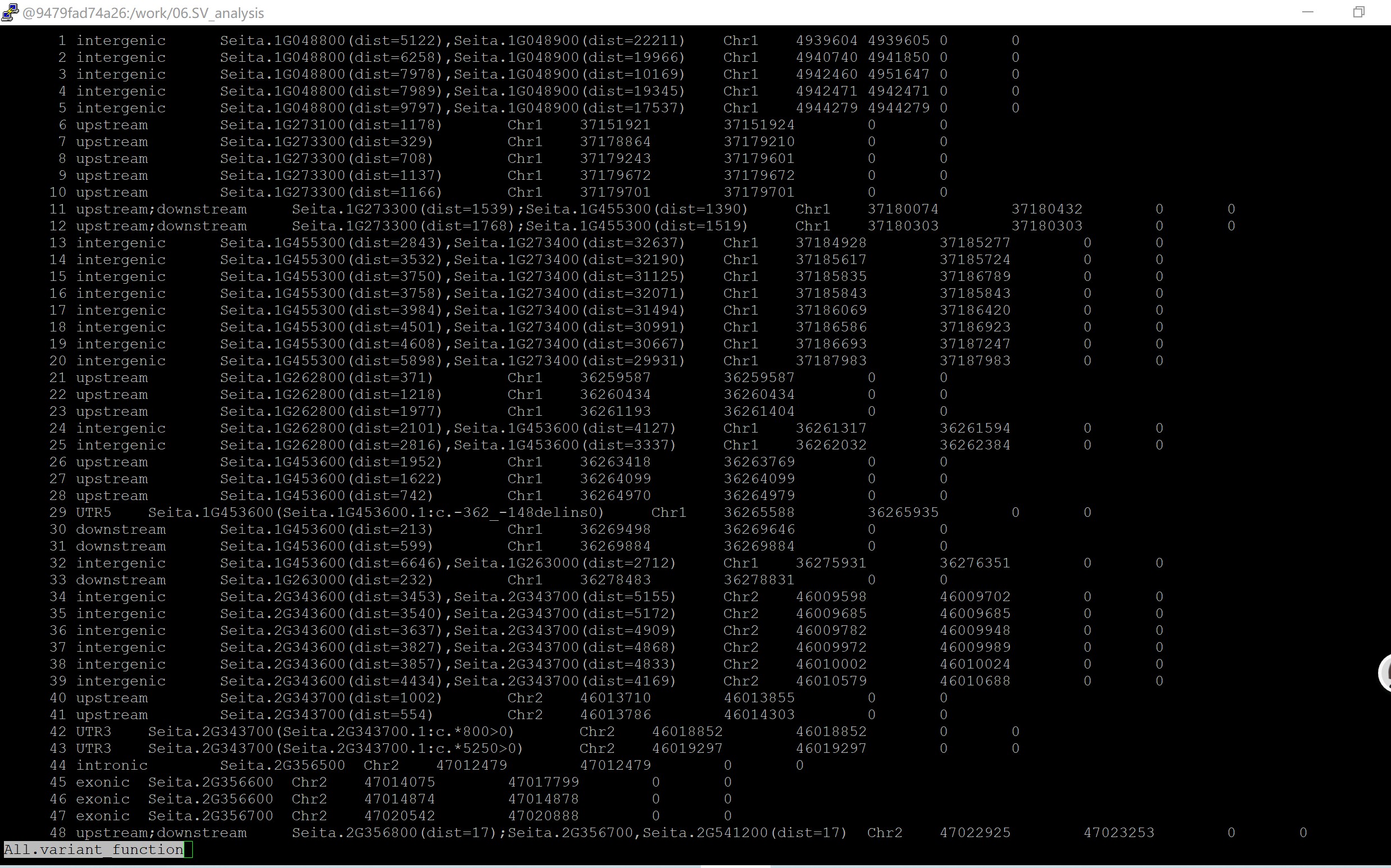
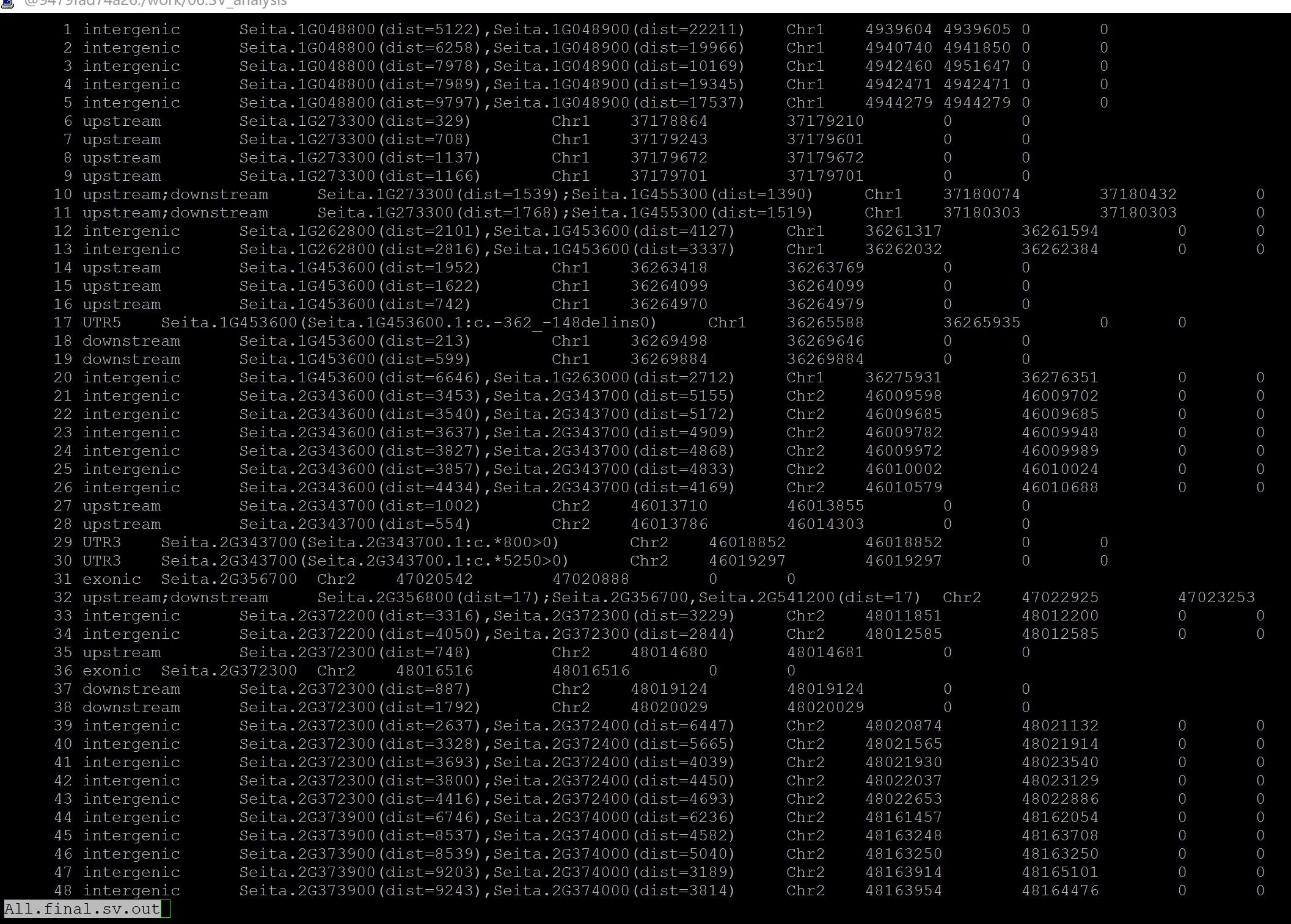
同学,我没有分析你的数据哈,具体情况我不是很清楚,你是自己跑的结构变异注释吗?如果你只用了一个参考基因组构建的注释信息库,你需要通过最后一步把对应家族编号的基因(在参考基因组中)提取出来。如果你是直接从作者那里获取到的结构变异注释结果,可能你需要问问作者相关的信息
从结构变异注释文件中提取WRKY基因结构变异信息
- 1 关注
- 0 收藏,1030 浏览
- Connie 提出于 2025-04-26 19:58
相似问题
- 泛基因家族分析,核心基因等数量统计 1 回答
- SV与家族基因的保守结构域分析 1 回答
- 泛基因家族分析之基因家族成员选择压力分析 1 回答
- 在PAV分析绘制热图的时候,运行R脚本出现如下错误,请问怎么解决?这个aplot安装包安装也一直安装不了 0 回答
- 泛基因家族分析数据准备里面,运行命令保留最长转录本结果都会报错,说是找不到文件,但文件存在在data目录下, ./.gff3.gz does not exist,Please verify the input file name/path 0 回答
- 客服说泛基因组分析中26Pan-genes.list对应泛基因集分析章节中的final.last.gene.pair.txt文件,但是两者之间有差异,像是缺了几列是怎么回事,如下面详述,请解答 1 回答