不好意思,第一次提问,下次一定写的详细来问您
我是这样运行的:
先创建了pep.fa文件
然后执行
[share] perl get_fa_by_id.pl AT.gff TAIR10.pep.all.fa pep.fa
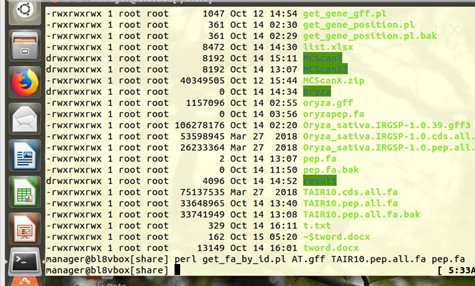
AT.gff是之前提取的基因位置信息,没有问题
运行完没有报错,但
pep.fa文件是0字节
cp
@omicsgene 老师,按照视频的脚本仔细核对没有错误,编译执行后也有没有报错,但pep.fa文件一直是空,文件名字没有空格,是不是文件读取权限向题
如果脚本有问题,编译起来就会报错,还得请教老师来解决,谢谢
use Bio::SeqIO;
use Bio::Seq;
my$in = Bio::SeqIO->new(-file => "$ARGV[1]" , -format => 'Fasta');
my$out = Bio::SeqIO->new(-file => "$ARGV[2]" , -format => 'Fasta');
my%keep=();
open IN, "$ARGV[0]" or die "$!";
while(<IN>)
{
chomp;
next if /^#/;
my@tmp=split(/\s+/);
$keep{"$tmp[1].1"}=1;
}
close(IN);
while (my $seq = $in->next_seq() )
{
my($id,$sequence,$desc)=($seq->id,$seq->seq,$seq->desc);
if(exists $keep{$id})
{
$out->write_seq($seq);
}
}
$in->close();
$out->close();
不好意思,第一次提问,下次一定写的详细来问您
我是这样运行的:
先创建了pep.fa文件
然后执行
[share] perl get_fa_by_id.pl AT.gff TAIR10.pep.all.fa pep.fa

AT.gff是之前提取的基因位置信息,没有问题
运行完没有报错,但
pep.fa文件是0字节