SNP不能转化成CAPS标记?来试试dCAPS吧!
之前在 如何将SNP标记转化成可跑胶验证的CAPS标记 文章中介绍了一种将SNP标记转换为CAPS标记的方法,今天,小编再给大家介绍一种应用范围更广的标记——dCAPS。
dCAPS简介
dCAPS听名字就知道和CAPS有很大的关系,它是由CAPS标记衍生的分子标记。dCAPS是通过引物引入错配的碱基,从而构建或者去除限制性内切酶识别位点的标记技术。
dCAPS的优势
CAPS仅限于突变位点能够产生或破坏限制酶识别位点的情况,而dCAPS没有此限制,几乎所有SNP都可转化为dCAPS标记。此外,CAPS的优势dCAPS同样具有。
dCAPS的工作原理
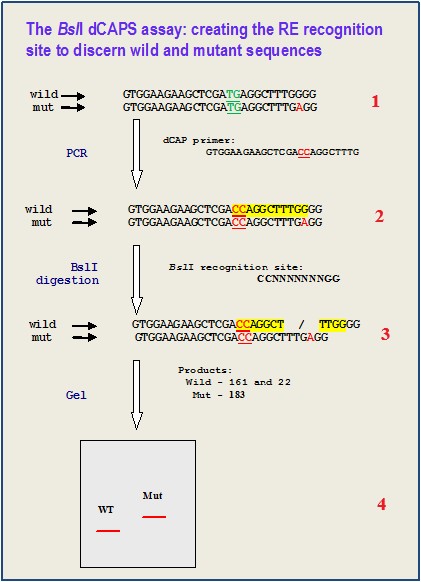
如图所示,1 中的两条序列分别是野生型与突变型个体的DNA序列,倒数第3位碱基为SNP位点,该位点不在限制性内切酶识别位点上。但若将TG(1 中绿色字体)改为CC,则该SNP位点将位于 BslI 限制性内切酶识别位点上。因此,通过引物引入错配的碱基CC,将构建限制性内切酶识别位点,酶切后通过琼脂糖或丙烯酰胺凝胶电泳分离时,由RE切割的PCR产物将容易区分出不同的模式。
dCAPS引物设计
dCAPS引物设计的软件较少,大多使用的都是网页版的 dCAPS Finder 2.0,少数引物的设计可以使用该软件,地址:http://helix.wustl.edu/dcaps/dcaps.html
使用方法:
分别输入两种类型的序列。需注意的是,除SNP外,两条序列是相同的;SNP最好位于序列中间,两边各有25bp左右的碱基;序列总长度不能超过60bp;并且只允许出现A、C、G、T四种字符。
第三个输入框中输入允许的错配碱基数,如果是0,则寻找的是CAPS标记。
提交运行,可能会花一点时间,耐心等候。
dCAPS Finder 2.0只是设计了一端的引物,另一端需要另外设计,使用primer 3.0或5.0都可以实现,这个属于常用技能了,在此就不额外介绍了。
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2018-05-30 13:41
- 阅读 ( 23168 )
- 分类:其他
