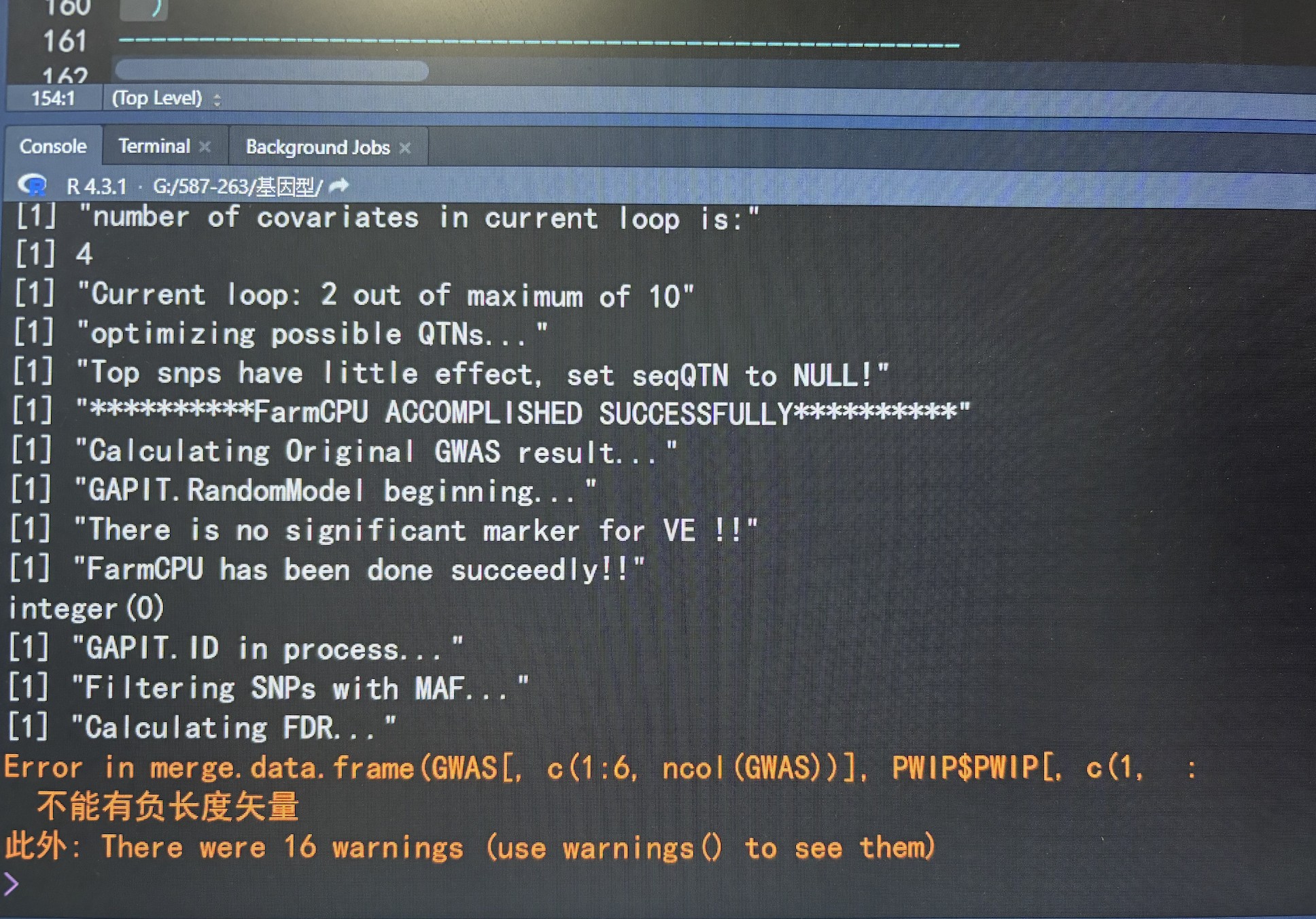
同学,你贴一下你的基因型文件和表型文件看看格式是不是有问题
- 1 关注
- 0 收藏,1770 浏览
- wangshengbnag 提出于 2024-08-26 10:02
相似问题
- 在用GEMMA软件进行GWAS分析的时候,需要加入Q矩阵,有一个疑问 1 回答
- 请问我在做性状绘图时出现报错,我的数据格式应如何修改 1 回答
- LDBlockShow -Cutline 7 -InVCF $vcf -OutPut LD.p \ -Region scaffoldC01:46993000-46994000 -OutPng -SeleVar 2 -InGWAS ld.pvalue.txt这条代码如何修改能使中间的字体大一点 1 回答
- tassel导入表型数据和Q文件,一直报错格式有误 0 回答
- tassel导入表型数据和Q文件,一直报错格式有误 0 回答
- 全基因组关联分析GWAS教程中的beagle填充问题 1 回答