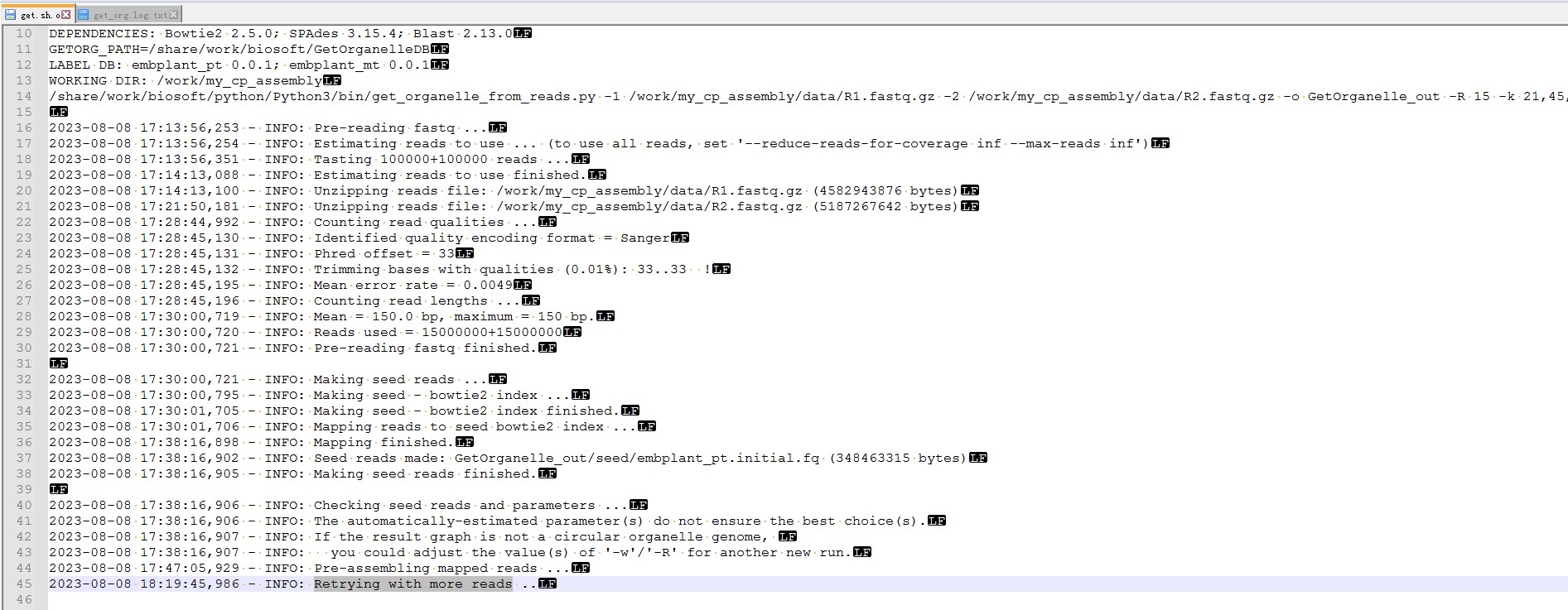
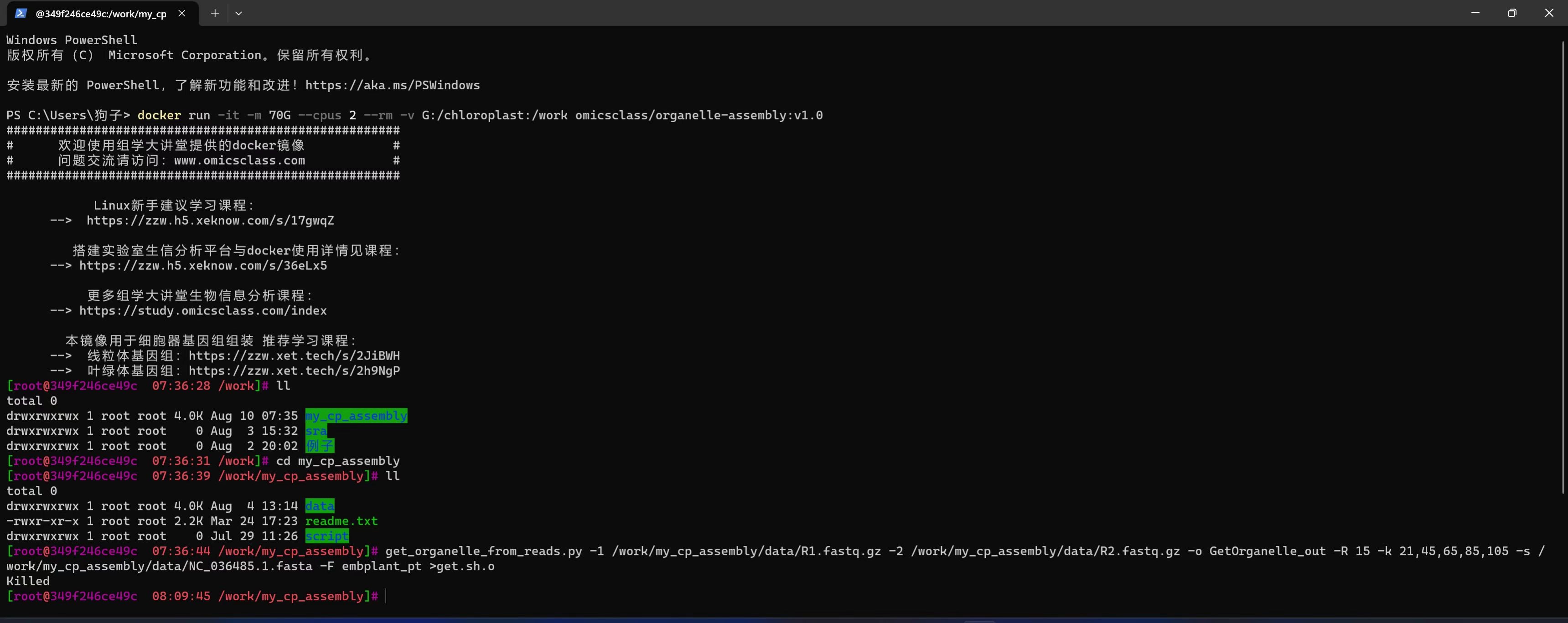
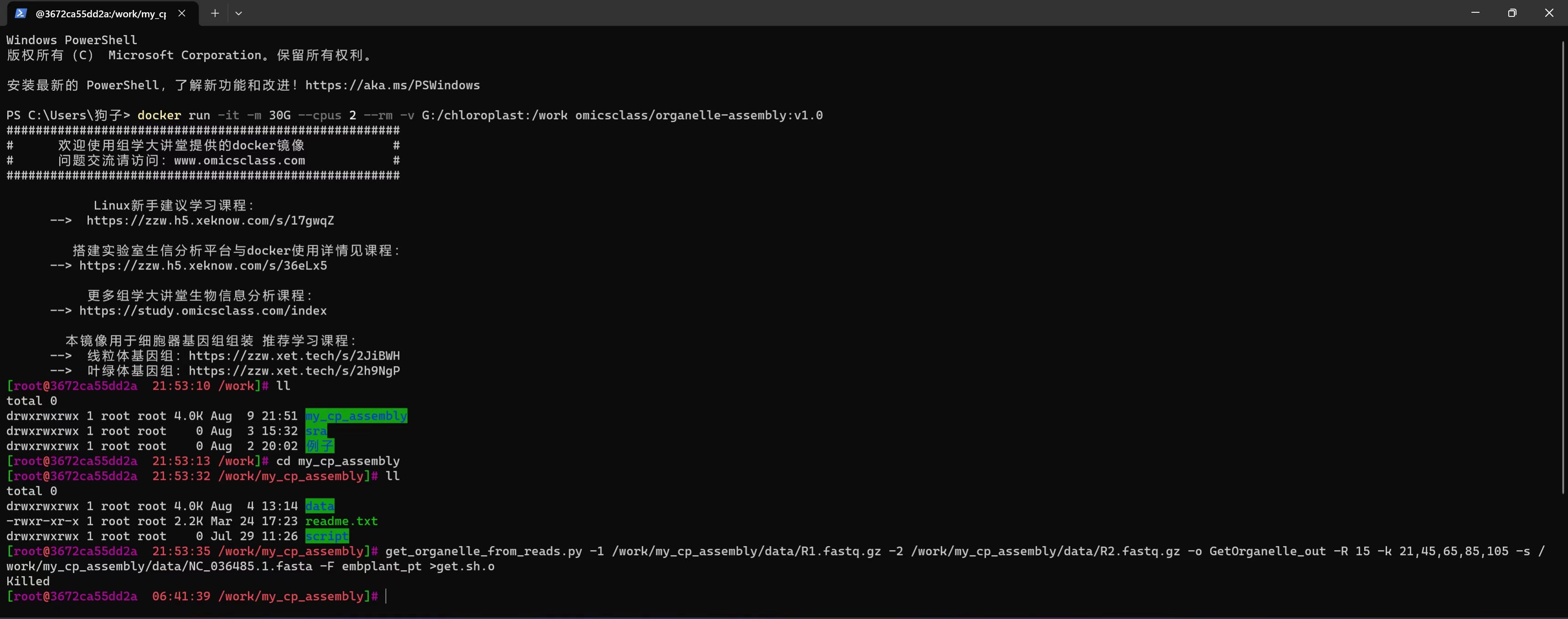
get_organelle_from_reads.py -1 /work/my_cp_assembly/data/R1.fastq.gz -2 /work/my_cp_assembly/data/R2.fastq.gz -o GetOrganelle_out -R 15 -k 21,45,65,85,105 -s /work/my_cp_assembly/data/NC_036485.1.fasta -F embplant_pt 跑了,后面出现“killed”就终止了。其它没有任何信息。
叶绿体基因组GetOrganelle组装时出现killed,内存从10G改为30G又改成70G,但是还是不行,程序终止。
3 个回答
- 2 关注
- 0 收藏,2959 浏览
- 蛋卷儿〜 提出于 2023-08-09 10:18