你好,我看了下你的文件,layout确实有问题,具体修改方法见下图:
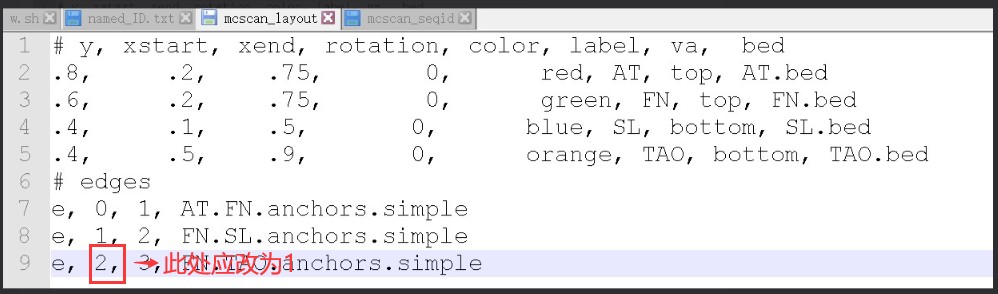
这里的0 1 2 3 可以理解为物种,从 0 开始,按照你的顺序依次编号,如下图:
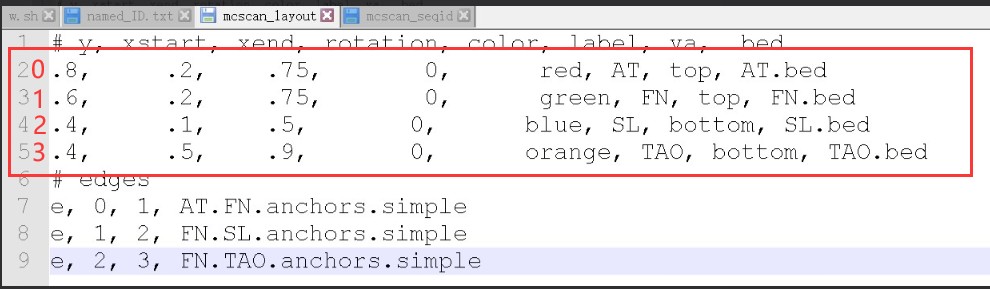
要画 FN 与 TAO的共线性,就要指定连线是从1到3,对应的两物种共线性文件就是FN.TAO.anchors.simple。
老师您好,之前我问过出现找不到基因的原因,您让我简化一下ID,https://www.omicsclass.com/question/5477。我简化后还是出现如下报错:
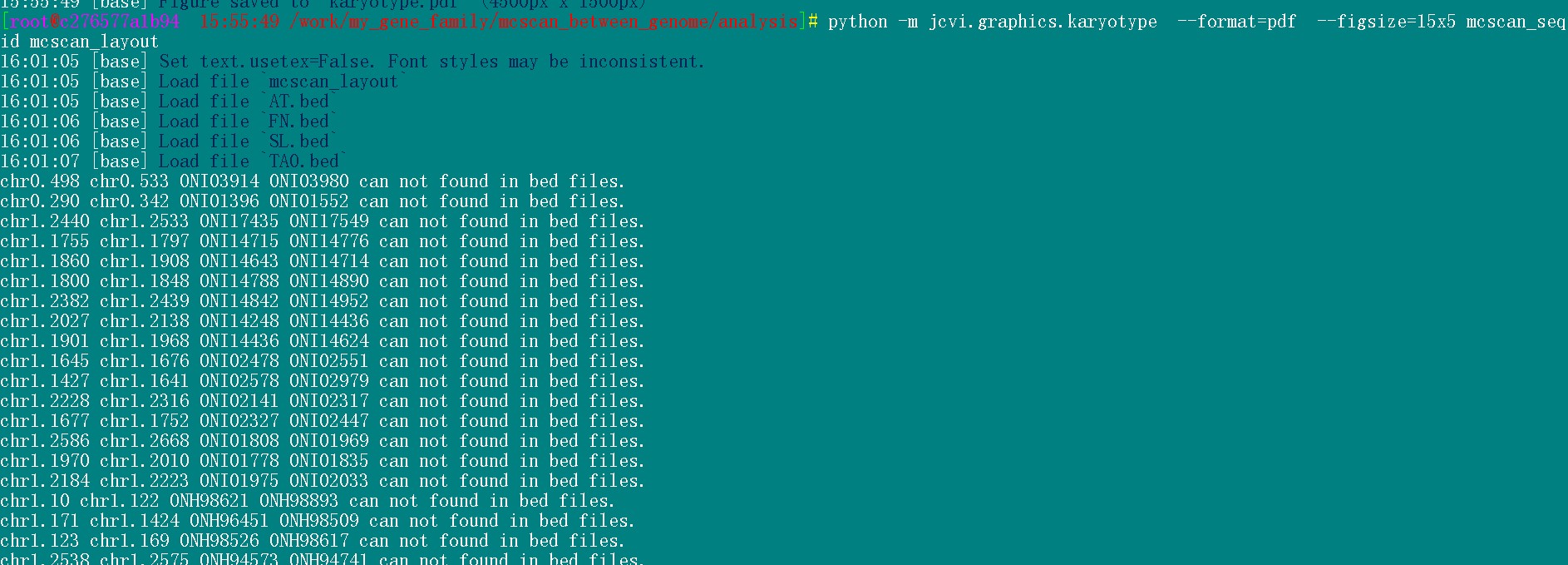
后来我先画了2个物种,可以出来图,再画了3个物种,也可以出来,,增加到4个物种时就又开始出现类似报错,,所以我想原因不是我的ID,是我的mcscan_layout文件与mcscan_seqid文件,,我想问下画更多物种时(4种),我是想画成您课程里介绍的那种5个物种甚至更多。这2个文件到底该如何整理。我看官方文档也很简洁 介绍了3个物种,可是我想画更多应该怎么处理呢,,以下是我画图时使用报错的mcscan_layout与mcscan_seqid


谢谢老师解答!