j结构域 domain 就是motif, 按理来说 这些家族成员都有相同的domain,都会找到motif;
你把 以下参数调整一下 可以把motif 最大长度调成200 试试,有可能找到更多motif
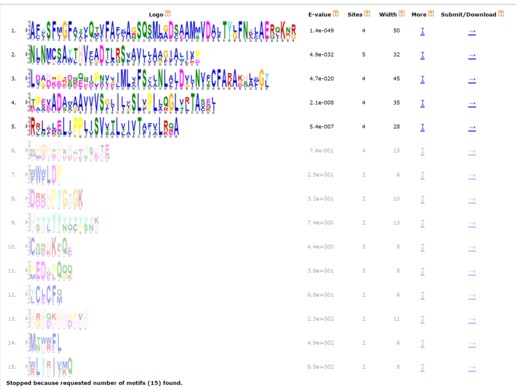
想请教老师一下。我搜出来的motif中,第6个开始,E值0.74,已经高于0.05了。这是不是意味着这些motif(6-15的)在我的基因家族里不存在?
所以我需要调整参数,将motif设置成5才对。是吧。
我在分析时可以说:根据预测结果,结合E值,我的基因家族中,有5个motif。
******************************************
我选择motif个数5重做了一次。结果基因家族中,有部分序列没有motif。我想这也可以理解,毕竟这些蛋白质序列与陆地上的植物相比,一致性都不高,20-30%左右。所以才没有搜到motif。这样解释合理吧:)
***********************************
没有motif不代表没有保守的domain,所以我又做了Pfam的保守domain分析。然后所有序列都能检测到保守domain。所以就解释得通了。没有motif,不代表没有domain。
请老师帮忙看看。
我觉得这么分析下来是合理的。