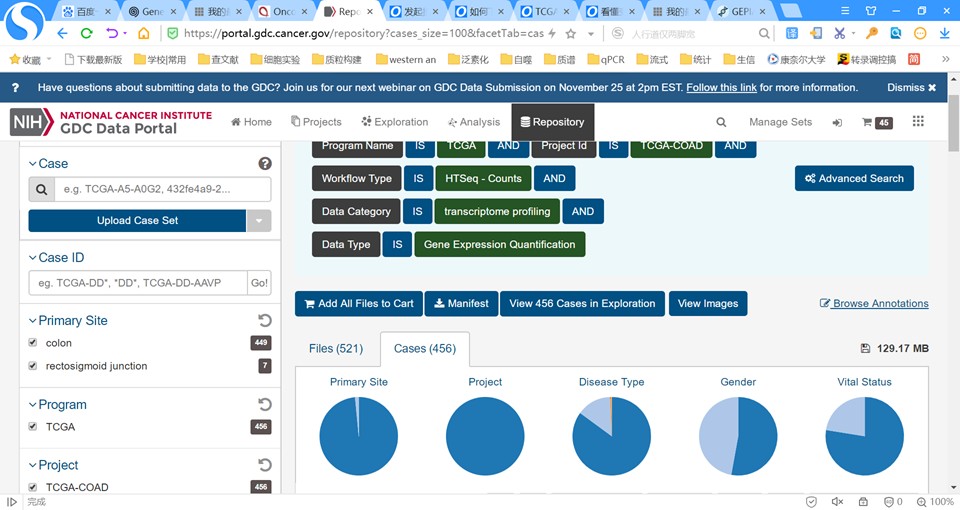
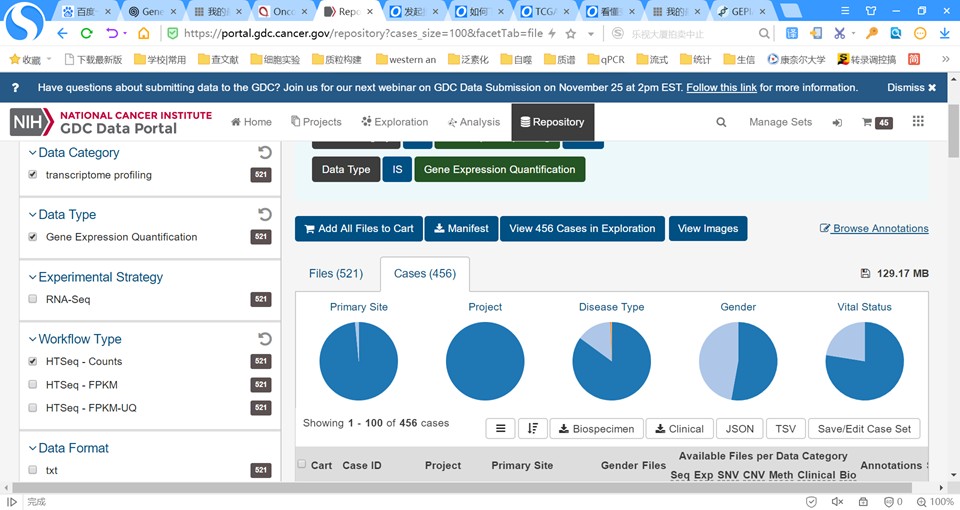
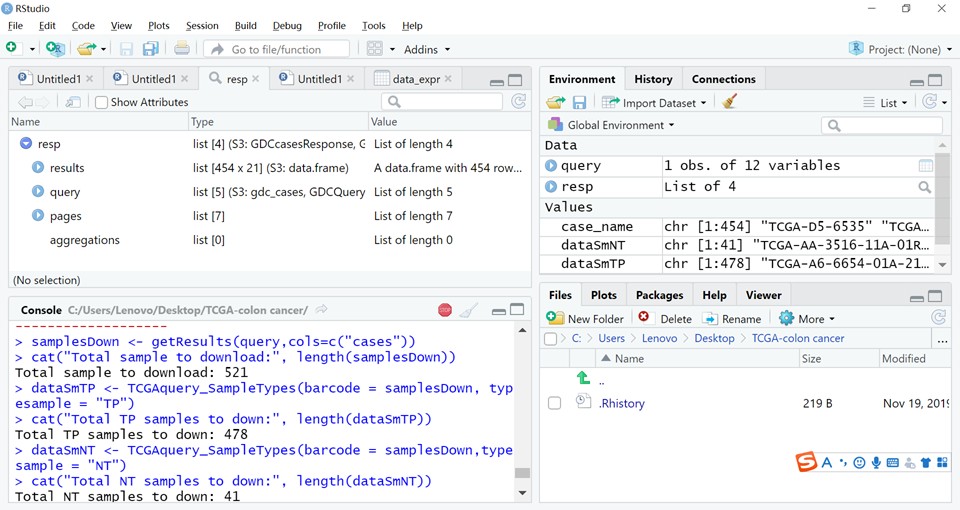
case是癌症病人编号,有时候一个癌症病人取样时同时取了多个组织(例如原发癌组织,转移癌组织,血液对照等),后期做实验一个case 多个组织都可能有实验数据,因此会存在文件数多于case数的情况,属于正常。
TCGA数据库下载数量不对
2 个回答
- 2 关注
- 0 收藏,6982 浏览
- jing 提出于 2019-11-19 14:43
相似问题
- R语言下载GSE数据一直在报错 0 回答
- 老师,下载数据是出现图片这个错误,我该怎么办? 1 回答
- 求助:用Rstudio下载数据,出现remaining后不动了,是不能下载了吗? 2 回答
- SRA数据下载 1 回答
- 高通量下载数据格式 1 回答
- 请问在运行利用exprs()获取表达矩阵 exprSet = exprs(gset)时,报错Error in (function (classes, fdef, mtable) : unable to find an inherited method for function ‘exprs’ for signature ‘"factor"’不知道什么原因? 1 回答