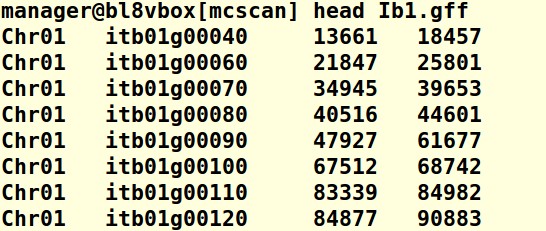
遇到这个问题,应该首先检查文件路径是否写错,文件格式总染色体基因ID是否统一;
如果以上检查都没有问题:可能是windows和linux系统编码的问题导致的,在linux中换行符一般是\n 而在windows中是\r
因此为了统一,应该统一编码UTF-8 千万不要用记事本:https://www.omicsclass.com/article/395
或者查看一下,gff文件是不是用tab分开的列,注意空白的地方不要有空格;
命令如下:
sudo java family_circle_plotter -g /home/manager/ERFfamily/MCScanX1/mcscan/Ib.gff -s /home/manager/ERFfamily/MCScanX1/mcscan/Ib1.collinearity -c /home/manager/ERFfamily/MCScanX1/family.ctl -f /home/manager/ERFfamily/MCScanX1/AP2_family.txt -o /home/manager/ERFfamily/MCScanX1/AP2.PNG
报错截图:

输入文件截图:




路径检查:

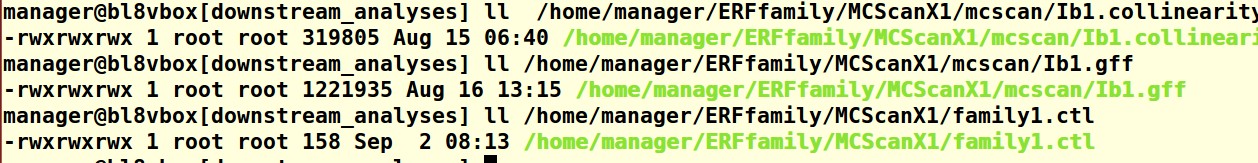
遇到这个问题,应该首先检查文件路径是否写错,文件格式总染色体基因ID是否统一;
如果以上检查都没有问题:可能是windows和linux系统编码的问题导致的,在linux中换行符一般是\n 而在windows中是\r
因此为了统一,应该统一编码UTF-8 千万不要用记事本:https://www.omicsclass.com/article/395
或者查看一下,gff文件是不是用tab分开的列,注意空白的地方不要有空格;
如果觉得我的回答对您有用,请随意打赏。你的支持将鼓励我继续创作!
