5 蛋白序列ID和cds序列ID不一致
回答问题即可获得 10 经验值,回答被采纳后即可获得 10 金币。
老师,我做基因家族分析
下面是蛋白ID
 下面是cds序列
下面是cds序列
 下面是gff文件
下面是gff文件
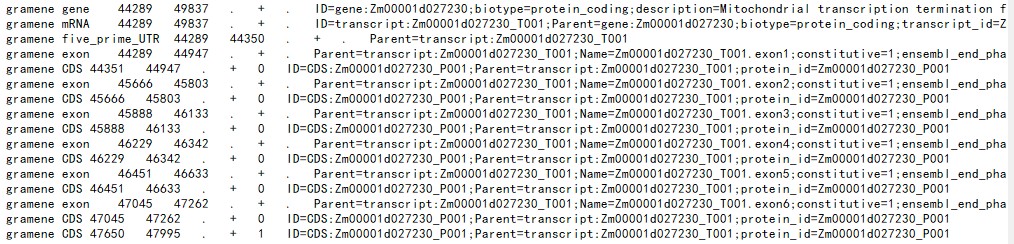 获取基因与mRNA的对应关系perl script/mRNAid_to_geneid.pl Arabidopsis_thaliana.TAIR10.41.gff3 mRNA2geneID.txt,提取到的ID=Zm00001d027240_T001,而在hmm搜索的结果ID=Zm00001d027240_P001,在去除重复的hmmer搜索的转录本ID时,ID不一致,同时hmm搜索的结果ID和cds序列里ID也不一致,无法提取cds序列
获取基因与mRNA的对应关系perl script/mRNAid_to_geneid.pl Arabidopsis_thaliana.TAIR10.41.gff3 mRNA2geneID.txt,提取到的ID=Zm00001d027240_T001,而在hmm搜索的结果ID=Zm00001d027240_P001,在去除重复的hmmer搜索的转录本ID时,ID不一致,同时hmm搜索的结果ID和cds序列里ID也不一致,无法提取cds序列
请先 登录 后评论
3 个回答
- 3 关注
- 0 收藏,9738 浏览
- 追梦 提出于 2019-03-20 14:12
相似问题
- 搜集的gff3文件;第一列不同,有的是chr,有的是id;这个影响吗? 2 回答
- 老师,我在做饭基因家族分析,我搜集的基因组的gff3文件,第一列有的是chr有的是基因id,这个会影响后续结果嘛? 2 回答
- 老师,为什么我的cds和pep文件导不出来呀?导出来报错,并且文件内容无。最后一张照片时我退出重进后的历史命令 2 回答
- 基因家族分析2.0,做基因结构分析时报错,完全按照提供指令做的 1 回答
- 老师,我这个是桑树的fasta和gff文件,不是从官网下载的。假如各个网站都找不到我想要的物种的基因组文件,只有手头这个文件,我要怎么做才能和教程的代码连接上呀?或者说碰到这种目标物种查不到的情况可以怎么做呢? 1 回答
- 老师,下面是报错、染色体信息、gff文件中的染色体和命令行 2 回答

