宏基因组结果解读和个性化分析课程上架
宏基因组测序技术是进行微生物资源开发利用、加速微生态科研深度研究的重要手段。随着高通量测序技术的普及,越来越多的研究人员开始着手与宏基因组分析。
但是,有相当一部分研究人员由于是初次接触宏基因组分析,对分析结果缺乏系统的了解,无法高效的挖掘和研究目的相关的结果,进而影响了文章的发表以及成果的转化。
课程内容
第一节:宏基因组基础知识
- 课程目标:快速掌握微生物群落结构、功能及生态学意义,理解物种信息表和功能信息表的核心内容
- 内容包括:
- 微生物群落结构与功能的基本概念:什么是宏基因组学?物种丰度表与功能信息表的作用
- 生态学意义:微生物群落与环境、宿主间的关系(如肠道菌群与健康)
- 数据表解读:物种丰度表(OTU/ASV表)结构、功能信息表(GO、KEGG)的组成与应用
第二节:Venn和UpSet图
- 课程目标:掌握绘制Venn图和UpSet图,展示物种或功能的分布与重叠
- 内容包括:
- Venn图与UpSet图的用途:展示物种或功能在不同样本间的重叠与独特性,突出样本间共有与独特元素的分布模式
- 使用R(ggVennDiagram、eulerr、UpSetR包)绘制不同特点的Venn图和UpSet图
§ggVennDiagram:绘制多组的Venn图,支持自定义颜色、标签和交集标注§eulerr:生成精确的Venn图,优化重叠区域的视觉表现,适合复杂数据集§UpSetR:绘制UpSet图,展示多组交集的矩阵式分布,突出高维数数的关系
第三节:物种组成堆叠柱状图
- 课程目标:掌握绘制堆叠柱状图,展示物种丰度或功能分布
- 内容包括:
- 堆叠柱状图:使用ggplot2绘制物种丰度分布,设置颜色分组和图例,展示不同样本或组别的丰度模式
- 使用ggalluvial绘制流动图:展示物种或功能在不同样本间的流动与分布,如何在图表中加入分组横条,突出动态变化
- 绘图优化:通过修改因子(factor)调整图例中物种或功能的顺序,优化视觉呈现和解读
第四节:物种丰度比较热图
- 课程目标:掌握绘制热图,展示物种或功能的多维分布与模式
- 内容包括:
- 热图:使用pheatmap包绘制物种或功能的丰度矩阵,展示多维分布与模式
- 多种热图类型:学习绘制层次聚类热图(基于距离和聚类方法)、非层次聚类热图(按自定义顺序)和双向聚类热图(同时聚类行和列)
- 桑基图与热图结合:使用R 和相关包(如networkD3、pheatmap)绘制桑基图与热图相结合的图表,展示物种或功能在不同样本间的流动与分布模式

第五节:Alpha多样性箱图
- 课程目标:掌握绘制多种类型的Alpha多样性图,分析微生物群落多样性特征
- 内容包括:
- 多种类型的Alpha多样性图:使用ggplot2绘制以下图表,展示群落内多样性,突出不同样本或组别的多样性差异:
§普通箱图:绘制Shannon、Simpson指数的箱线图,展示中位数、四分位距和离群值 §提琴图:绘制提琴图,展示数据分布密度 §缺口图(Notched Boxplot):绘制带缺口的箱图,比较不同组别中位数的显著性差异 §箱图和提琴图结合的图:综合展示分布和统计信息 §半箱图和半提琴图:绘制半箱图(仅显示一半箱线)和半提琴图(仅显示一半密度分布),优化空间利用和对比效果
- 统计检验: 学习t检验、Wilcoxon秩和检验、Kruskal-Wallis检验,用于比较不同组别或样本的Alpha多样性指数(如Shannon、Simpson、Chao1、ACE指数)差异,结合P值校正(如FDR)确保结果可靠性
第六节:Beta多样性散点图和箱图
- 课程目标:掌握绘制Beta多样性散点图和箱图,展示微生物群落间差异
- 内容包括:
- Beta多样性散点图和箱图:使用ggplot2和phyloseq绘制PCoA/NMDS散点图和箱图,展示不同样本或组别的群落间距离与差异
- 多种优化类型:
§PCoA/NMDS散点图:绘制带颜色编码的散点图,突出样本分组(按环境或处理分组),添加置信椭圆(confidence ellipses)或凸包(convex hulls)增强可视化 §Beta多样性箱图:绘制基于距离矩阵(如Bray-Curtis、Jaccard)的箱图,展示群落间差异的中位数和分布范围 §散点图与箱图,山脊图结合:展示样本分布的密度轮廓,提升美观性和信息密度
- 假设检验:学习PERMANOVA(Adonis)检验、ANOSIM检验用于比较不同组别或样本的Beta多样性距离(如Bray-Curtis、Jaccard距离)差异,结合P值校正(如FDR)评估群落结构显著性差异
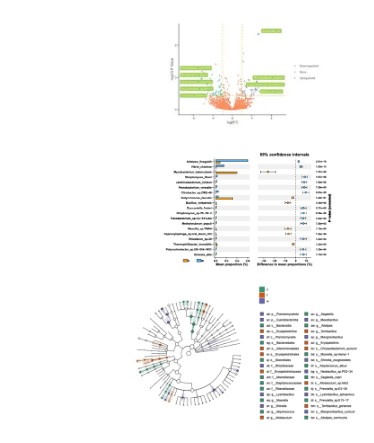
第七节:差异分析(火山图、LEfSe柱状图和误差条图)
- 课程目标:掌握差异分析的可视化,识别差异微生物或功能
- 内容包括:
- 差异分析:绘制火山图(ggplot2)、LefSe分支进化图(R包)和误差条图,识别显著差异的物种或功能
- 具体类型与优化:
§火山图:使用ggplot2绘制火山图,展示差异微生物的log2折叠变化(log2FC)与P值或调整后的P值(-log10P),标注显著性阈值(如log2FC > 1,adj.P < 0.05),突出上调和下调的物种 §LEfSe分支进化图:使用LEfSe R包绘制图, 标识在不同组别中显著富集的微生物或功能 §误差条图:绘制误差条图(errorbar),展示不同数据拼合后的物种或功能的平均丰度及其标准误差(SEM)或置信区间(CI),突出不同组别间的差异,呈现拼合数据的变化
§统计检验:学习t检验、Wilcoxon秩和检验、Kruskal-Wallis检验,用于比较组间微生物或功能的丰度差异;使用FDR(False Discovery Rate)或Bonferroni校正控制多重检验错误
第八节:基因差异和富集分析
- 课程目标:掌握基因水平差异分析与功能富集的可视化
- 内容包括:
- 基因差异分析:识别基因水平的显著差异,绘制相关图表,展示差异特征:
§火山图:使用ggplot2绘制基因的log2FC与P值或调整后的P值(-log10P)突出上调和下调基因。 §柱状图:绘制显著差异基因的丰度或表达量变化,优化颜色分组和排序,突出关键基因。
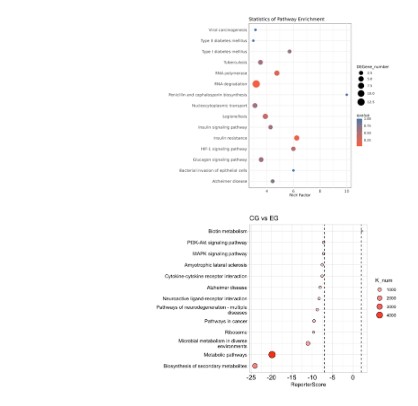
- 功能富集分析:使用KEGG/GO数据库进行富集分析,绘制富集分析图,展示功能分布:
§富集分析图:使用ggplot2或clusterProfiler R包绘制KEGG富集泡泡图,展示富集的代谢通路。
- 统计检验: t检验、Wilcoxon秩和检验用于基因差异分析;使用Fisher精确检验或超几何分布检验进行功能富集分析,结合P值校正(如FDR)评估显著性
第九节:环境因子分析
- 课程目标:掌握环境因子与微生物关系的可视化,探索相关性
- 内容包括:
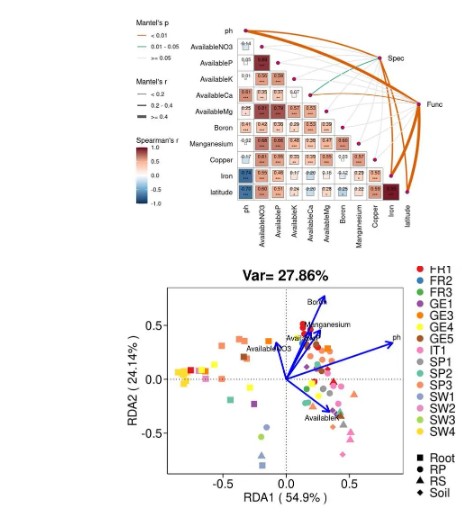
- 环境因子分析:结合环境变量(如pH、温度、湿度),绘制相关性图表,展示微生物与环境间的关系:
§相关性散点图:使用ggplot2绘制物种丰度与环境变量(如pH)的散点图,添加拟合线(线性或非线性)和相关系数(Spearman/Pearson),突出正/负相关性
§相关性热图:使用ggplot2绘制环境变量与微生物丰度的相关性矩阵,优化颜色渐变和标签,展示多维相关模式。
第十节:相关性网络
- 课程目标:掌握绘制微生物或功能的共存网络,识别关键节点
- 内容包括:
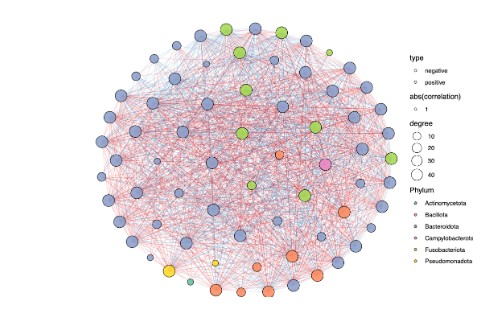
- 相关性网络:使用igraph构建共存网络,展示物种或功能间的关系:
§网络构建:使用igraph计算Spearman或Pearson相关系数,生成有向/无向网络,设置相关性阈值(如>0.6或<-0.6)筛选显著边
§网络可视化:绘制节点(物种或功能)和边(相关性)的网络图,优化节点大小(按度数或丰度)、颜色(按分类或模块)和边粗细(按相关性强度)
§关键节点识别:使用igraph分析网络中心性(如度中心性、介数中心性),突出生态学重要性节点
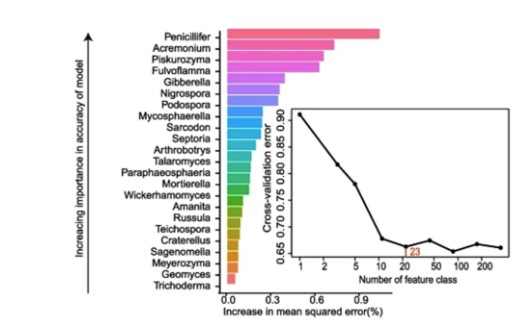
第十一节:随机森林(分类和回归)
- 课程目标:掌握使用随机森林进行微生物分类和回归分析的可视化
- 内容包括:
- 随机森林应用:使用R Markdown和randomForest包进行分类和回归分析,预测微生物与环境/功能的关系:
§分类分析:使用randomForest进行微生物群落分类(如健康/疾病状态),绘制混淆矩阵热图(confusion matrix)或分类精度图,评估模型性能
§回归分析:使用randomForest预测微生物丰度与环境变量(如pH、温度)的关系,绘制回归预测图或重要性排序图(variable importance plot)
§可视化优化:调整图表颜色、标签和图例,突出重要变量和预测结果,生成符合文献要求的专业可视化。
第十二节:肠型分析
- 课程目标:掌握肠型分析的可视化与应用,理解肠道微生物群落的典型模式
- 内容包括:
- 肠型分类:识别常见肠道微生物群落类型(如Enterotype 1、2、3),基于物种丰度表进行聚类分析:
§聚类方法:使用R Markdown和stats包(如k-means或PAM聚类)识别肠型,基于物种丰度表的主成分分析(PCA)或PCoA
§可视化:绘制PCA散点图或PCoA散点图,标注肠型分组(颜色编码),突出不同肠型的分布模式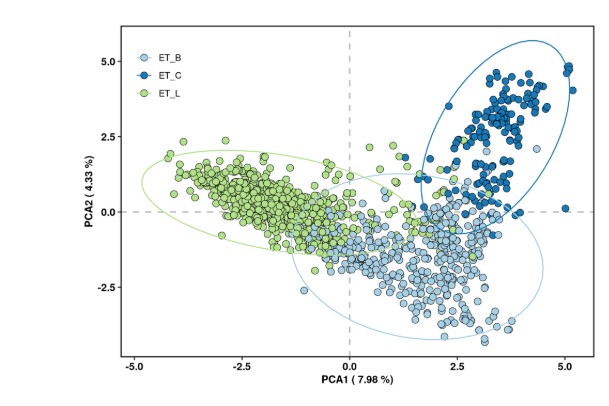
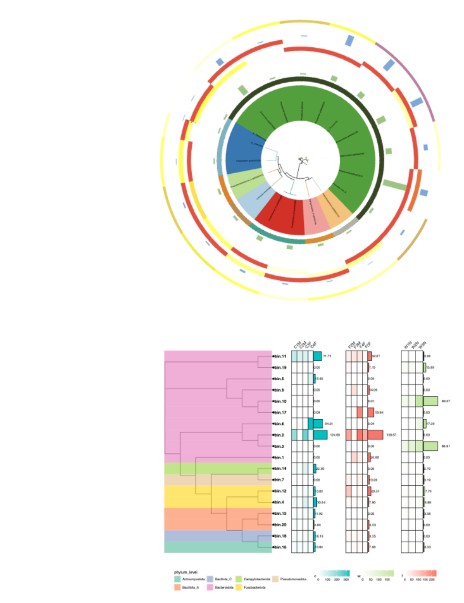
第十三节:分箱结果系统发育树绘制
- 课程目标:了解宏基因组数据的分箱目的和结果类型,并借助分箱结果绘制多种类型的系统发育树
- 内容包括:
- 进化树的解读和简单修改:了解进化树的一些基本知识,并对其做一些简单的修改:
§R包介导的进化树美化:对于矩形进化树,可以直接借助R包来进行美化
§高级可视化:对于一些复杂的圆形进化树,很难在R里直接得到想要的结果,所以我们借助R生成一些注释文件,并通过itol网站进行进化树的美化
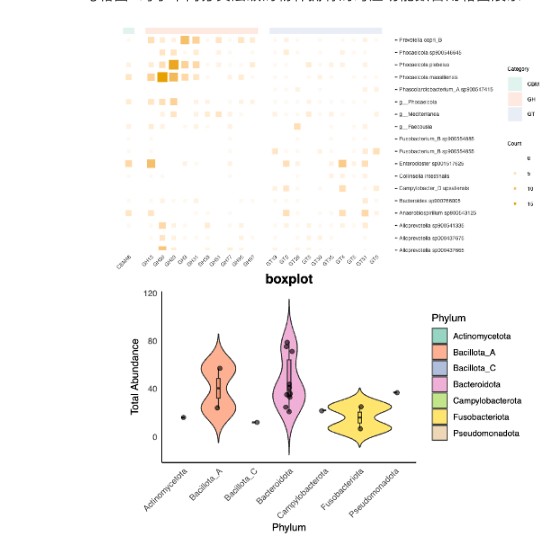
第十三节:分箱结果功能展示
- 课程目标:了解常见宏基因组数据分箱结果的展示类型
- 内容包括:
- 功能组成饼图:对功能的层级结构多采用旭日图进行展示
§热图: 对于不同mags的结果的功能丰度用热图展示
§箱图: 对于不同分类层级的物种拥有的对应功能数目用箱图展示
课程目录展示
课程目录展课程示

扫描二维码或以下链接观看

- 发表于 2025-06-30 11:01
- 阅读 ( 1694 )
- 分类:宏基因组
你可能感兴趣的文章
- 紧扣前沿热点--多种研究培训,总有你需要的! 6137 浏览
- 微生物与环境因子,代谢物,功能等相关性分析linkET 3466 浏览
- 宏病毒组分析一区 1741 浏览
相关问题
- 老师您好,我在运行megahit对200多个环境微生物宏基因组数据进行组装时,megahit报错Exit code -9 0 回答
- binning 想做功能注释,但还是不知道该用哪些文件进行基因预测和功能注释? 1 回答
- binning物种注释的时候,gtdbtk软件和数据包已经更新,旧版的匹配不上,请问应该怎么更新一下软件,谢谢 1 回答
- 想咨询一下,可否有contig的NR库的物种和功能注释代码和教程? 1 回答
- 想咨询一下,宏基因组功能注释的视频中,有一步是用cds.fa文件注释物种,想咨询一下,这个物种注释出来后,应该如何计算丰度?是根据前面cds.count来计算吗,这样计算出的物种丰度可靠吗?因为有些物种可能基因很多。 1 回答
- 请问,我弄了一个256线程,1t内存的服务器,想处理500G的宏基因组注释,大概需要跑多少天,全套下来 1 回答
