tcga生存分析
自己按照老师在社区里面发的那个http://www.omicsclass.com/article/173
整理了一个exprSet文本,前面运行都是正常的,
到unicox(‘某基因’)这一步以后,能导出计算的数值,
我想画成图,比如has.mir.206这个miRNA,
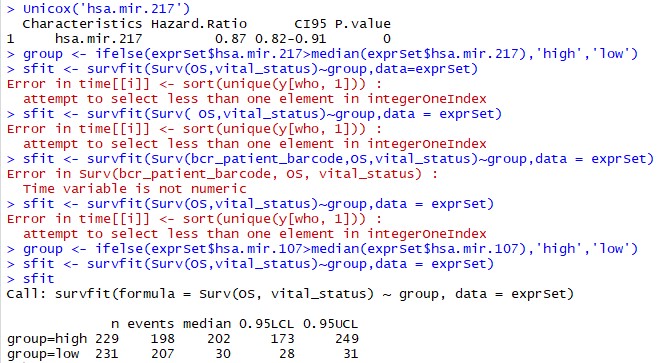
> group <- ifelse(exprSet$hsa.mir.206>median(exprSet$hsa.mir.206),'high','low')
> sfit <- survfit(Surv(OS,vital_status)~group,data=exprSet)
到sfit这一步报错了,
Error in time[[i]] <- sort(unique(y[who, 1])) :
attempt to select less than one element in integerOneIndex
我导入的exprSet是,
exprSet <- read.table("E:/TCGA/mirna-log2-2.txt",header = T,sep="\t",na ="NA")
不知道该怎么解决,问题是我还成功绘制过一张图片。。。不知道怎么解决
求教。。不太明白为何能计算出值,但是不能绘图
老师,我发现我的数据原本是count数据,然后我对他进行log2,出现了0和不可计算的值,我对出现的#NAME,我全部替换成NA,然后我发现还有一些数据能计算那个cox计算,但是它们里面存在0值的就无法绘图,是不是也需要把0值替换掉。求教了,因为我发现可以绘图的那几个数据不存在0值。。。。。。
请先 登录 后评论
- 1 关注
- 0 收藏,6158 浏览
- belive 提出于 2018-07-23 17:17