老师给我的指导如下
按理说一个线程应该只需要1.5g左右内存,肯定跑得过去的
可能其他容器占用的内存没有释放
你可以退出去把你有的容器全姗了
docker ps -a 查看计算机里的容器
docker rm -f [容器id] 删掉所有的容器,
然后重新启动,重新运行,把
p-n-job 改成 20
再跑就行
以后需要把你的问题整理一下挂到问答社区,涉及代码请粘贴代码及报错信息,然后在群里发布该问题链接等待回答(工作日);直接在群内提问,未提交问答社区的问题则被视为自由讨论问题!
我个人的设备:CPU 2*40, 内存64G.
docker设置 CPU40,内存54G
docker run -it -m 50G --cpus 35 --rm -v D:/ampliseq:/work omicsclass/ampliseq:v2.0
然后跑
### 物种注释 16S/18S #全长作为分类数据库:
qiime feature-classifier classify-sklearn \
--i-classifier $silva_classifier \
--i-reads ../02.feature_table/repset-seqs.qza \
--p-reads-per-batch 20 \
--p-n-jobs 20 \
--o-classification taxonomy.qza
5个小时后,报错,提示内存不够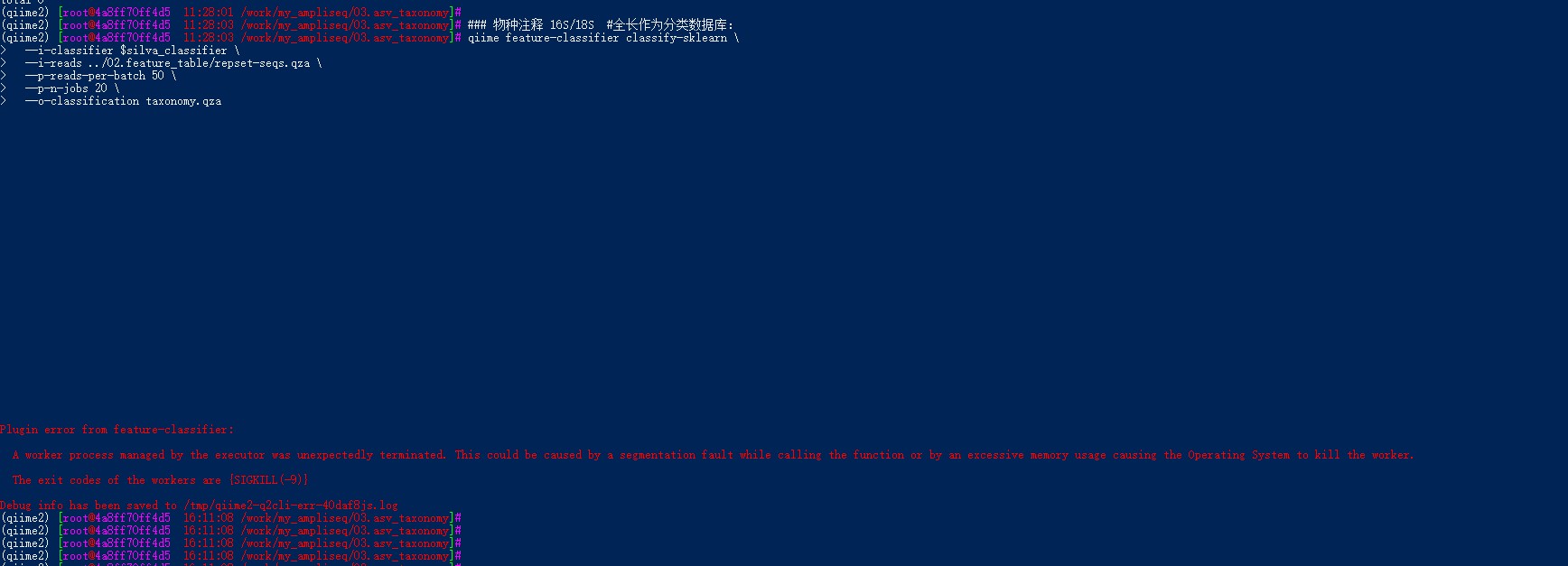
我的设备如何设置参数,保证能够跑下来这个分析?