性状数据分两种:
1. 数量数据:如基因的表达量,这个就采用基因的表达量即可,不需要改变
2. 类别数据: 这个数据需要转换成0,1 编码
WGCNA分析进行模块和性状相关性分析时,trait文件如何做?
例如以下面paper中的图为例
实验设计是:选取了两个大豆品种做转录组分析,分别是:大籽粒Wandou 28 (V1),小籽粒Peixian Layanghuang (V2),取样时期为三个时期:seed set (S1), seed growth (S2), and early seed maturation (S3),其中前两个时期的取样部位分别为:Seed pod with whole seed(S1),Whole seed(S2),S3时期取了两个部位分别为:Seed coat(S3-1),Seed cotyledon(S3-2),两个品种每个样品三个生物学重复共24个样品。

下图是课程中给的trait文件模板
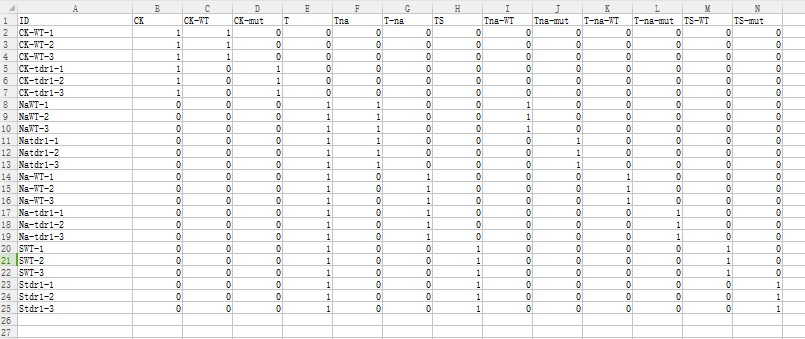 第一张图片对应的表格应该怎么制作?数值也是非0即1吗?横着的是性状名称,竖着的是样品名称,在这个案例中是一样的吗?对应的都是V1S1、V1S2、V1S3-1、V1S3-2、V2S1、V2S2、V2S3-1、V2S3-2?
第一张图片对应的表格应该怎么制作?数值也是非0即1吗?横着的是性状名称,竖着的是样品名称,在这个案例中是一样的吗?对应的都是V1S1、V1S2、V1S3-1、V1S3-2、V2S1、V2S2、V2S3-1、V2S3-2?