lncRNA功能主要通过cis或trans(进行trans作用分析需要至少5个样品)方式作用于蛋白编码靶基因来实现;
- Cis作用靶基因预测基本原理认为lncRNA的功能与其坐标临近的蛋白编码基因相关,于是将lncRNA临近位置的(上下游100k)蛋白编码基因筛选出来作为其靶基因。
- trans作用靶基因预测基本原理认为lncRNA的功能不依赖于和编码基因的位置关系,而与其共表达的蛋白编码基因相关。可以通过样本间lncRNA与蛋白编码基因的表达量相关性分析或共表达分析方法来预测其靶基因。当样本量>=5时采用Pearson相关系数法分析样本间lncRNA与蛋白编码基因的相关性,取相关性较高的蛋白编码基因进行功能富集分析预测lncRNA的主要功能;当样本数>=25时可采用WGCNA[41]将不同的组织、处理或者时间点间表达模式相似的基因聚类,得到不同的共表达模块,根据模块内已知的蛋白编码的基因功能进一步探索研究lncRNA的功能。
- 还有一种基于lncRNA与mRNA碱基互补配对而产生作用,利用LncTar[40]靶基因预测工具对我们的LncRNA进行靶基因预测。LncTar利用LncRNA和mRNA之间的互补序列,计算配对位点自由能和标准化自由能,标准化自由能阈值以下的则认为是LncRNA的靶基因。
你的样品数量太少无法通过相关性预测表达相关。
如果要通过相关性做的lncRNA-MRNA的互作关系,可以将每个mRNA的在不同样品的中表达量, 与每一个lncRNA 在不同的样品中的表达量做相关性分析;
R代码示例:
mrna<-c(33,44,55,62,11,22) #分别在多个样品中的表达量
lncrna<-c(22,33,2,43,2,44)
cor(mrna,lncrna,method="pearson")
如果想批量做相关性,建议学习R语言基础:R语言画图、R语言快速入门与提高、
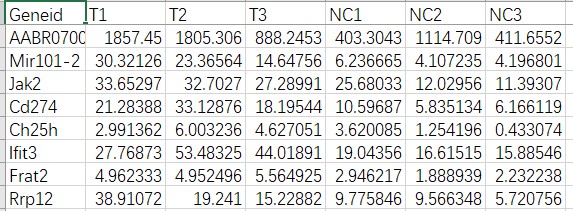 两组共6个样本,上千个差异lncRNA 、mRNA,表中数据是RPKM值,如何用R语言做lncRNA-mRNA皮尔森相关性分析?cor函数输入文件是什么格式?
两组共6个样本,上千个差异lncRNA 、mRNA,表中数据是RPKM值,如何用R语言做lncRNA-mRNA皮尔森相关性分析?cor函数输入文件是什么格式?