输入文件格式都没问题,运行的时候有报什么错误吗?
circos不能正常生成配置文件
把准备的几个文件都放在circos目录下,几个文件如下
gff文件CM.gff:
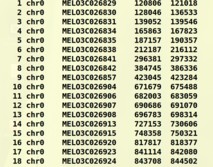
list文件CM_WRKY.collinear.pairs:

colline文件CM.collinearity:
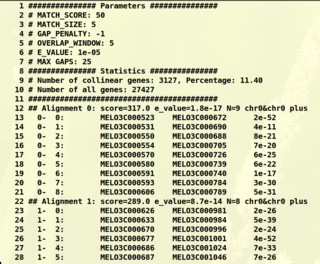
生成配置文件的命令如下:
perl ../script/colline_v2.pl -gff CM.gff -list CM_WRKY.collinear.pairs -colline CM.collinearity -od data -name WRKY
生成的data目录里只有WRKY.txt的文件是正常的
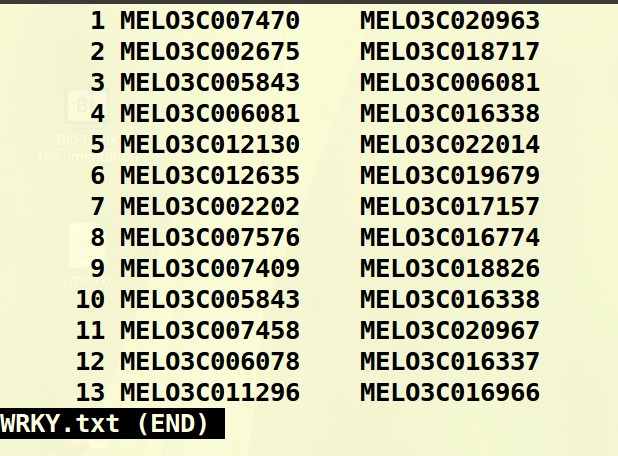 align文件只显示第一列
align文件只显示第一列
其余三个文件都是空的。
请先 登录 后评论
2 个回答
擅长:重测序,遗传进化,转录组,GWAS
输入的GFF文件里面的空格你搜索删除一下,可能是不必要的空格导致程序出错:
或者试试新脚本:
脚本下载:colline_v3.pl
使用方法:
#准备circos绘图数据文件,脚本从gff里面获得位置信息并整理数据
perl ../script/colline_v3.pl -gff ../MCScanX/AT.gff -list WRKY.collinear.pairs -colline AT.collinearity -od data -name WRKY
注意:gff文件是用于mcscanX分析的生成的GFF文件。
请先 登录 后评论
如果觉得我的回答对您有用,请随意打赏。你的支持将鼓励我继续创作!

- 2 关注
- 0 收藏,5516 浏览
- zhoy 提出于 2019-10-09 00:22
