单细胞转录组数据挖掘流程记录-LUAD(GSE123902)
单细胞转录组数据挖掘流程记录-LUAD(GSE123902)
数据介绍:
来自谷歌翻译:
通过转录分析跨肺腺癌不同进展阶段患者的单细胞,我们提供了未治疗原发肿瘤上皮再生的证据,以及跨越胚胎发育关键阶段和转移中肺形态发生的表型连续体。我们通过转录分析分析了 41,384 个来自非肿瘤相关肺(n=4)、原发性肺腺癌(n=8;7 个未治疗和 1 个新辅助化疗后)以及脑(n=3)、骨(n=1)和肾上腺(n=1)肺腺癌转移的单细胞。这些样本来自不同肿瘤进展阶段的患者,且未对特定细胞类型进行富集,确保整个肿瘤及其微环境均以无偏见方式采样。所有患者的数据被合并,创建了正常肺、原发肿瘤和转移的全球细胞图谱。
数据下载地址:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE123902
数据下载:
wget -c "https://www.ncbi.nlm.nih.gov/geo/download/?acc=GSE123902&format=file" -O GSE123902_RAW.tar #解压 tar xvf GSE123902_RAW.tar
准备map文件,链接统一文件名:
| GSM3516662_MSK_LX653_PRIMARY_TUMOUR_dense.csv.gz | LX653T |
| GSM3516663_MSK_LX661_PRIMARY_TUMOUR_dense.csv.gz | LX661T |
| GSM3516664_MSK_LX666_METASTASIS_dense.csv.gz | LX666M |
| GSM3516665_MSK_LX675_PRIMARY_TUMOUR_dense.csv.gz | LX675T |
| GSM3516666_MSK_LX675_NORMAL_dense.csv.gz | LX675N |
| GSM3516667_MSK_LX676_PRIMARY_TUMOUR_dense.csv.gz | LX676T |
| GSM3516668_MSK_LX255B_METASTASIS_dense.csv.gz | LX255BM |
| GSM3516669_MSK_LX679_PRIMARY_TUMOUR_dense.csv.gz | LX679T |
| GSM3516670_MSK_LX680_PRIMARY_TUMOUR_dense.csv.gz | LX680T |
| GSM3516671_MSK_LX681_METASTASIS_dense.csv.gz | LX681M |
| GSM3516672_MSK_LX682_PRIMARY_TUMOUR_dense.csv.gz | LX682T |
| GSM3516673_MSK_LX682_NORMAL_dense.csv.gz | LX682N |
| GSM3516674_MSK_LX684_PRIMARY_TUMOUR_dense.csv.gz | LX684T |
| GSM3516675_MSK_LX684_NORMAL_dense.csv.gz | LX684N |
| GSM3516676_MSK_LX685_NORMAL_dense.csv.gz | LX685N |
| GSM3516677_MSK_LX699_METASTASIS_dense.csv.gz | LX699M |
| GSM3516678_MSK_LX701_METASTASIS_dense.csv.gz | LX701M |
# 链接文件 |
cat map.txt |while read a b;do ln -s $a $b.csv.gz;done
数据分析:
样本准备mata文件:meta.tsv
| Sample | Patient_id | Tissue | Histology | Smoking | Pathology | EGFR_MUT | EGFR_Type | Stages | Stages_Status |
| LX653T | MSK-LX653 | tLung | ADC | Never | NA | EGFR_KMT2D_U2AF1_RET | MUT | IA | Low |
| LX661T | MSK-LX661 | tLung | ADC | Ex | NA | EGFR_TP53 | MUT | IA | Low |
| LX666M | MSK-LX666 | mBone | ADC | Never | NA | EGFR | MUT | IV | High |
| LX675N | MSK-LX675 | nLung | ADC | Never | NA | EGFR | MUT | IV | High |
| LX675T | MSK-LX675 | tLung | ADC | Never | NA | EGFR | MUT | IV | High |
| LX676T | MSK-LX676 | tLung | ADC | Cur | NA | Negative | WT | IA | Low |
| LX255BM | MSK-LX255B | mBrain | ADC | Ex | NA | EGFR_TP53 | MUT | IV | High |
| LX679T | MSK-LX679 | tLung | ADC | Ex | NA | KRAS | WT | IIA | Low |
| LX680T | MSK-LX680 | tLung | ADC(Double) | Ex | NA | TP53 | WT | IB | Low |
| LX681M | MSK-LX681 | mBrain | ADC | Ex | NA | Negative | WT | IV | High |
| LX682T | MSK-LX682 | tLung | ADC | Ex | NA | KRAS | WT | IB | Low |
| LX682N | MSK-LX682 | nLung | ADC | Ex | NA | KRAS | WT | IB | Low |
| LX684T | MSK-LX684 | tLung | ADC | Ex | NA | KRAS | WT | IA | Low |
| LX684N | MSK-LX684 | nLung | ADC | Ex | NA | KRAS | WT | IA | Low |
| LX685N | MSK-LX685 | nLung | ADC | Never | NA | NA | NA | IA | Low |
| LX699M | MSK-LX699 | mAdrenal | ADC | Ex | NA | KRAS_TP53 | WT | IV | High |
| LX701M | MSK-LX701 | mBrain | ADC | Never | NA | EGFR_TP53 | MUT | IV | High |
这次的数据是 h5 格式的也可以直接读入:
注意数据列分割设置逗号,需要转置一下
cat ~/LUAD/data/meta.tsv |sed '1d'| \
parallel -j 10 --colsep '\t' '
Rscript $scripts/seurat_sc_qc.r \
--count ~/LUAD/data/{1}.csv.gz \
--sep "," --transpose \
-p {1} --project {1} \
--nUMI.min 100 \
--nUMI.max 150000 \
--nGene.min 200 \
--nGene.max 10000 \
--mito.gene.pattern "^MT-" \
--percent_mito 20 \
--log10GenesPerUMI 0.8 \
--metadata.col.name Sample Sample_Origin Patient_id Tissue Histology Smoking Pathology EGFR_MUT EGFR_Type Stages Stages_Status \
--metadata.value {1} {3} {2} {3} {4} {5} {6} {7} {8} {9} {10}
'
cat ~/LUAD/data/meta.tsv | grep "^LX" | \
parallel -j 10 --colsep '\t' '
Rscript $scripts/seurat_CellCycleScoring.r -i {1}.afterQC.qs \
-c $scripts/cell_cycle/human_cell_cycle_genes.tsv -p {1}.CellCycleScoring
'
#2.3 双细胞分析
#DoubletFinder 分析需要做分群聚类分析之后才可以分析,另外作者不建议合并之后分析:
#参考:https://github.com/chris-mcginnis-ucsf/DoubletFinder?tab=readme-ov-file#input-scrna-seq-data
# 因此这里需要提前跑一下seurat的分群聚类分析
#seurat默认的标准化; 另外 SCT标准化需要加参数:--sctransform
cat ~/LUAD/data/meta.tsv|grep "^LX"|while read Sample Patient_id Tissue Histology Smoking Pathology EGFR_MUT EGFR_Type Stages Stages_Status;do
Rscript $scripts/seurat_sc_cluster.r --rds $Sample.CellCycleScoring.qs \
--resolution 0.5 -d 30 \
-p $Sample -o $Sample --cpu 20
## 如果是 SCT 标准化需要加参数:--sct
## 如果要去除双细胞增加参数:--removeDoubletCells
Rscript $scripts/DoubletFinder.r -i $Sample/$Sample.qs \
-p $Sample --annotations seurat_clusters --removeDoubletCells
done
#合并样本,
Rscript $scripts/merge_seurat_obj.r -i *.doubletFinder.qs -p all.sample.merged
# 分群聚类
Rscript $scripts/seurat_sc_cluster.r --cpu 10 --rds all.sample.merged.qs \
--integrate.method harmony --batch.id Sample \
--resolution 0.2 -d 50 \
-p luad.harmony -o luad.harmony
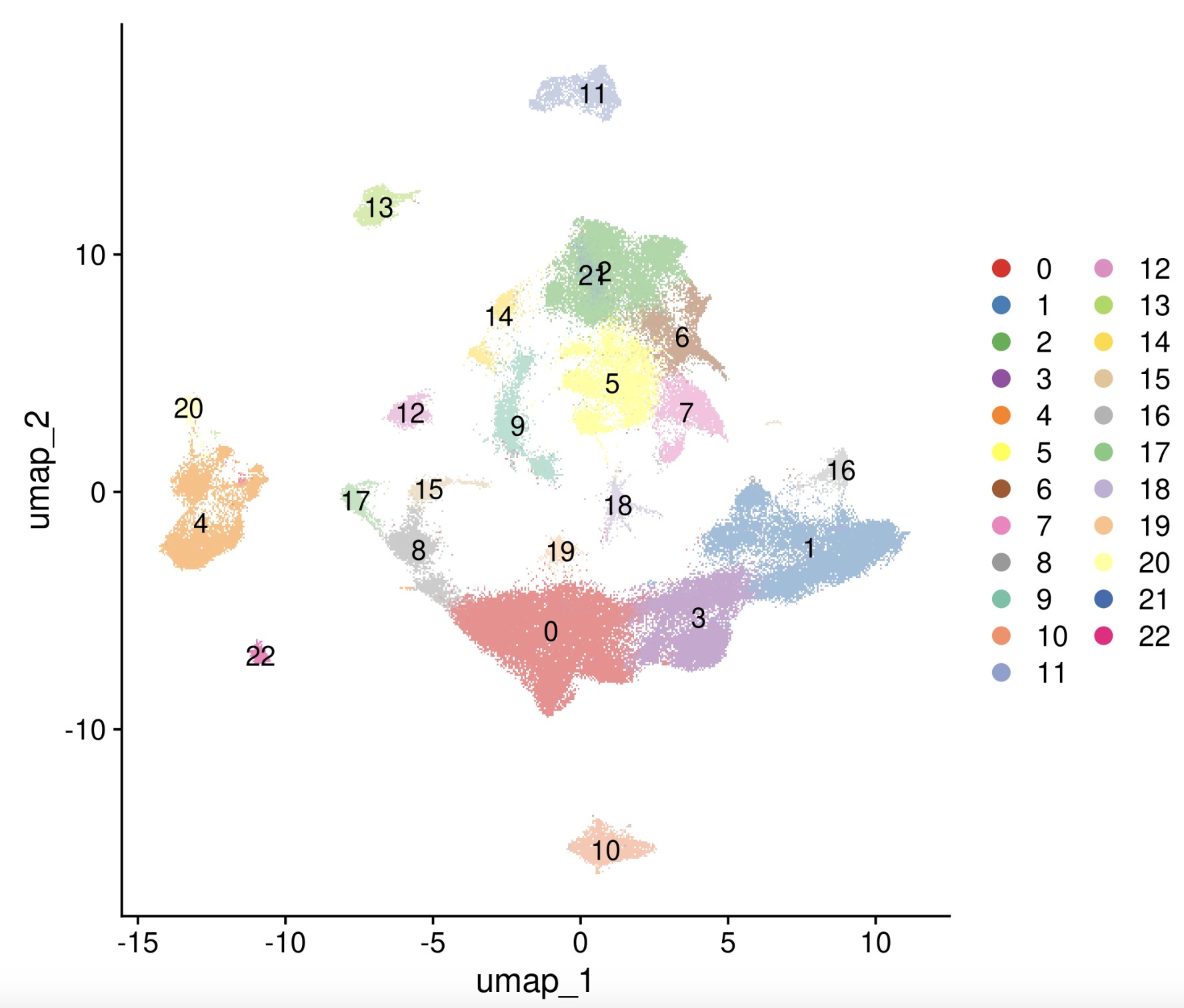
分析结果:
- 发表于 2026-01-13 14:21
- 阅读 ( 106 )
- 分类:转录组
你可能感兴趣的文章
相关问题
0 条评论
请先 登录 后评论
