修改单细胞转录组分组
修改单细胞转录组分组
修改单细胞转录组分组,
# 加载所需的R包
library(qs) # 用于快速读写R对象的包
library(dplyr) # 数据操作包
# 从指定路径读取Seurat对象
obj = qread("cnv_celltype_all.qs")
# 使用dplyr包对对象的元数据进行操作
obj@meta.data <- obj@meta.data %>%
mutate(
STAGE = case_when(
Stage == "I" ~ "StageI/II", # 将Stage I和II重新归类为"StageI/II"
Stage == "II" ~ "StageI/II", # 将Stage I和II重新归类为"StageI/II"
Stage == "III" ~ "StageIII/IV", # 将Stage III和IV重新归类为"StageIII/IV"
Stage == "IV" ~ "StageIII/IV", # 根据TNM分期系统进行重新分组
Stage == "T1N0" ~ "StageI/II", # T1N0期归为早期
Stage == "T2N0" ~ "StageI/II", # T2N0期归为早期
Stage == "T2N1" ~ "StageI/II", # T2N1期归为早期
Stage == "T2N2" ~ "StageIII/IV", # T2N2期归为晚期
Stage == "T2N2a" ~ "StageIII/IV", # T2N2a期归为晚期
Stage == "T4aN0" ~ "StageIII/IV", # T4aN0期归为晚期
Stage == "T4aN1" ~ "StageIII/IV", # T4aN1期归为晚期
TRUE ~ Stage # 其他未指定的Stage保持原值
)
)
# 将更新后的元数据保存为制表符分隔的文本文件
write.table(
data.frame(barcode=row.names(obj@meta.data), obj@meta.data),
file=paste("cnv_celltype_all_stage", ".metadata.tsv", sep=""),
sep="\t", # 使用制表符作为分隔符
quote=F, # 不对字符串添加引号
row.names=F # 不输出行名
)
# 将处理后的Seurat对象保存为qs格式文件
qsave(obj, "cnv_celltype_all_stage.qs")
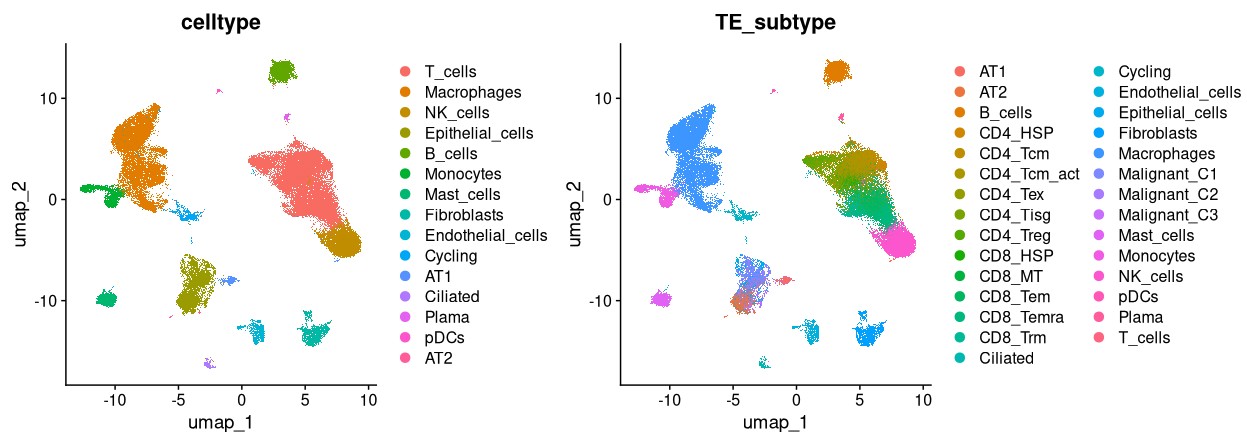
将分好的亚组按照细胞ID添加到总的seurat对象中:
tmeta=read.table("T_cells.harmony_addcelltype.metadata.tsv",header=TRUE,check.names = F,sep="\t",stringsAsFactors = F,row.names = 1)
emeta=read.table("Epi.harmony_addcelltype.metadata.tsv",header=TRUE,check.names = F,sep="\t",stringsAsFactors = F,row.names = 1)
obj_all=qread("luad.added.celltype.qs")
# 按**行名(细胞名)**把
# tmeta$Tsubtype
# emeta$Esubtype
# 加到 obj_all@meta.data 里
# 合并成一个新列 TE_subtype
# 其他没匹配到的细胞,保持原来的 celltype
# 先初始化 TE_subtype 为 celltype
obj_all@meta.data$TE_subtype <- as.character(obj_all@meta.data$celltype)
# 找到 T 细胞中能在 obj_all 里匹配到的行名
t_cells <- intersect(rownames(obj_all@meta.data), rownames(tmeta))
obj_all@meta.data[t_cells, "TE_subtype"] <- tmeta[t_cells, "Tsubtype"]
# 找到 E 细胞中能在 obj_all 里匹配到的行名
e_cells <- intersect(rownames(obj_all@meta.data), rownames(emeta))
obj_all@meta.data[e_cells, "TE_subtype"] <- emeta[e_cells, "Esubtype"]
- 发表于 2025-11-13 09:58
- 阅读 ( 1174 )
- 分类:转录组
你可能感兴趣的文章
- 单细胞分析 质控 线粒体 红细胞 核糖体 1453 浏览
- Infercnv|单细胞转录组拷贝数分析 5617 浏览
0 条评论
请先 登录 后评论
