IF=8.5|一千多个酵母基因组分析揭示基因家族进化的独特轨迹
“ 基因的获得和丢失是基因组进化的主要驱动力。文章以所有已知的酵母基因组为数据集,研究其基因家族进化轨迹,研究结果强调了基因家族收缩在酵母代谢、基因组功能和物种形成进化中的重要作用,并表明基因家族进化轨迹在主要真核生物谱系之间存在显著差异”
本文对1154个酿酒酵母(Saccharomycotina)基因组中基因家族的大规模比较表明,基因的获得和丢失驱动着酵母的进化。进化速度越快的谱系丢失的基因越多,物种形成的速度也越快,这凸显了基因家族收缩的作用。
01
—
文章内容
1. 基因家族多样性与真核生物的总基因含量相关
借助OrthoFinder工具分别分析四个群体(1154个酵母基因组、761个丝状子囊菌基因组、83个动物基因组及1178个植物基因组)的直系同源基因群(基因家族)。得到每个群体中的基因家族数目,揭示不同真核生物类群中保守基因家族的分布差异。并绘制四个群体中每个基因家族的平均覆盖率,如下图:
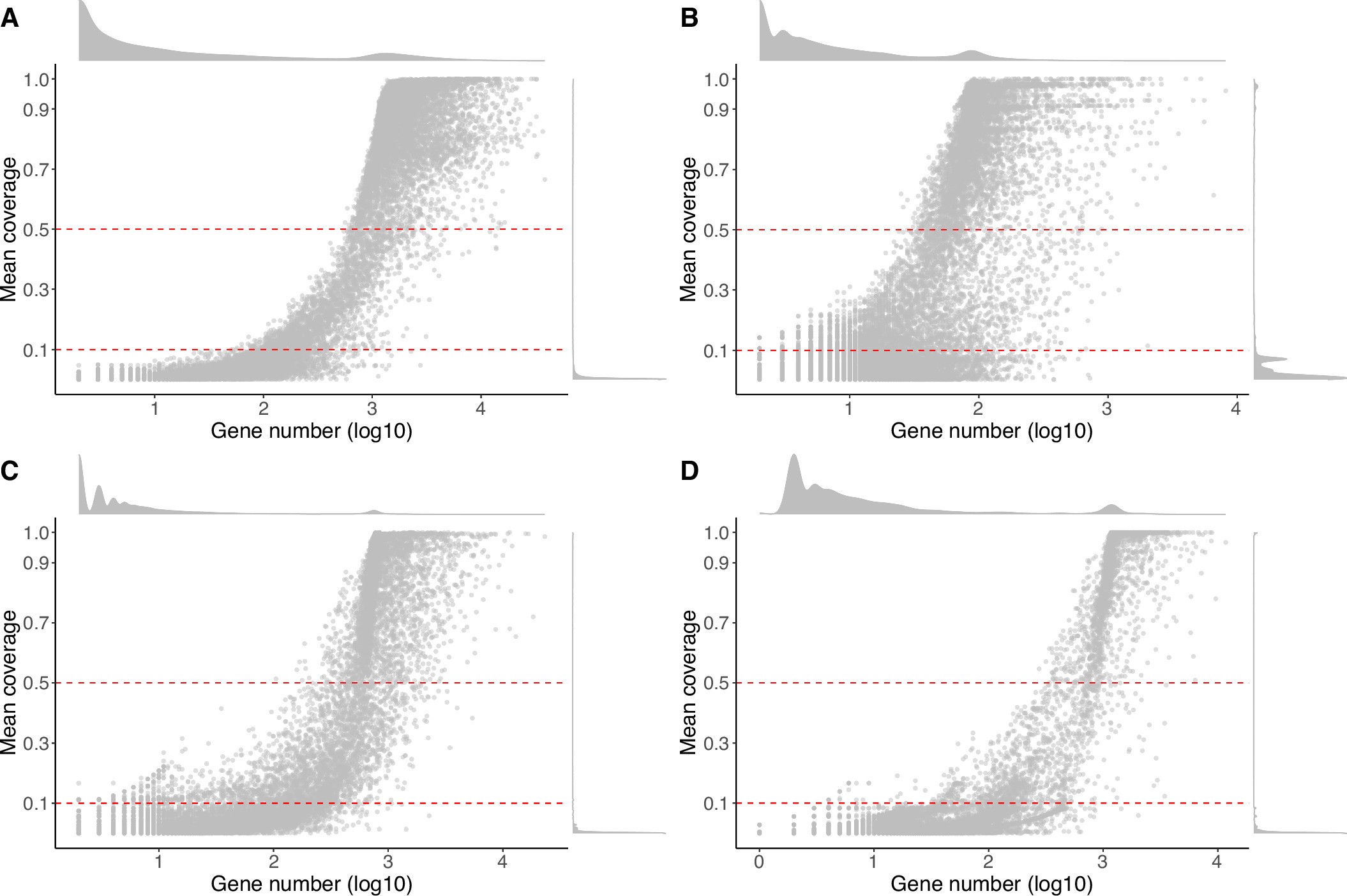
平均覆盖度表示跨进化枝的基因家族平均覆盖度( A ) 植物;( B ) 动物;( C ) 盘菌亚门;( D ) 酿酒酵母
之后计算每个群体的加权平均基因家族大小,得到酵母和丝状子囊菌的加权平均基因家族大小通常小于动物和植物。如下图A: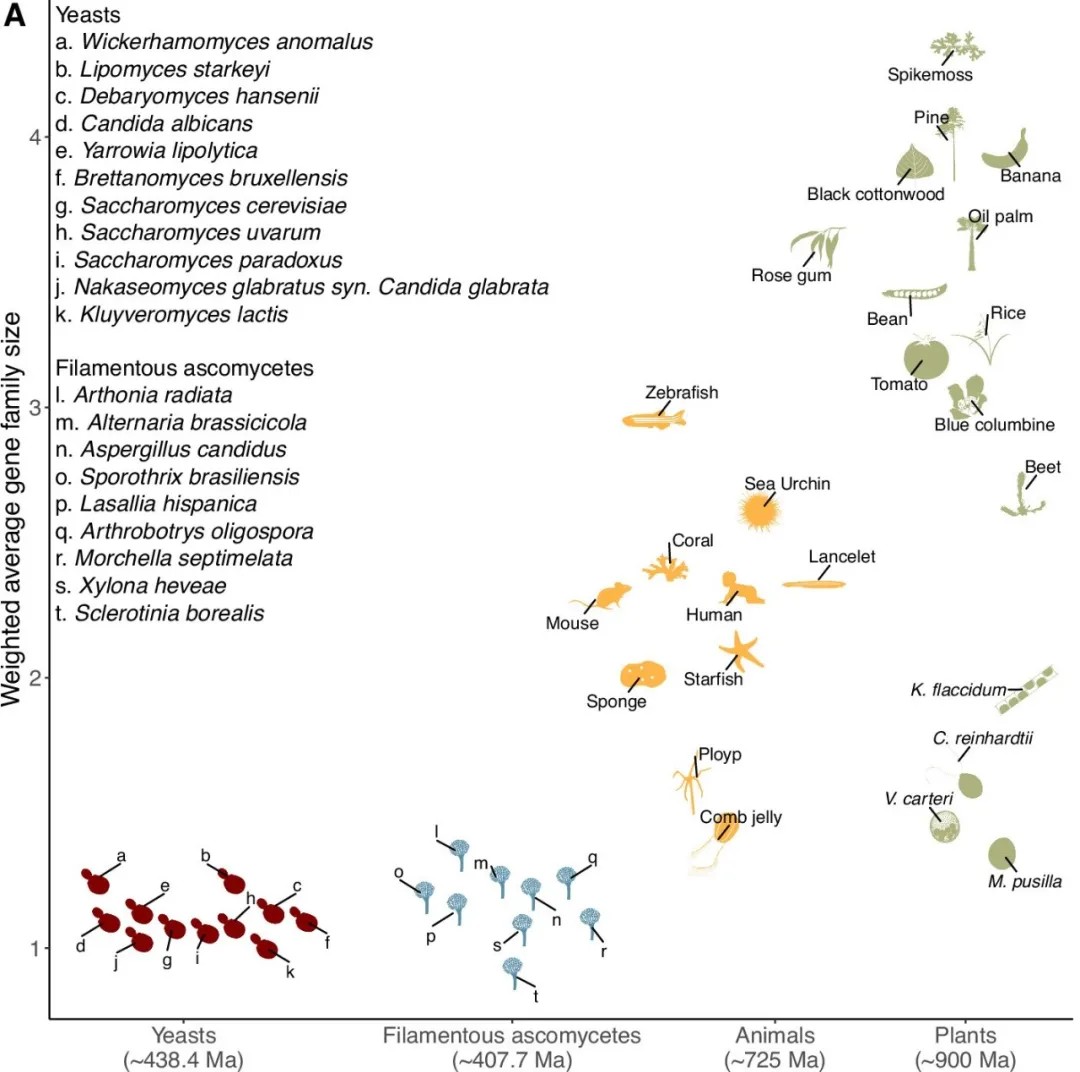 酵母、丝状子囊菌、动物、植物基因家族的加权平均大小
酵母、丝状子囊菌、动物、植物基因家族的加权平均大小
添加相应群体的蛋白质编码基因数值,作者发现加权平均基因家族大小的系统发育独立对比(PIC)与蛋白质编码基因的数量 (基因数) 之间存在很强的正相关。如下图C:
这表明,随着基因数量的增加,酵母往往具有更大的基因家族。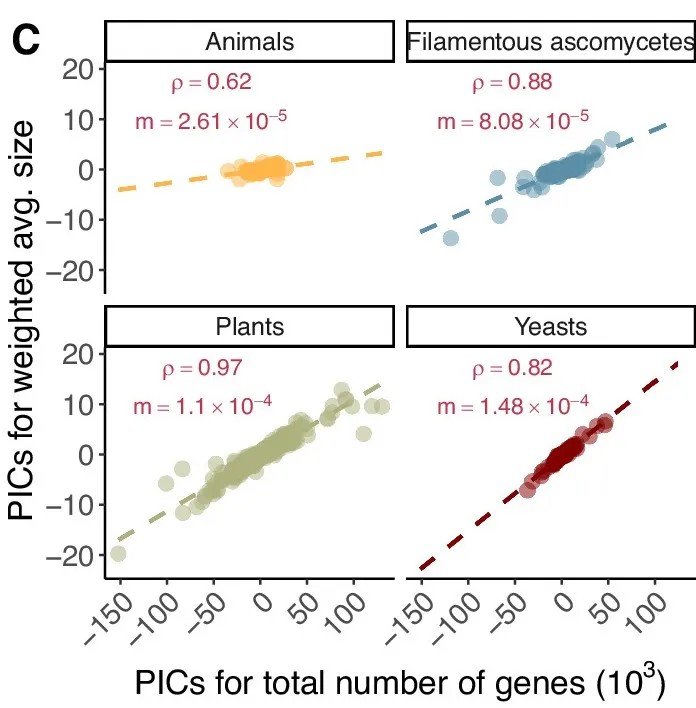 酵母、丝状子囊菌、动物和植物的加权平均基因家族大小的 PIC 与蛋白质编码基因总数之间的相关图
酵母、丝状子囊菌、动物和植物的加权平均基因家族大小的 PIC 与蛋白质编码基因总数之间的相关图
2. 基因家族含量的减少与基因组序列的快速进化有关
按照先前文献(Opulente et al,2024)中将1154种酵母分为12目,分别计算12目中加权平均基因家族大小Weighted avg. size和进化速率Evolutionary rate,并绘制箱线图A;针对在进化速率数值和加权平均基因家族大小数值存在显著差异的3个菌目绘制系统发育树图B;针对3个菌目中每个菌株的进化速率数值(图C)和加权平均基因家族大小数值(图D)进行位置展示。得到进化速率与基因家族数目有关且呈负相关,酵母中快速进化谱系(FELs)通过基因家族缩减和加速进化来驱动谱系分化。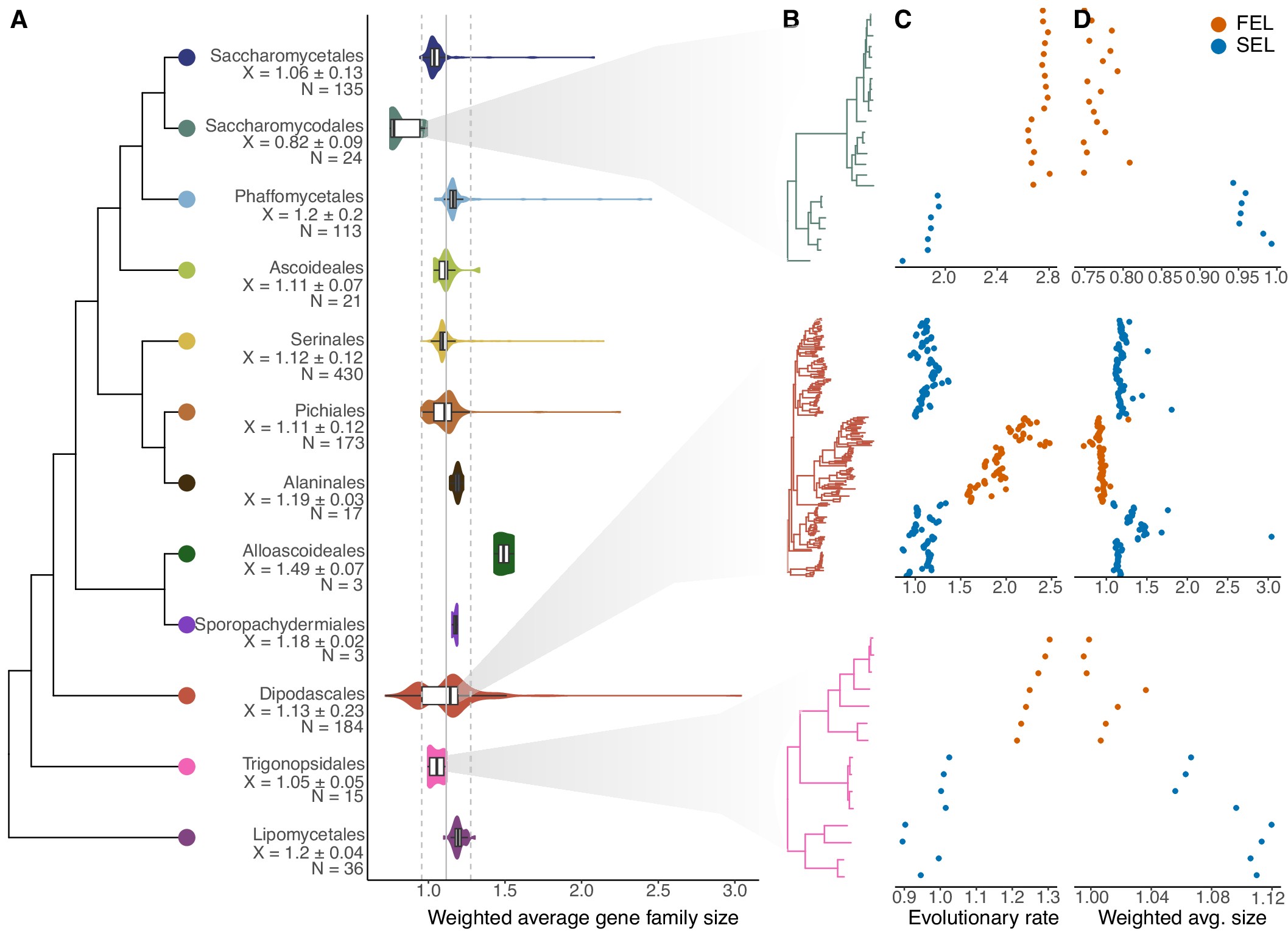
( A ) 来自先前研究 (Opulente et al, 2024 ) 的 1154 种酵母的系统发育。每个目的加权平均基因家族大小 (X) 和基因组数量 (N) 显示在相应目名称下方。
( B ) 由于 Trigonopsidales、Dipodascales 和 Saccharomycodales 3个目中的菌株之间的进化关系
(C、D)特定目内进化速率/加权平均基因家族大小的差异。每个点代表相应系统发育中的一种酵母
之后验证了3个菌目中快速进化谱系(FEL)经历了明显更多的基因家族收缩和丢失。如下图:
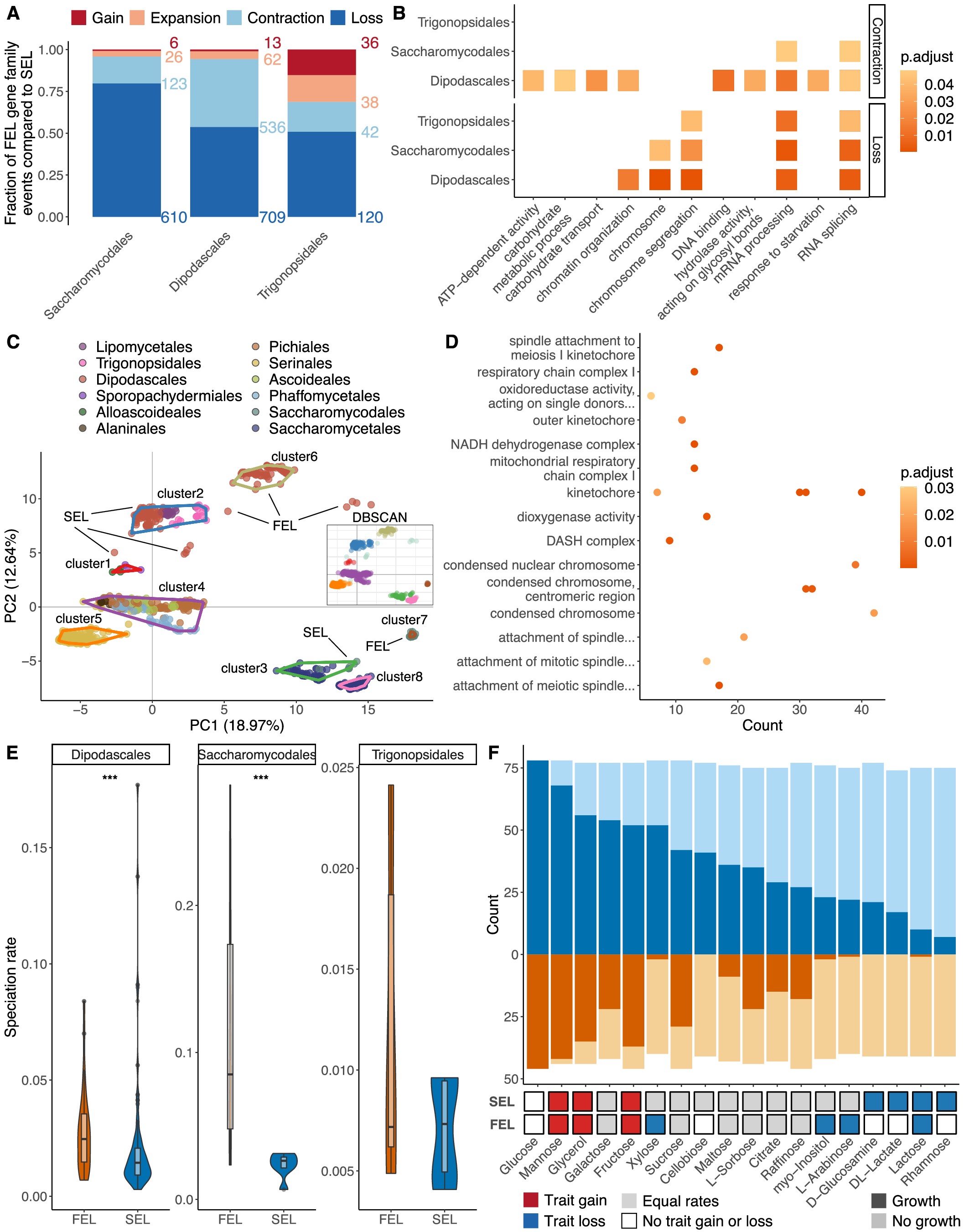 ( A ) 与 Dipodascales、Saccharomycodales 和 Trionopsidales 中的 SEL 相比,FEL 中的基因家族动态(丢失、收缩、扩增和增加)存在显著差异。( B ) 对基因家族中显著收缩或丢失的 GO 富集分析。( C ) 利用存在和缺失数据对 4262 个基因家族进行 PCA 分析。不同颜色点代表不同目,并使用颜色区分各个聚类(D)PC1 中前 610 个基因家族的 GO 富集分析。( E)使用 Wilcoxon 符号秩检验比较 Trigonopsidales、Dipodascales 和 Saccharomycodales 中的 FEL 和 SEL 的物种形成率(F) Dipodascales的 FEL 和 SEL 中 17 种碳源性状的进化史。深色表示能够利用该碳源的酵母数量。图中显示了三种不同的进化模型:性状获得(红色)、性状丧失(蓝色)以及性状获得和丧失速率相等(灰色)
( A ) 与 Dipodascales、Saccharomycodales 和 Trionopsidales 中的 SEL 相比,FEL 中的基因家族动态(丢失、收缩、扩增和增加)存在显著差异。( B ) 对基因家族中显著收缩或丢失的 GO 富集分析。( C ) 利用存在和缺失数据对 4262 个基因家族进行 PCA 分析。不同颜色点代表不同目,并使用颜色区分各个聚类(D)PC1 中前 610 个基因家族的 GO 富集分析。( E)使用 Wilcoxon 符号秩检验比较 Trigonopsidales、Dipodascales 和 Saccharomycodales 中的 FEL 和 SEL 的物种形成率(F) Dipodascales的 FEL 和 SEL 中 17 种碳源性状的进化史。深色表示能够利用该碳源的酵母数量。图中显示了三种不同的进化模型:性状获得(红色)、性状丧失(蓝色)以及性状获得和丧失速率相等(灰色)
3. 快速进化谱系FEL丢失了与 RNA 剪接、细胞分裂和代谢相关的基因
对3个菌目里FEL中收缩或丢失的基因家族的功能进行注释,如上图B,发现与RNA 剪接和 mRNA 加工相关的功能通路。在Dipodascales菌目 FEL中发现与碳水化合物代谢相关的基因家族经历了显著收缩。为了确定哪些基因家族的存在或缺失导致了 PCA 散点图(上图C)中酵母之间的分布差异,作者研究了酵母基因家族的存在与否与其在主成分上的坐标之间的相关性。最终鉴定出 610 个基因家族,如下图:强烈的负相关性表明 PC1 坐标的增加与 610 个基因家族的丢失相关。 在 PCA 中以 0.5 的覆盖阈值识别代表 PC1 的 610 个基因家族。
在 PCA 中以 0.5 的覆盖阈值识别代表 PC1 的 610 个基因家族。
4. 基因家族的丢失表明Dipodascales的FEL内存在非规范的剪接体、代谢途径和 DASH 复合物
基于前期分析结果和富集注释结果,作者重点关注了Dipodascales目中的前mRNA剪接途径、代谢途径和DASH复合物相关功能(图A)结果表明,Dipodascales FEL物种的基因缺失和收缩显著改变了它们的代谢能力。并展示在前mRNA剪接途径(图B)和碳代谢途径(图D)基因家族基因存在的位置和收缩扩张情况DASH复合物在真核细胞分裂中起着至关重要的作用,特别是在有丝分裂期间的染色体分离中。但是,与DASH复合物相关的基因在Dipodascales FEL中大量丢失(图C)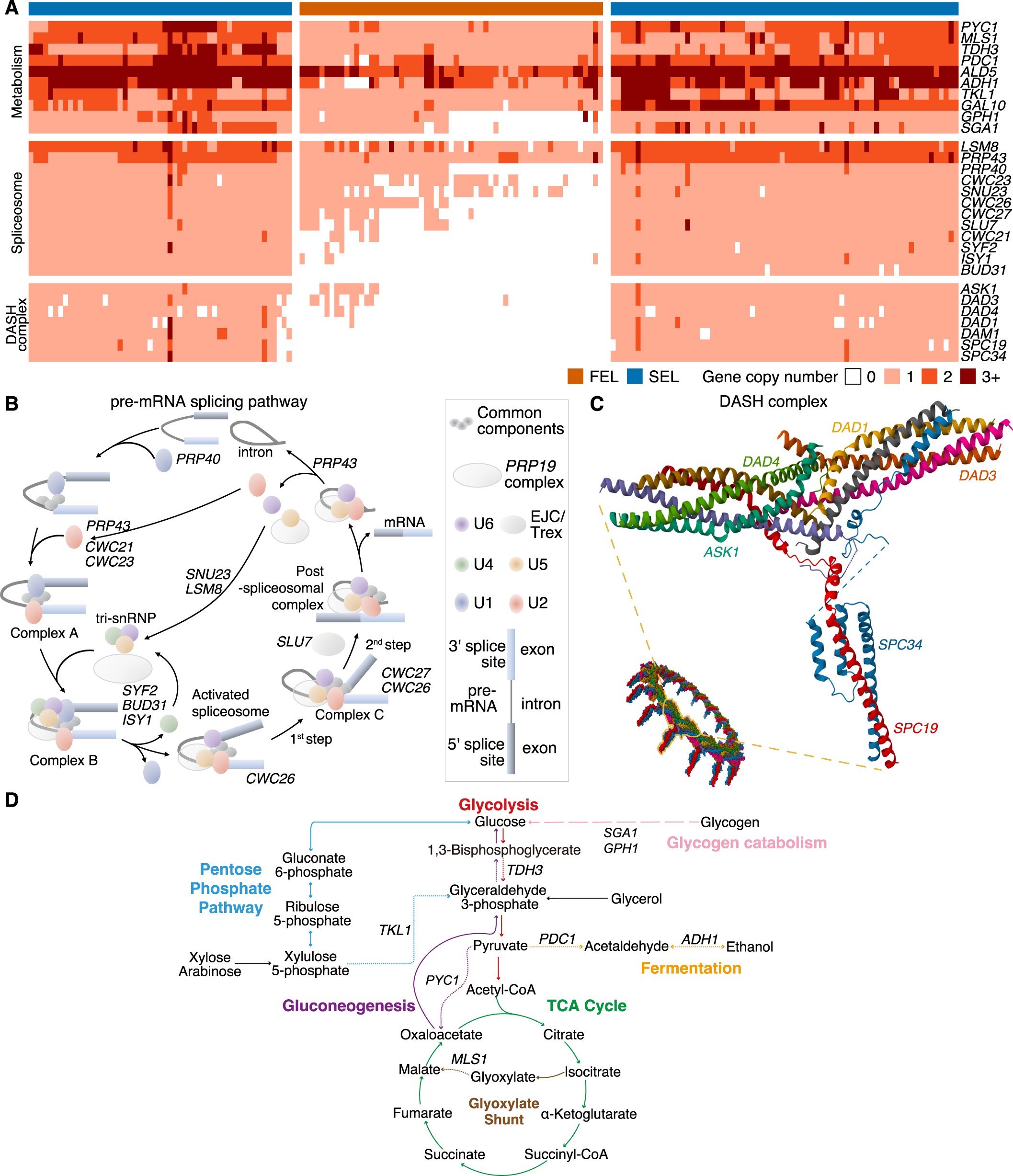 ( A ) Dipodascales 中代谢途径(10 个基因家族)、前 mRNA 剪接途径(12 个基因家族)和 DASH 复合物(7 个基因家族)的基因拷贝数详细图片。列颜色表示 SEL(蓝色)和 FEL(橙色)。( B ) 前 mRNA 剪接途径。基因家族名称标记在 FEL 中经历收缩或丢失的途径中编码的特定步骤。( C ) 编码 DASH 复合物的基因。( D ) Dipodascales FEL 中包含广泛基因丢失或收缩的碳代谢途径。途径名称和反应以相应的颜色表示。黑色箭头表示甘油(在FEL中获得)以及木糖和阿拉伯糖(在FEL中损失)进入中心碳代谢的位置。
( A ) Dipodascales 中代谢途径(10 个基因家族)、前 mRNA 剪接途径(12 个基因家族)和 DASH 复合物(7 个基因家族)的基因拷贝数详细图片。列颜色表示 SEL(蓝色)和 FEL(橙色)。( B ) 前 mRNA 剪接途径。基因家族名称标记在 FEL 中经历收缩或丢失的途径中编码的特定步骤。( C ) 编码 DASH 复合物的基因。( D ) Dipodascales FEL 中包含广泛基因丢失或收缩的碳代谢途径。途径名称和反应以相应的颜色表示。黑色箭头表示甘油(在FEL中获得)以及木糖和阿拉伯糖(在FEL中损失)进入中心碳代谢的位置。
5. 绘制酵母基因家族收缩扩张进化树图,呈现基因净丢失的总体趋势
为了探究在 CAFE 分析中,每个酵母目中经历显著收缩或扩张的基因家族的进化趋势,作者计算了这些基因家族的净变化(所有分支的净增加或减少)。如下图所示:为了探索在每个目的最近的共同祖先中获得的新基因,作者选择针对孤儿基因家族(即目特异性基因家族)进行检查,揭示不同目之间的差异。在 NCBI 非冗余(NR)蛋白质数据库中识别孤儿基因的潜在同源蛋白后,我们发现 24,577 个孤儿基因(96.5%)似乎是从头出现的。只有 36 个基因(0.2%)可能起源于酵母菌门最近的共同祖先之后的物种形成或复制事件,而 865 个基因(3.4%)可能是水平基因转移的结果。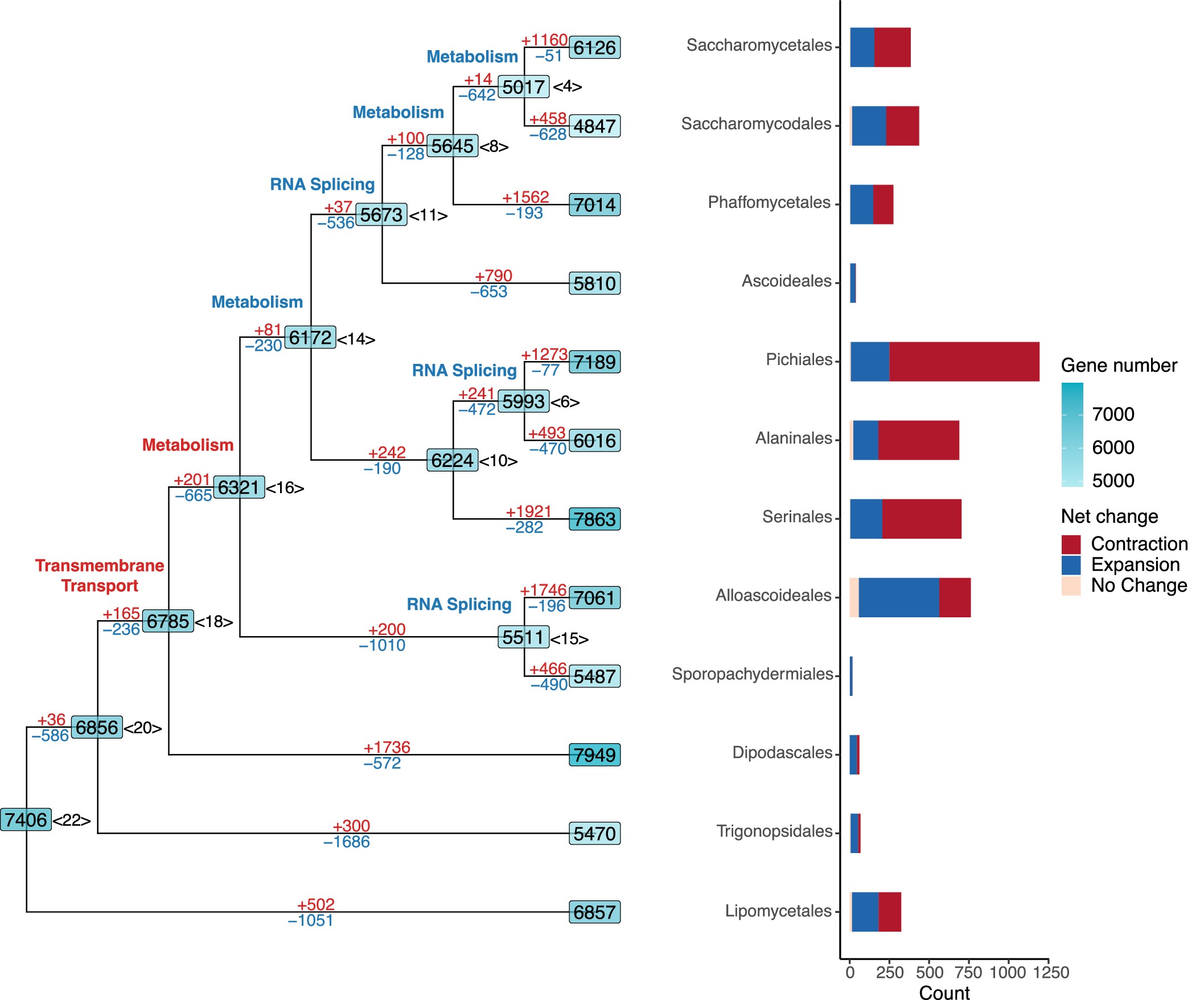 为了简化树结构,每个目的 MRCA 之后的分支均已折叠。每个节点上都标记了基因计数,此外,分支用富集 GO 术语中的关键术语进行注释(P ≤ 0.05)。树右侧的条形图量化了每个目的 MRCA 之后系统发育中基因家族的净变化。y 轴标记为“计数”,反映经历净变化的基因家族数量(分为扩张/收缩/无变化)
为了简化树结构,每个目的 MRCA 之后的分支均已折叠。每个节点上都标记了基因计数,此外,分支用富集 GO 术语中的关键术语进行注释(P ≤ 0.05)。树右侧的条形图量化了每个目的 MRCA 之后系统发育中基因家族的净变化。y 轴标记为“计数”,反映经历净变化的基因家族数量(分为扩张/收缩/无变化)
结果总结
一些基因家族的收缩和 FEL 的丢失可能有助于酵母提高进化和物种形成率,因为它们能够快速进行基因组适应,从而优化在多样化和具有挑战性的环境中生存和繁殖所必需的细胞过程。
与 SEL 相比,FEL 表现出更多的基因丢失;然而,这些丢失并不经常与特定的性状丢失相关,而是呈现复杂的进化图景,既可能是环境适应的主动选择结果,也可能是基因组精简的被动产物
酵母以基因家族收缩为主导的进化路径与其他真核生物依赖基因复制的扩张模式形成鲜明对比,揭示了不同谱系适应策略的根本差异。
真菌基因组多样性由渐进性基因家族收缩(适应持续生态压力)和偶发性扩张(如全基因组复制)共同塑造,反映了长期精细适应与短期爆发式创新的动态平衡。
本研究开发了一种全面的基因家族分析方法,探索酵母谱系之间和谱系内的基因家族大小差异,并比较酵母与其他三个古老谱系(植物、动物和丝状真菌)之间的基因家族进化。
这些分析揭示了塑造基因家族组成的进化压力,并重建了超过1000个基因组的祖先基因家族历史。该方法建立了一个可应用于生命树其他主要分支的比较框架。
文章链接:http://dx.doi.org/10.1038/s44320-025-00118-0
更多生信课程
- 发表于 2025-06-03 10:14
- 阅读 ( 1150 )
- 分类:文献解读
你可能感兴趣的文章
- 怎么得到基因组之间的结构变异结果? 1072 浏览
- cactus 软件:从基因组比到泛基因组分析 1956 浏览
- cactus下载 781 浏览
- cactus报错:Failed to get attribute: partialOrderAlignmentProgressiveMaxRows from cactus XML 764 浏览
- IF=4.1|玉米ARF基因家族的泛基因组分析和表达验证 1808 浏览
相关问题
- 泛基因组 1 回答
- 在运行table_annovar.pl脚本的时候报错 0 回答
- 运行go_enrich.r报错 0 回答
- 在运行core_pan_gene_curve.r的时候报错 1 回答
- 泛基因组 eggNOG得不到结果-2 0 回答
- 泛基因组 eggNOG得不到结果 0 回答
