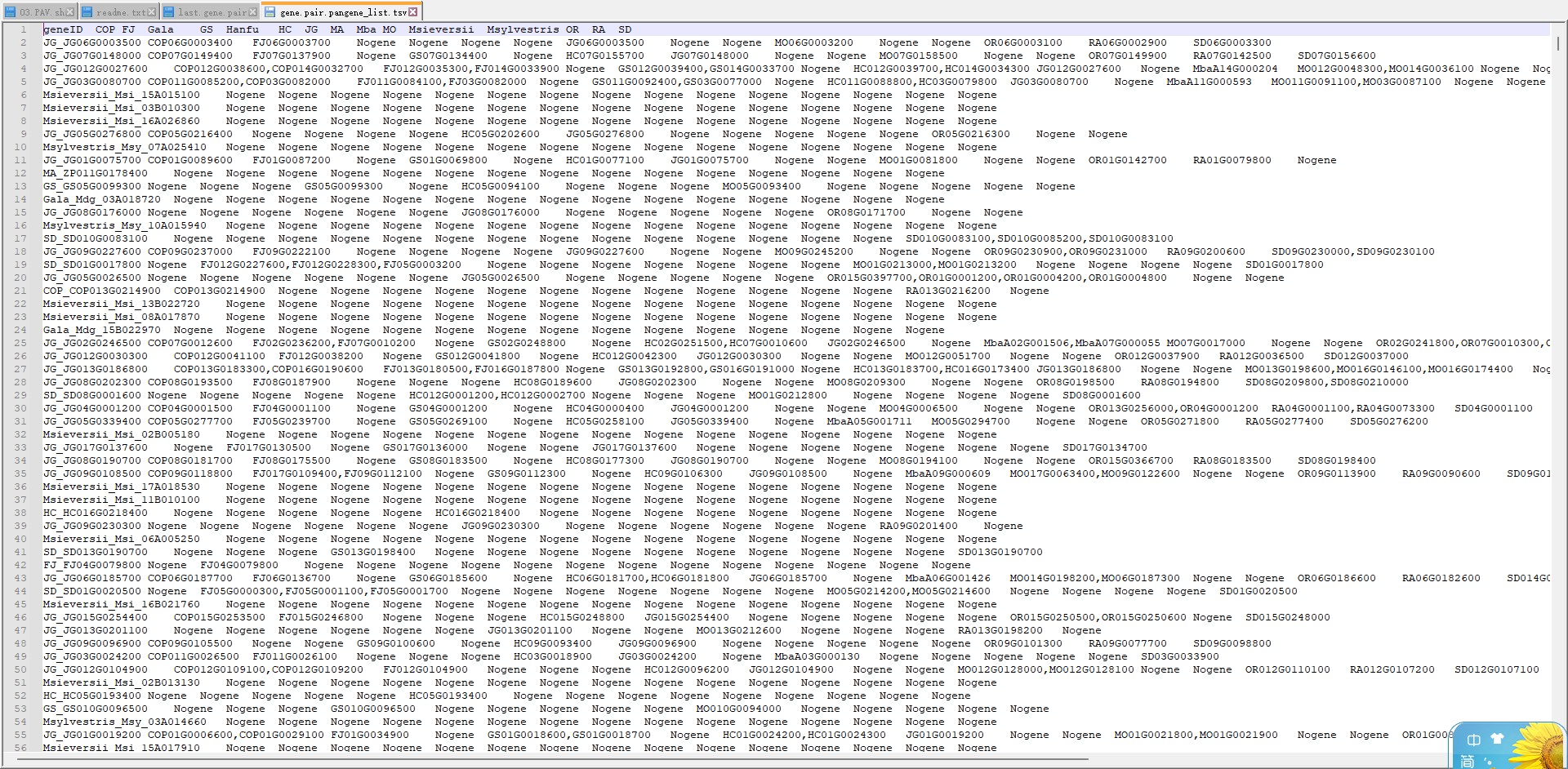
第一个问题:项目结果文件有个readme文件,里面有每个文件的说明:表格pangene_list.tsv每一行是一个基因,及其对应不同基因组的基因ID,每一列是一个基因组。
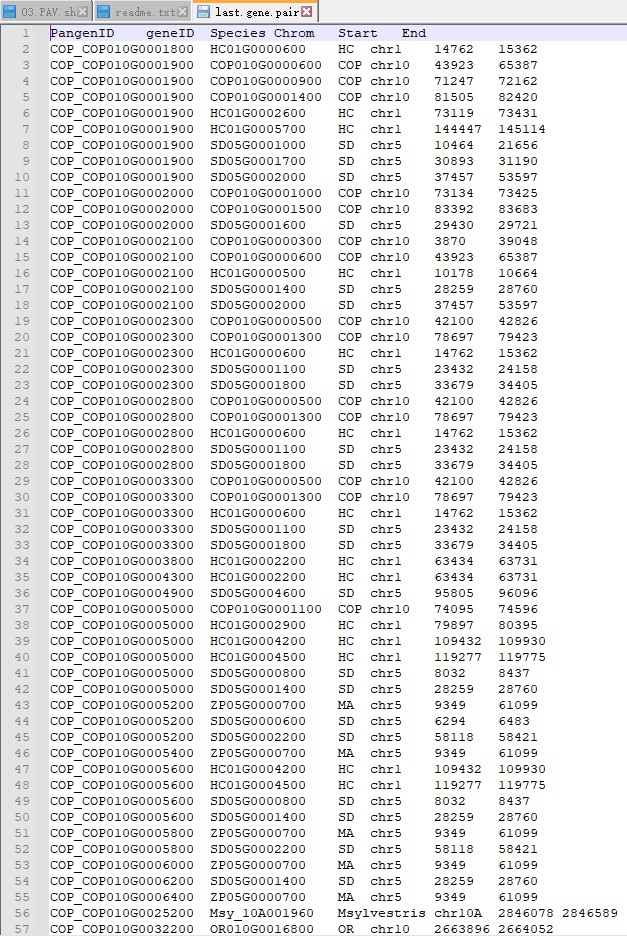
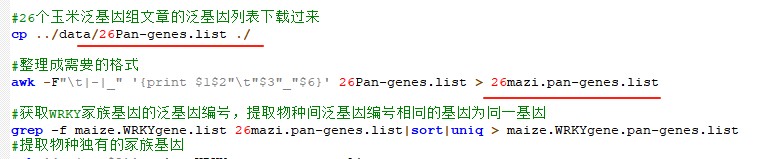
第二个问题:后续用来做泛基因家族分析需要用到的文件是*.gene.pair,该文件仅保留了有共线性关系的最佳匹配基因对。需要整理成后续脚本需要的格式。shell中的26Pan-genes.list文件是从文献中下载的,也是需要整理成后续脚本需要的格式。
泛基因家族分析
1 个回答
- 1 关注
- 0 收藏,791 浏览
- 提出于 2025-07-28 09:44