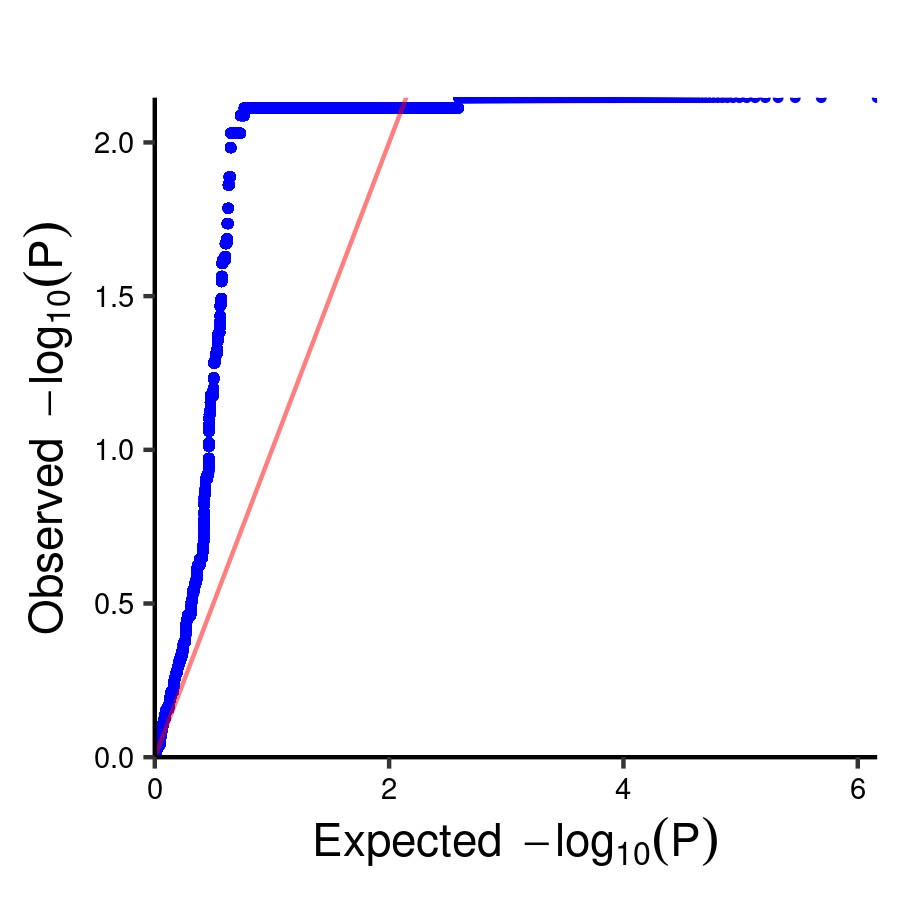
整整两天,梳理了两三遍数据,原来是因为表型数据的样本名称和.vcf文件的样本名称不匹配(-和_不统一),从而导致模型拟合度很差,P值填充的NaN值比例高达44%,而正常绘图的NaN值比例仅为10%左右,下面这两张曼哈顿图也是同样的原因
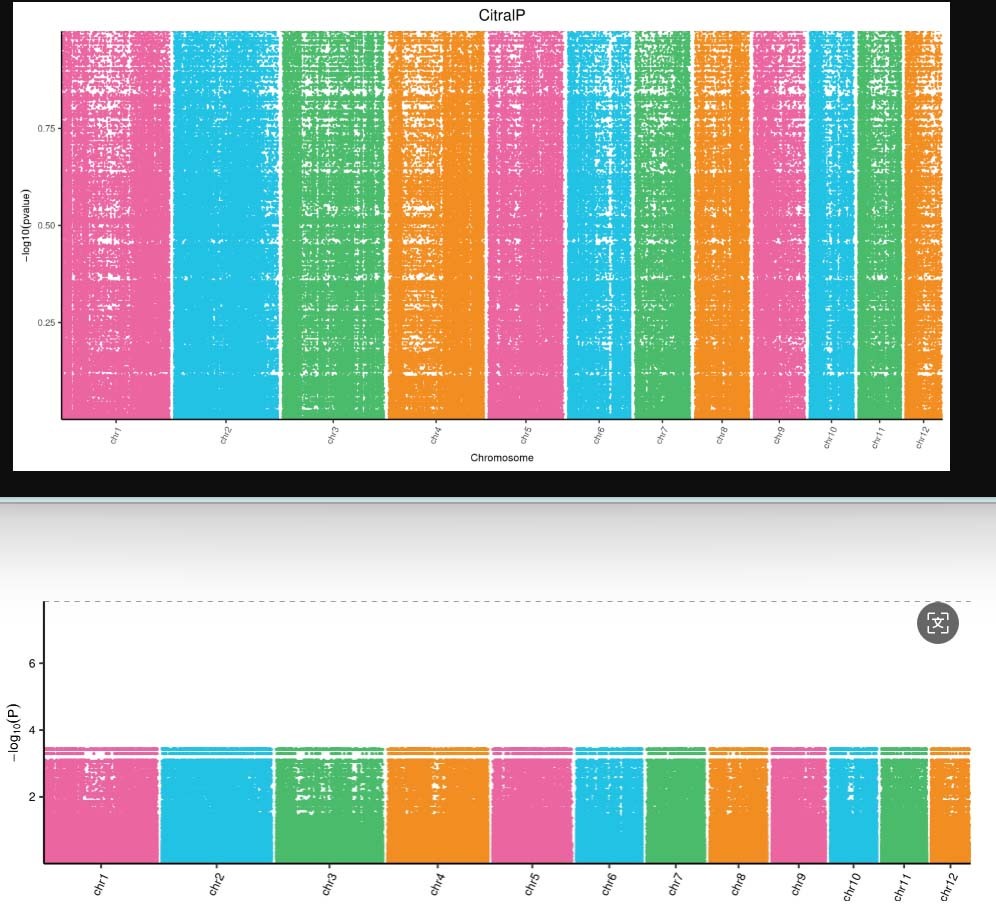
老师您好,想请教您个问题,我有两年数据,两年合在一起的数据和20220年数据分析正常,但是2025年的数据分析异常,用tassel分析了一般线性模型和混合线性模型,用GEMMA做了一般线性模型,结果都如图所示,运算脚本
tassel GLM模型:ohup run_pipeline.pl -Xmx60G -fork1 -h hapmap_2025.ordered.hmp.txt \
-fork2 -importGuess $tassel_trait_2025 \
-combine3 -input1 -input2 -intersect -FixedEffectLMPlugin \
-endPlugin -export CitralP_GLM_2025&
输入文件的内容:
可视化脚本:
cut -f 2,6 CitralP_GLM_20251.txt|sed 's/:/\t/' >CitralP_glm_2025_pvalue.txt
sed -i '1s/Marker/chr\tpos/' CitralP_glm_2025_pvalue.txt
nohup Rscript $scriptdir2/qq_plot.r -i CitralP_glm_2025_pvalue.txt -n CitralP_glm2025_qq&
输入文件内容:
1 chr pos p
2 chr1 19907 0.67482
3 chr1 26506 0.72296
4 chr1 26507 0.72296
5 chr1 26632 0.72296
6 chr1 26634 0.72296
7 chr1 26641 0.72296
8 chr1 26645 NaN
9 chr1 26672 0.72296
10 chr1 26711 NaN
11 chr1 26735 0.76476
12 chr1 26737 0.76476
13 chr1 26793 0.72296
出错的QQ图如下: