- 0 关注
- 0 收藏,3831 浏览
- 黄新鑫 提出于 2019-11-19 10:36
相似问题
- linux 下 R 命令怎么后台运行 2 回答
- wgCNA一个模块中找hub基因 我把cytoScapeinput-edges文件输入cytoscape中 用cytohubba计算 但是每次出来的结果都不一样 试了很多遍也不行 求解答 谢谢! 0 回答
- 我得到转录组分析的wgcna数据需通过cytoscape筛选关键模块的hub基因,边的权重应该设定多少来筛好 1 回答
- WGCNA中过滤低表达量(0.5)时datExpr0=datExpr0[1:n,datExpr0[n+1,] > meanFPKM]不成功,且变量从上步30769变成了0. ? 2 回答
- rJava 安装包错 1 回答
- R包安装 1 回答
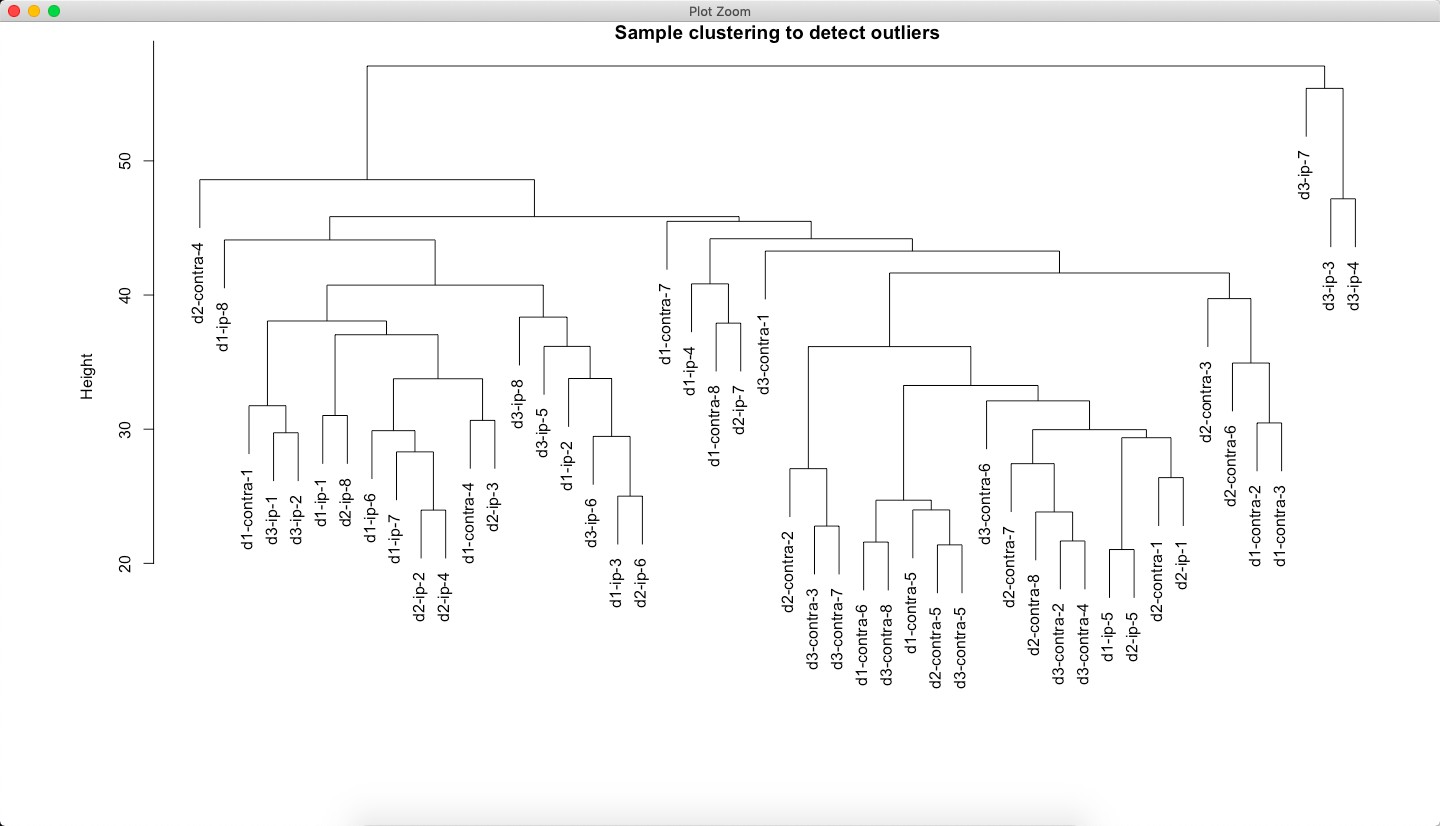 请问这个聚类树高度为什么这么高?我看其他人的数据一般只有零点几。
请问这个聚类树高度为什么这么高?我看其他人的数据一般只有零点几。