索引文件没有建立成功,可能是内存不够导致的,你按照这个方法尽量多个docker内存:
https://www.omicsclass.com/article/1413
老师,我利用demo数据进行转录组分析。
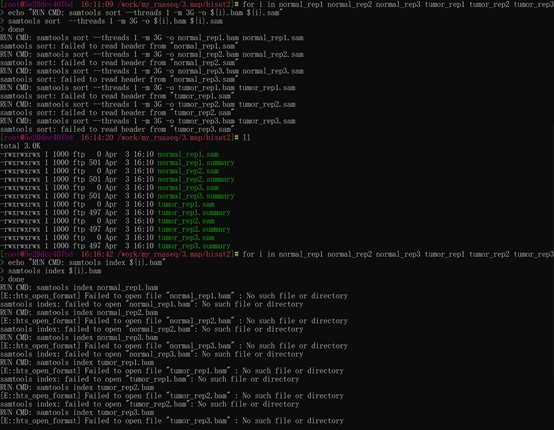
在尝试将demo的数据,将sam 转换成bam 格式并排序时并没有转换成功。不太清楚哪里出了问题。
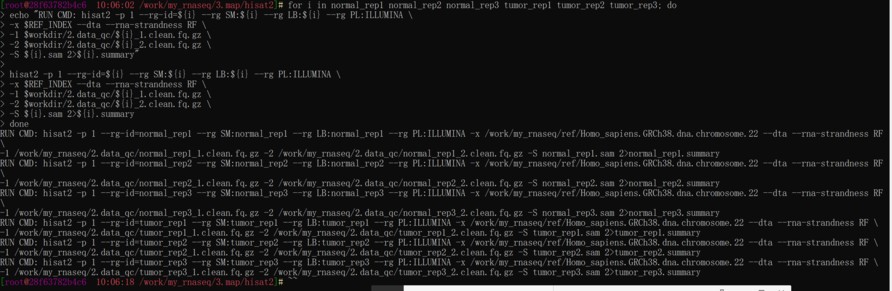
我不知道demo数据是否是链特异性文库,所以也就没去设置(RF or FR)。也不会设置。
所以,是不是文库设置这里出问题了?导致后面的sam转换成bam不成功?
……………………………………………………………………
查看normal_rep1.sam,里面内容为空;
查看normal_rep1.summary,结果如下:

好生奇怪,不知那里出错?
提示hisat2-align exited with value 1。 这个问题怎么解决?