做GWAS分析很多软件直接输入VCF文件就可以 比如TASSEL,为啥一定要生成VCF表格呢? VCF文件看懂说明见:https://www.omicsclass.com/article/6
你需要用什么软件做GWAS你查看软件说明就好了,要求输入什么文件格式你自己准备一下;
你自己的数据编号有啥区别,我怎么会知道呢?
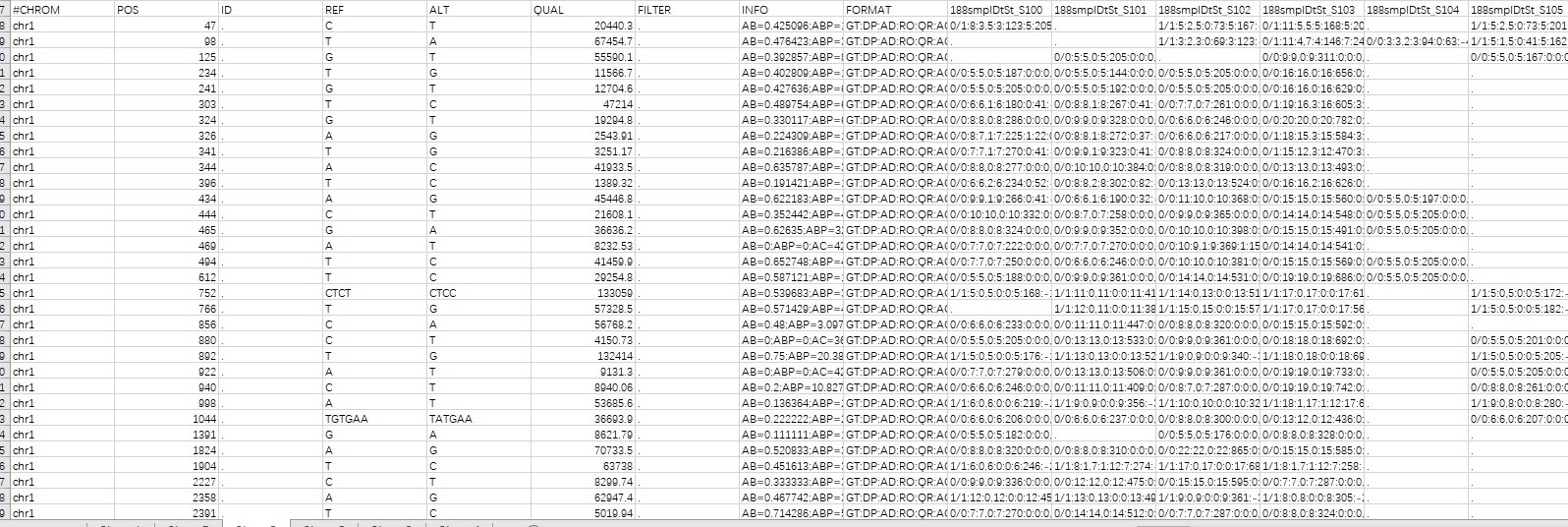
老师您好,我得问题是这样的,这是我拿到的VCF文件,我想生成SNP表来进行GWAS分析,我有一下几个问题:
1.REF和ALT如果显示碱基数量大于一个,但是只有一个有差异,那我在生成SNP表格的时候是按照那个不一样的表示吗?
2.关于样本的命名:每一个前面都有sm的标号,这个文件里有188sm 90sm和9sm,老师请问不同sm标号是因为他们测序时分批测得吗?是不是可以理解为188sm命名得那一组属于一个时间段测序的。
3:老师,样本下的GT如果为杂合子0/1,请问老师这个SNP位点的碱基参考的是REF还是ALT
4:老师,有的样本下面为.表示什么意思?在生成SNP表时有什么表示呢?
谢谢老师