我将序列ID的后缀t1或者p1全部都去掉,使基因ID,转录本ID和蛋白质序列ID都一致,且没有后缀,然后就可以了。
共线性分析之前提取的蛋白质序列为空。如何解决?
要通过allmRNAID.txt文件,从蛋白质序列文件中获取对应蛋白序列,使用命令perl ../script/get_fa_by_id.pl allmRNAID.txt ../Phaeodactylum_tricornutum.ASM15095v2.pep.all.fa pep.fa。结果所得文件内容为空。查看pep.all.fa文件,里面的蛋白质序列后缀是.p1,而非allmRNAID.txt中转录本序列的后缀.t1.
请教老师,这个时候,我该如何使它们的ID一致呢?将pep.all.fa中的“p1”全部替换成“t1”吗?
该物种的gff3.文件里,gene的ID没有后缀,mRNA和exon的后缀都是t1,而CDS的后缀是p1.如图1.
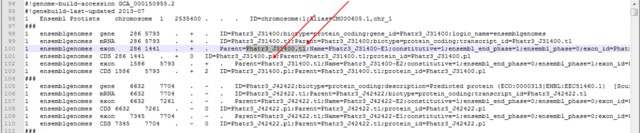 图1:gff3的数据
图1:gff3的数据
该物种的.pep.all-.fa文件里,蛋白序列ID的后缀是p1.如图2.
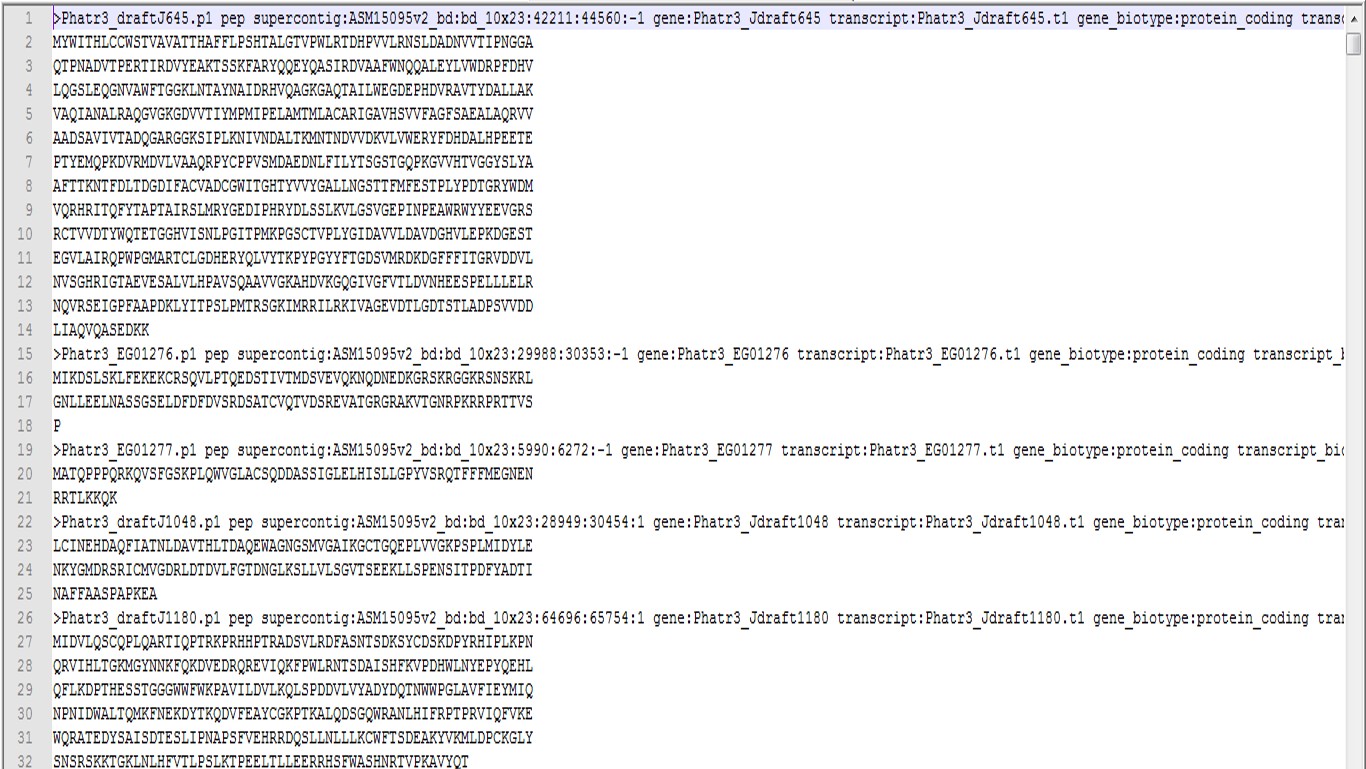
图2 pep.all.fa的数据
请先 登录 后评论
- 1 关注
- 0 收藏,2595 浏览
- 星野 提出于 2020-05-10 22:03