数据下载,可根据TCGA网站的筛选条件添加GDCquery()参数,详情见:
关于TCGA的下载,学了课程还是遇到点问题,求老师指点
老师的课程是讲的胆管癌,样本少,用老师的代码也没问题。但是自己实操中,遇到情况就有点复杂,不会用了。
我是要结肠癌和直肠癌,并且要腺癌的样本数据,有点复杂,这是我网页筛选条件,代码不知道怎么写。
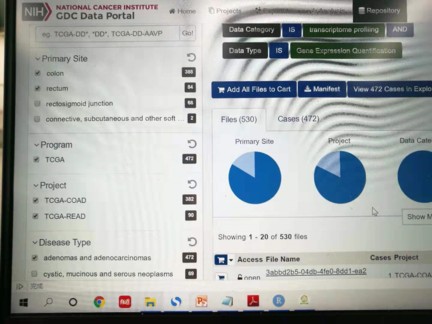
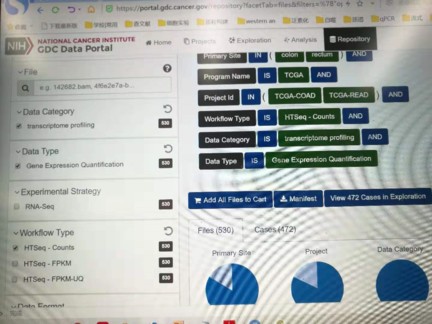
看了论坛里老师筛选原发位点的帖子
 我想先根据原发位点筛一下
我想先根据原发位点筛一下
> library(GenomicDataCommons)
> resp = cases() %>% filter(~ {project.project_id=="TCGA-COAD"|project.project_id=="TCGA-READ"}&{primary_site =="colon"|primary_site="rectum"}) %>% GenomicDataCommons::select(c(default_fields(cases()),'samples.sample_type')) %>% response_all()
报错了。。。。。Error: No method asJSON S3 class: formula
然后其实也不明白就是获得了sample_download,后边的代码怎么写,请老师看看是这样么?
GDCdownload(query = sample_download,files.per.chunk=6, method='client')
data <- GDCprepare(query=sample_download)
data_expr <- assay(data)
因为我最终需要的是结直肠癌腺癌基因表达数据。我也想过先结肠癌、直肠癌一个个下载下来,然后手动删掉不是腺癌的。可是老师的代码都是基于query,就是我手动整理好我也不知道后边代码怎么写。比如GDCprepare(?)
请先 登录 后评论
1 个回答
- 1 关注
- 0 收藏,2810 浏览
- jing 提出于 2019-11-11 14:49
相似问题
- TCGA下载 docker报错 0 回答
- TCGA差异分析如何根据临床信息分组? 1 回答
- TCGA用R数据下载错误 1 回答
- 老师,想请教一下TCGA基因表达数据的问题,我从xena.ucsc网页上下载了基因表达数据TCGA-CESC.htseq_counts.tsv;然后发现该数据中只有Ensembl格式的基因ID ,没有SYMBOL格式的。所以接下来进行基因ID格式转换,却发现同一个SYMBOL ID对应的多个Ensembl格式的ID,想问下老师,这种情况该怎么处理?同一个SYMBOL ID所对应的多个Ensembl格式ID的基因表达数据应该留下哪一个? 1 回答
- TCGA的RNA-seq count annotation过滤,有些不知道是否需要删除 0 回答
- 代码报错 1 回答
