不太了解,没有用过这个软件。
各位老师,想问一下关于LAI评估基因组 组装质量的问题
我用ltr-finder 找ltr,然后用ltr-retriever 计算LAI,基本跑通了,但是遇到点问题如下:
1 有个小警告,但是可以继续运行
 2 最后结果总是ltr太少,我想知道是我的运行的命令问题还是我输入的基因组有问题呢?
2 最后结果总是ltr太少,我想知道是我的运行的命令问题还是我输入的基因组有问题呢?
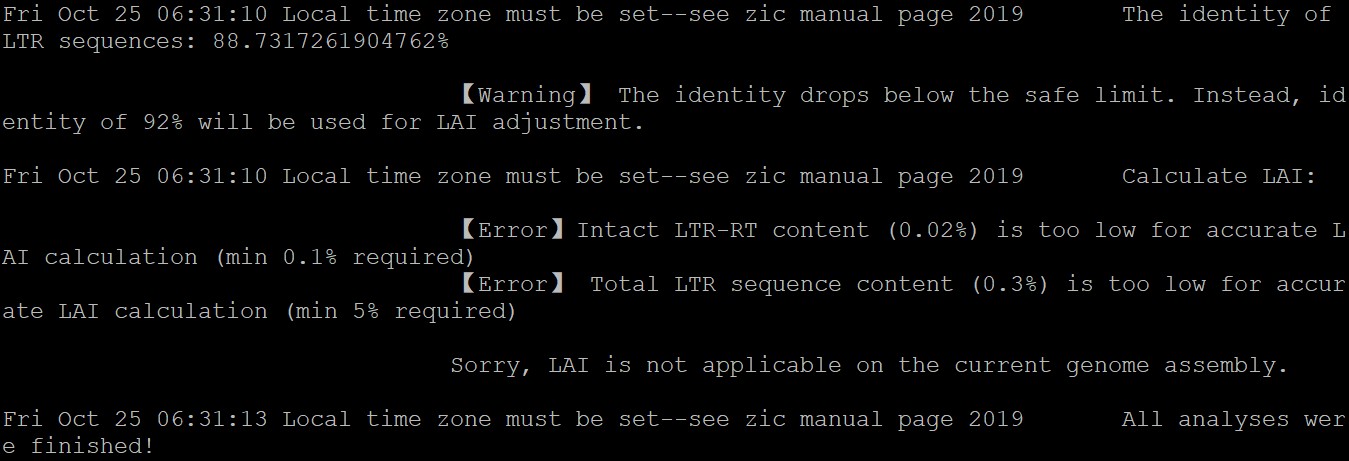
3 我输入的是全基因组fasta文件,总共344M,把它分成了8 个小库分别运行,每个.fa 与.scn 都是对应的。我的命令是:
./app/LTR_Finder/source/ltr_finder -D 15000 -d 1000 -L 7000 -l 50 -p 20 -C -M 0.8 ./rawdata/smdlt_test.fa > ./rawdata/smdlt.finder.scn
./LTR_retriever/LTR_retriever -threads 4 -genome ./rawdata/1.fa -infinder ./rawdata/1.finder.scn
各位有用过这个软件的吗?谢谢大家了~~~
请先 登录 后评论
1 个回答
- 1 关注
- 0 收藏,3326 浏览
- 取个名字好伤脑子哦..... 提出于 2019-10-25 16:08
