5 circos基因加倍分析最后一步报错
用circos进行基因加倍分析时,最后一步,提示如下错误。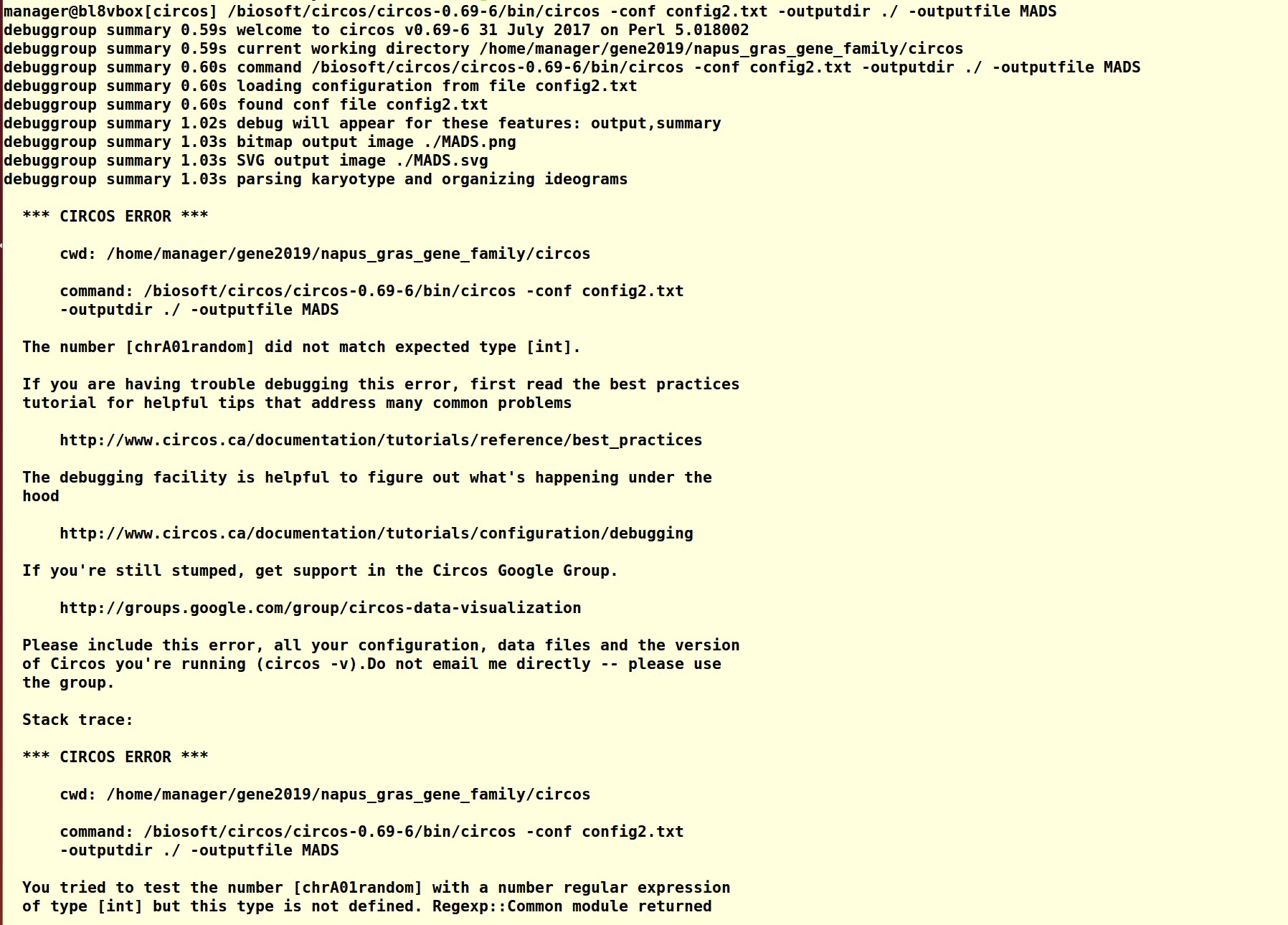

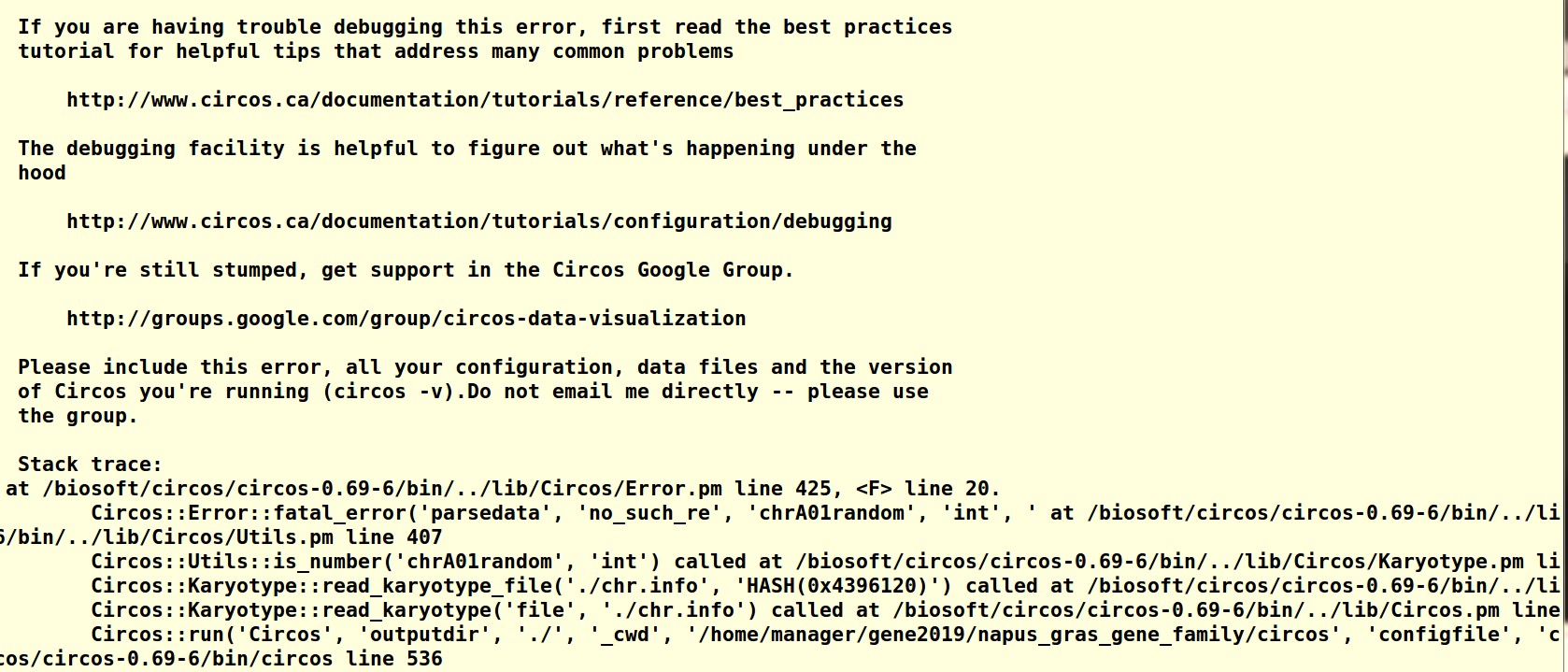
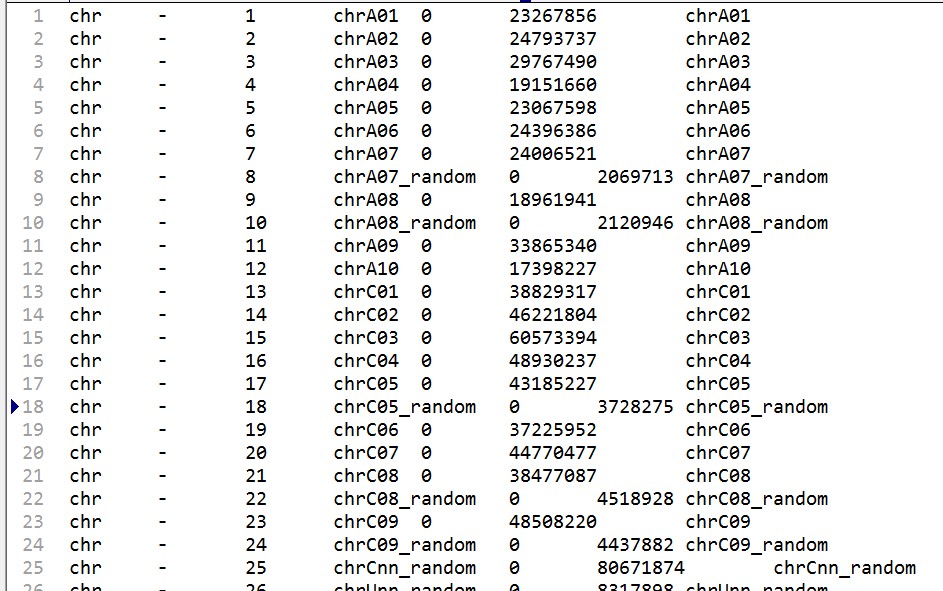
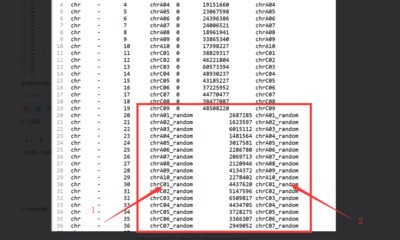
用的config文件是培训课上config2,txt文件,chr.info截图如下: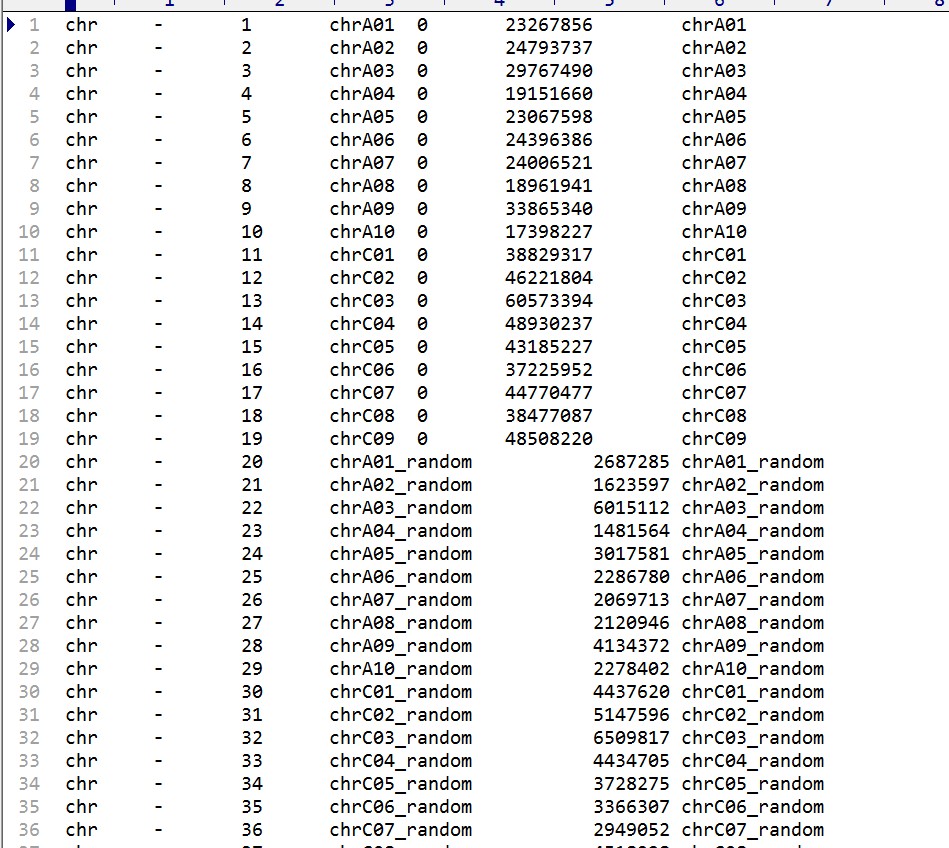
请问哪里出问题了呢?
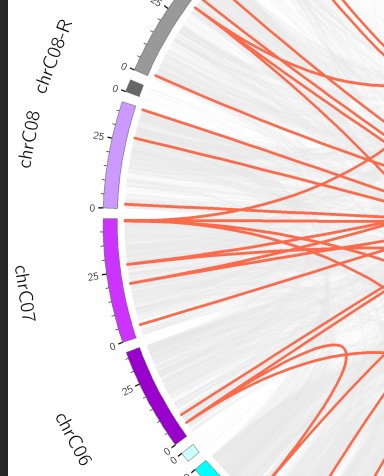 这是我的图的一部分
这是我的图的一部分
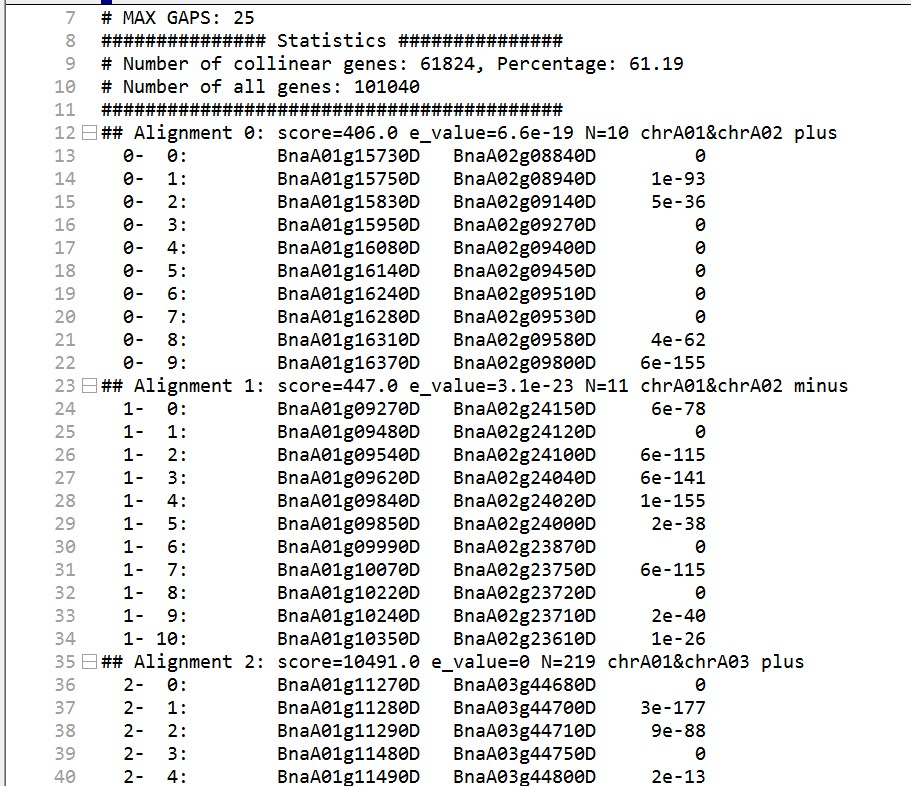
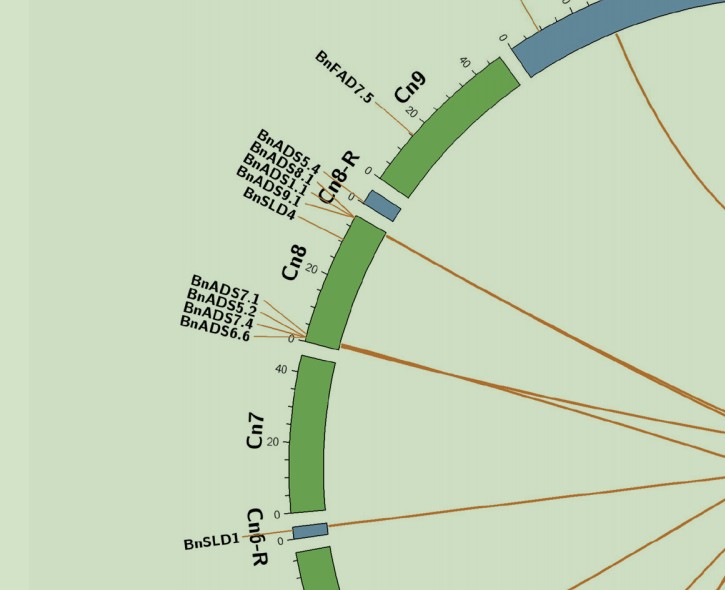 这是别人发表文章当中的图。
这是别人发表文章当中的图。
请先 登录 后评论
其它 3 个回答
擅长:perl,基因家族,linux,chip-seq
你的chr.info文件是有问题的,你可以参考一下这篇文章:https://www.omicsclass.com/article/644。
最后一列是按照人的染色体编号来设置染色体的颜色。
请先 登录 后评论
如果觉得我的回答对您有用,请随意打赏。你的支持将鼓励我继续创作!

这只是一个绘图软件,不是加倍分析软件。。。。。
根据提示应该是核型文件 里面染色体相关的错误,第三列对应的是实际的染色体,第四列是标注染色体编号,而最后一列表示的是颜色,并没有你这个格式编码对应的颜色形式,你可以修改一下看是1部分染色体无法识别(三列四列)还是2部分(最后一列)颜色格式的问题。并且,注意你后半段染色体信息种没有第五列的 0
可以参考一下:https://www.omicsclass.com/article/644
请先 登录 后评论
- 4 关注
- 0 收藏,3316 浏览
- 吹不成调 提出于 2019-05-13 20:28