合并要修改下ID吧,毕竟domain1和domain2的的ID 都是同一个 会有重复
合并提取后的domain序列之后,linux系统中的clustalw不能读出蛋白信息
根据下面的脚本操作:
#提取结构域序列,最后的evalue值根据实际情况可调,注意脚本提取的是第一个domain,如要提取其他domain,请修改脚本27行$a[9]==1为第一个,$a[9]==2为第二个,依次类推
perl script/domain_xulie.pl WRKY_hmm_out.txt Arabidopsis_thaliana.TAIR10.pep.all.fa WRKY_domain.fa 1.2e-28
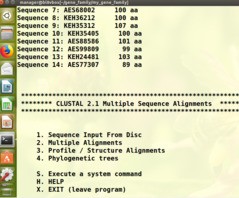
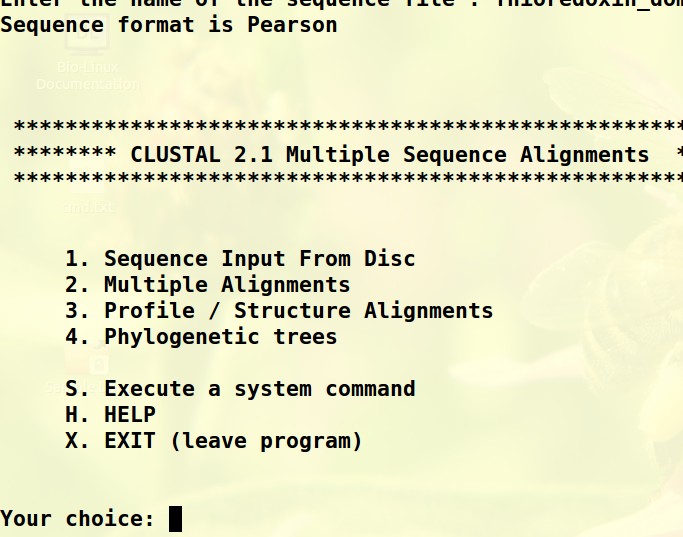
把WRKY_domain.fa和WRKY_domain2.fa的序列copy到同一个fasta文件中(用Editplus软件,复制两个文件的序列粘贴到一个新建文件中),clustalw不能读出蛋白信息。如下图:第一张图是Sequence Input From Disc输入的WRKY_domain.fa文件,回车后可以出现序列对应的蛋白名称和AA长度;第二张图是Sequence Input From Disc输入的合并后的domain.fa文件 sequence is pearson下面显示的是空白,没有蛋白相关信息。
请问如何操作处理?怎样将把WRKY_domain.fa和WRKY_domain2.fa两个文件的序列正确合并?
请先 登录 后评论
3 个回答
- 3 关注
- 0 收藏,3669 浏览
- Maggie 提出于 2019-04-11 16:16
